Si preguntamos en la calle ¿cuál es la principal diferencia entre un varón y una mujer?, seguramente las respuestas más frecuentes serán los senos, los órganos reproductores, la barba, la obsesión por los zapatos o por los videojuegos, entre otros. Pero muy pocos —tal vez algunos biólogos que cayeron en la encuesta— dirán “¡los cromosomas sexuales!”. Y tendrían razón.
La diferencia más sustancial, a partir de la cual se originan todas las demás, son los cromosomas sexuales. En los mamíferos, las hembras tienen dos cromosomas X (XX) y los machos un cromosoma X y un cromosoma Y (XY). A pesar de ser chiquito, el cromosoma Y porta un gen esencial para lograr la diferenciación masculina. De no ser por él, prácticamente todos seríamos hembras, así tuviéramos solo un cromosoma X (X0) como en el
Síndrome de Turner.
Entonces, serán los machos quienes finalmente determinen el sexo de los hijos porque sus espermatozoides portarán o bien el cromosoma X o bien el cromosoma Y; mientras que los óvulos sólo portarán el cromosoma X. Esto trae como resultado que las hembras posean un cromosoma X proveniente del padre (Xp) y un cromosoma X proveniente de la madre (Xm).
Creo que todos aprendimos esto en nuestra clase de biología del colegio. Sin embargo, lo que no todos saben es que en las hembras, uno de sus cromosomas X se inactiva.
Seguro se estarán preguntando ¿por qué lo hace?
El cromosoma X tiene alrededor de 1000 genes. La gran mayoría son esenciales para el desarrollo y viabilidad de las células, pero no para la diferenciación sexual y la reproducción. Los machos, a pesar de tener un solo cromosoma X, no presentan problemas en su desarrollo. Por lo tanto, si las hembras tuvieran los dos cromosomas X activos, las células recibirían una doble dosis de cada uno de los productos de estos 1000 genes, provocando un desequilibrio metabólico que podría alterar su normal desarrollo.
En 1961,
Mary Lyon descubrió que uno de los cromosomas X de los mamíferos se inactiva convirtiéndose en una masa oscura llamada
corpúsculo de Barr. Y ¿cuál de los dos se inactiva? Hice esta pregunta a unos amigos biólogos por aquí y la mayoría me respondió el X paterno. Creo que es la primera respuesta que se nos viene a la cabeza. Pero no es así. En en algunas células el Xp es el inactivo mientras que en otras el Xm lo es, y al parecer esta selección es al azar.
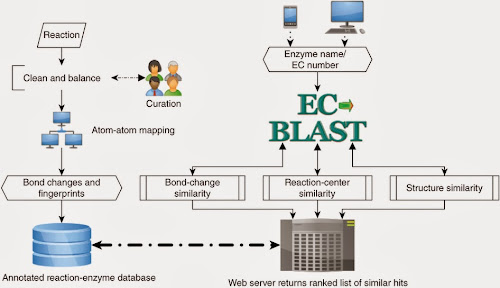
Recientemente, un grupo de investigadores del Howard Hughes Medical Institute y de la Jhons Hopkins University (Estados Unidos) han desarrollado una forma de ver la inactivación del cromosoma X directamente en el cuerpo, célula por célula. El estudio fue publicado el 8 de enero en Neuron.
La técnica consiste en iluminar a las células de dos diferentes colores dependiendo del cromosoma X que inactiven. Para ello, insertaron el gen de la proteína fluorescente verde (GFP) en los cromosomas X de un ratón (digamos, el padre) y el gen de una proteína fluorescente roja (tdTomato) en los cromosomas X de otro ratón (la madre). Luego cruzaron estos dos ratones y seleccionaron a las crías hembras, las cuales deberían tener los cromosomas X del padre y de la madre con sus respectivas proteínas fluorescentes. Si el Xp se inactiva, no se expresa la GFP —pero sí la tdTomato— y las células brillarían de color rojo. Si el Xm se inactiva, pasaría lo contrario y las células brillarían de color verde.
Cuando se analizaron los diferentes tejidos del ratón hembra bajo el microscopio de fluorescencia observaron este maravilloso mosaico:
Cabeza de un embrión de ratón.
Corazón (músculo cardiaco).
Corte transversal de la lengua.
Variación en el mosaicismo entre la retina izquierda y derecha de un ratón adulto.
Mosaicismo en un cerebro adulto que muestra la asimetría en la inactivación del cromosoma X entre el hemisferio izquierdo y derecho.
Vellosidades intestinales.
Espectacular, ¿cierto? Se ve claramente que tanto el cromosoma X paterno como el materno pueden ser inactivados, y como mencionamos anteriormente, este proceso puede ser al azar. Sin embargo, las imágenes también nos muestran que existen tejidos en los cuales predomina un sólo tipo de células (rojas o verdes). Aún se desconoce por qué ocurre esto y qué efecto podría tener sobre el organismo, especialmente, a nivel del sistema nervioso central, ya que los alelos (variantes del mismo gen) que posee el cromosoma X paterno pueden ser distintos al materno.
Referencia:
Hao Wu, Junjie Luo, Huimin Yu, Amir Rattner, Alisa Mo, Yanshu Wang, Philip M. Smallwood, Bracha Erlanger, Sarah J. Wheelan, Jeremy Nathans. Cellular Resolution Maps of X Chromosome Inactivation: Implications for Neural Development, Function, and Disease.
Neuron. 81(1): 103-119. doi:
10.1016/j.neuron.2013.10.051
Además, te recomiendo que leas
este gran artículo de Carl Zimmer en
The New York Times referente a este tema.













 Capsicum annuum. Fuente:
Capsicum annuum. Fuente: