Hablar del origen de la vida es, sin dudas, un tema que me apasiona. Poder explicar algún día como se originó todas las formas de vidas que hoy conocemos, saber si tuvimos un sólo ancestro común o muchos que se fueron extinguiendo a través del tiempo, si los eucariotas descienden de la unión de los procariotas y las arqueas, cual fue la primera molécula con capacidad replicativa e informativa, explicar la paradoja del huevo y la gallina, etc. sin dudas son temas fascinantes.
Personalmente, creo que todos los seres vivos venimos de un mundo primitivo en base al ARN. Las evidencias son muchas: las ribozimas, cadenas de ARN con la capacidad de autoreplicarse; los ribosomas, la maquinaria de transformación de la información genética en proteínas, cuanta con secuencias de ARN con actividad enzimática; la replicación del ADN necesita de un cebador hecho a base de ARN; la presencia de moléculas de ARN capaces de regular la expresión de los genes (microARNs, ARN de interferencia, ARN largos no codificantes, los riboswitches, etc.) y los ARN de transferencia que transportan los aminoácidos para su polimerización y formación de proteínas. Todo el funcionamiento de una célula se basa en el ARN!.
Pero, hasta antes de leer este artículo publicado ayer en Nature Communications, mi concepto del origen de la vida a partir del ARN era completamente diferente. Yo creía que la formación de las secuencias de ARN capaces de autoreplicarse se dio en un medio líquido, a una temperatura relativamente elevada – algo así como un “caldo primitivo” – ya que las altas temperaturas favorecen las reacciones químicas – antes de la presencia de las enzimas –; aún así, las enzimas requieren de temperaturas por arriba del punto de congelación del agua para poder funcionar. El agua debe estar en estado líquido para que las reacciones químicas puedan llevarse a cabo. Sin embargo, el ARN es una molécula sumamente inestable a temperaturas altas… esta es una de las razones de porque la información genética se almacena ahora en forma de ADN (es más estable que el ARN).
Este artículo nos da una prueba muy convincente de que el mundo del ARN pudo haberse desarrollado en el hielo!. Ahora le explico como…
En primer lugar, todos sabemos que el agua se congela a 0°, pero cuando tiene una determinada concentración de otros compuestos disueltos en él, esta temperatura se verá reducida. Entonces, ¿que pasa si sometemos a esta solución a una temperatura menor a su temperatura de congelamiento?… Se formará un sistema bifásico!. El agua se separará de los iones y se empezará a congelar; los iones se concentrarán en determinadas zonas manteniéndose líquidas a bajas temperaturas. Digamos que a parte de los iones tenemos ribonucleótidos (principal componente del ARN); al concentrarse en estas regiones intersticiales ¿no aumentará la probabilidad de que reaccionen y formen estructuras más complejas como el ARN? El ARN a bajas temperaturas es una molécula sumamente estable!
Attwater et al. aprovechó de esta característica del agua para realizar sus experimentos. Lo que hizo fue tomar una ribozima modelo, la Round-18 (R18), y una ARN Polimerasa (T7 RNA pol) que sirvió de control, para evaluar la capacidad de replicar pequeñas moléculas de ARN en el hielo. Adivinen que… La R18 lo podía hacer a -7°C! (Ver la figura y explicación)
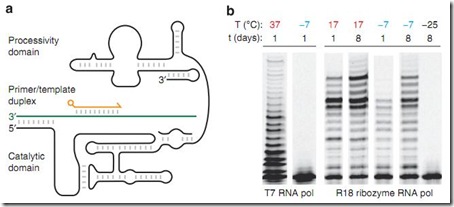
La figura a. muestra la estructura de la ribozima R18. Esta ribozima no es más que una secuencia de ARN de unos 195 nucleótidos con la capacidad de replicar secuencias de ARN, sólo mediante ataques nucleofílicos (reordenamiento de enlaces). Pero, lo mejor de todo está en la figura b. Como pueden ver T7 RNA pol actúa perfectamente a 37°C pero a -7°C pierde completamente su actividad. En cambio, la R18 es activa a 17°C y a -7° (tampoco se maleen y quieran que sea activa a -25°C). Y no sólo eso, la ribozima estuvo activa hasta por 8 días!. Esto indica que las ribozimas pueden funcionar en el hielo, donde el ARN es más estable y la baja temperatura no afecta su actividad, tal como antes se creía.
Además demostraron que si las soluciones de reacción están altamente diluidas, o sea, la concentración de ribonucleótidos, R18 y el MgCl2 (recordemos que el Mg es un cofactor que es necesario para la replicación y transcripción del material genético) son hasta 200 veces menores, la replicación del ARN no se detiene.
Ahora quiero que ven esta imagen:

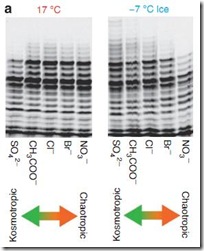 Si no entendían bien lo que era el sistema bifásico y el espacio intersticial ahora lo podrán distinguir claramente. Ese surco que hay entre los bloques (hielo) es el espacio intersticial, donde se encuentran los iones concentrados en solución líquida. Ahora recuerden que el Mg es un cofactor importante en la replicación y transcripción del material genético, ya que gracias a él las polimerasas y las ribozimas pueden realizar la ligación del nuevo nucleótido a la cadena que se va formando (actividad ligasa). Y si recuerdan un poco más, ¿en que forma agregan el Mg a su PCR?… ¿En forma de MgCl2 cierto?… Y si cambiamos al Cl (contraión) por otro, ¿tendrá algún efecto?…
Si no entendían bien lo que era el sistema bifásico y el espacio intersticial ahora lo podrán distinguir claramente. Ese surco que hay entre los bloques (hielo) es el espacio intersticial, donde se encuentran los iones concentrados en solución líquida. Ahora recuerden que el Mg es un cofactor importante en la replicación y transcripción del material genético, ya que gracias a él las polimerasas y las ribozimas pueden realizar la ligación del nuevo nucleótido a la cadena que se va formando (actividad ligasa). Y si recuerdan un poco más, ¿en que forma agregan el Mg a su PCR?… ¿En forma de MgCl2 cierto?… Y si cambiamos al Cl (contraión) por otro, ¿tendrá algún efecto?…
Los investigadores reemplazaron el Cl por: (SO4)-, (CH3COO)-, Br- y NO3- (están en orden, del más kosmotrópico al más caotrópico: Ver el Efecto Hofmeister para entender el significado de esto); con el fin de determinar su efecto sobre la replicación. A 17°C el más kosmotrópico (aumenta la tensión superficial del solvente) tenía un efecto negativo en la replicación; sin embargo, a -7°C, claramente se veía que a mayor kosmotropicidad, la replicación era más beneficiada. Este es un gran descubrimiento, ya que en un ambiente primitivo, la concentración de iones sería muy diversa y podemos apreciar que en el hielo, no perjudican la replicación del ARN!.
Ahora pueden entender por qué los espacios intersticiales son diferentes… con MgSO4 es “más bonito” porque la tensión superficial es mayor y su punto eutéctico aumentará (-33°C en el MgCl2 a -4°C en el MgSO4). Este fenómeno es muy importante para que se de otro de los grandes logros de la vida… la compartamentalización.
Si bien el Mg es importante para el funcionamiento de la ribozima, su presencia en altas concentraciones reduce el tiempo de vida media de la enzima a 52 horas, cuando ésta trabaja a 17°C. Los investigadores encontraron que a las mismas concentraciones altas de Mg, su tiempo de vida media se prolongaba a 17 días, cuando se trabajaba a -7°C. Entonces el hielo puede mantener a la ribozima estable por mucho tiempo, lo cual no sería posible a temperatura ambiente.
Pero, ¿como se comporta en cuanto a las mutaciones?. Ya mencionamos en un artículo anterior el problema de las ribozimas… su alta tasa de mutación. Al tener una alta tasa de mutación, las nuevas ribozimas formadas pierden su actividad autoreplicativa, así que al final las ribozimas desaparecerían y la vida no hubiera podido avanzar. Aquel artículo menciona una bonita estrategia hecha por estas ribozimas primitivas para evitar su desaparición debido a las mutaciones perjudiciales. Como las ribozimas dependen mucho de la estructura secundaria del ARN y las horquillas que se forman para poder replicarlo fidedignamente, de repente una baja temperatura puede afectar esta estructura y generar más mutantes de lo normal. Los investigadores observaron que a 17°C la fidelidad de replicación (replicación sin errores) de la ribozima fue del 97.1%, muy cercano al 95.9% obtenido en el hielo a -7°C. Además, en ambos casos, se obtenían la misma proporción de substituciones la única diferencia fue que a -7°C el porcentaje de deleciones fue mayor.
En realidad son datos y resultados muy alentadores pero el estudio tiene algunas desventajas. En primer lugar, la ribozima usada, R18, si bien es la mejor opción de una ribozima análoga a una primitiva, no es la más adecuada. Esta R18 no está adaptada a funcionar a -7°C, ella ha evolucionado para funcionar a 22°C. De repente con una ribozima primitiva los resultados serían diferentes (para mejor o para peor). Además, como máximo se obtuvieron ARNs replicados de 33 nucleótidos de longitud. Por lo menos se requieren 195 nucleótidos para generar un ARN con capacidad autoreplicativa. Además, nadie nos garantiza que los sustratos usados en el presente trabajo y las concentraciones usadas también estuvieron presentes en un hielo primitivo.
Sin embargo, estos resultados son más que alentadores y nos abren toda una nueva perspectiva de como se pudo haber originado la vida. De todas maneras se requieren más estudios en este campo. La importancia de entender como se originó la vida es inmensa ya que también impulsaría el campo de la astrobiología y entender los principios de la evolución (si en realidad se dio, porque también tengo mi teoría que en otro momento les comentaré).
Perdonen por la extensión de este post, pero cuando escribo de estos temas me emociono. El artículo lo pueden descargar libremente para que puedan profundizar más en el tema.
Referencia:
 Attwater, J., Wochner, A., Pinheiro, V., Coulson, A., & Holliger, P. (2010). Ice as a protocellular medium for RNA replication Nature Communications, 1 (6), 1-8 DOI: 10.1038/ncomms1076
Attwater, J., Wochner, A., Pinheiro, V., Coulson, A., & Holliger, P. (2010). Ice as a protocellular medium for RNA replication Nature Communications, 1 (6), 1-8 DOI: 10.1038/ncomms1076