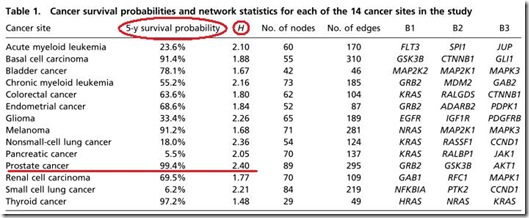
Nature publica uno de los dos controversiales artículos sobre el virus de la gripe aviar desarrollado en el laboratorio con el potencial de desarrollar una pandemia.

En agosto del 2011, dos grupos de investigadores, uno liderado por el Dr. Ron Fouchier del Centro Médico Erasmus (Holanda) y el otro por Dr. Yoshihiro Kawaoka de la Universidad de Wisconsin-Madison (EEUU), mandaron sus respectivos estudios a las dos principales revistas científicas —Science y Nature, respectivamente— en los cuales reportaban el desarrollo de cepas mutantes del virus de la gripe aviar (H5N1) con la capacidad de transmitirse entre mamíferos a través del aire.
En diciembre, la Junta Nacional de Asesoría para la Bioseguridad de la Ciencia de los Estados Unidos (NSABB) recomendó a las dos revistas censurar estos artículos, o al menos, la metodología esencial y los datos, por considerarlos una amenaza a la seguridad mundial, alegando que la información podría ser usada por grupos terroristas para el desarrollo de armas biológicas.
Después de varias semanas de acalorado debate entre diversos expertos en el tema, la NSABB cambió su postura y también dio luz verde a la publicación de los dos artículos, sumándose así a la misma recomendación que dio la Organización Mundial de la Salud (OMS) semanas antes. El pasado miércoles 2 de mayo, Nature tomó la decisión de publicar en acceso abierto el artículo que le correspondía. Science lo deberá estar haciendo en los próximos días.
 Virus H5N1
Virus H5N1
Existen muchos virus responsables de la gripe, uno de ellos son los del tipo A. Estos virus son identificados en función qué versión de proteína Hemaglutinina (HA) y Neuraminidasa (NA) tengan en su superficie. La primera es responsable de que el virus reconozca específicamente a la célula que va a infectar, mientras que la segunda se hace cargo de la diseminación del virus hacia otras células. Los genes que codifican estas proteínas se encuentran en constante cambio, dificultando así el desarrollo de fármacos eficaces para su tratamiento.
En 1997, el mundo se puso alerta cuando un virus de gripe infectó a 18 asiáticos de los cuales seis murieron. No se volvió a saber de él hasta que en el 2003 reapareció en Hong Kong. Las investigaciones apuntaban a que se trataba de un virus de origen aviar, específicamente del tipo H5N1, el cual infectaba tanto a aves acuáticas como a las de corral. Desde entonces ha infectado a 603 personas de las cuales 356 han muerto (casi un 60% de mortalidad). Por razones aún en investigación, este virus es extremadamente agresivo en humanos.
En aves, el H5N1 reconoce a las proteínas receptoras a través del ácido siálico unido a la galactosa mediante un enlace α2,3 (Siaα2,3Gal), mientras que en humanos lo hace a través de las mismas moléculas pero con un enlace α2,6. Esta diferencia explica por qué el H5N1 infecta muy raras veces a los humanos.
Generación del mutante
Con el fin de identificar nuevos virus H5N1 que se transmitan eficientemente en humanos, Kawaoka y su equipo introdujeron mutaciones al azar en la HA de una cepa aislada en el 2004 de un paciente vietnamita. Tomaron el gen responsable de la síntesis de esta proteína y lo sometieron a una PCR propensa a errores. En términos sencillos, esta técnica permite sacar miles de copias de un gen pero con errores en su secuencia, generando así miles de versiones mutantes.
Luego, los investigadores insertaron cada uno de los HA mutantes en cultivos de células humanas. Como el virus de la gripe consta de ocho genes, los siete genes restantes necesarios para generar un nuevo virus fueron obtenidos de aislamientos de H1N1 de la pandemia del 2009 (la famosa gripe porcina). De esta manera se obtuvo una gran cantidad de virus híbridos H5N1/H1N1, cada uno con una versión de HA diferente.
En total fueron 2.1 millones de virus mutantes de los cuales 370 lograron reconocer la versión Siaα2,6Gal de la proteína receptora humana. Estos virus pasaron por una segunda ronda de selección de los cuales nueve mostraron un alto grado de aglutinación en los receptores. Al analizar sus secuencias de aminoácidos hallaron varias mutaciones que podrían estar relacionadas con el aumento de la eficiencia de dichos receptores, incluso algunas de ellas ya habían sido descritas anteriormente. En una tercera y última ronda de selección, se probó la especificidad de los nueve finalistas por Siaα2,6Gal y no por otro receptor similar. Sólo uno de los mutantes logró pasar la prueba.
Mejora del mutante
El virus seleccionado mostraba cuatro mutaciones en la HA: E119G, V152I, N224K y Q226L. Cada letra representa un aminoácido y el número la posición. E119G quiere decir que el Glutamato (E) de la posición 119 fue cambiado por la Glicina (G).
 Para determinar cuál(es) de estas cuatro mutaciones era responsable del reconocimiento del receptor Siaα2,6Gal, Kawaoka y su equipo empezaron a cambiar los aminoácidos de las posiciones 119, 152, 224 y 226, probando diferentes combinaciones y demostrando que las mutaciones N224K y Q226L juntas eran claves en el cambio de reconocimiento del receptor aviar por el humano, especialmente por las células de la tráquea.
Para determinar cuál(es) de estas cuatro mutaciones era responsable del reconocimiento del receptor Siaα2,6Gal, Kawaoka y su equipo empezaron a cambiar los aminoácidos de las posiciones 119, 152, 224 y 226, probando diferentes combinaciones y demostrando que las mutaciones N224K y Q226L juntas eran claves en el cambio de reconocimiento del receptor aviar por el humano, especialmente por las células de la tráquea.
Así surgía una primera pregunta: ¿éste nuevo virus mutante podrá replicarse normalmente en los humanos? Lo ideal hubiera sido hacer pruebas en humanos, pero razones obvias no es posible. Entonces, se usaron a los hurones, unos pequeños mamíferos que son usados como buenos modelos biológicos para estudiar la transmisión de la gripe.
Los investigadores infectaron a seis hurones con este virus mutante. Una semana después de iniciado el experimento, los virus seguían replicándose eficientemente. Tomaron una muestra de ellos y les analizaron la secuencia de aminoácidos de HA, encontrando dos nuevas mutaciones en la posición 158 (N158D y N158K). Los virus que tenían las tres mutaciones se hicieron más abundantes hacia el sexto día en comparación con los virus que solo tenían las dos mutaciones originales. Esto indicaba que los cambios en la posición 158 mejoraban la capacidad de replicación del virus, siendo N158D la más eficiente.
Este cambio en la posición 158 provocaba que el HA del virus perdiera su capacidad de glicosilarse (la unión de una glucosa a un determinado aminoácido). Un estudio publicado en el 2009 demostró que la pérdida de glicosilación en las posiciones 158 a 160 mejoraba la transmisión del virus por contacto directo entre cobayos (cuyes). La pregunta ahora era, ¿ocurrirá lo mismo en hurones?
Los investigadores volvieron a infectar seis hurones, esta vez con el triple mutante, y los pusieron en jaulas contiguas con otros seis hurones no infectados formando pares. Las predicciones se hicieron realidad, dos de los hurones fueron contagiados a través del aire.
Kawaoka y su equipo volvieron a analizar la secuencia de aminoácidos de HA, esta vez de los virus aislados de los hurones contagiados, encontrando una nueva mutación (T318I) en uno de los pares. Para tener una mejor idea de la distribución de las cuatro mutaciones obtenidas en este experimento, miren el siguiente gráfico que representa la estructura de la Hemaglutinina:
Para corroborar sus resultados, los investigadores repitieron el experimento anterior, pero esta vez usando al cuádruple mutante. Cuatro de los seis hurones sanos mostraron la presencia del virus al tercer y séptimo día de contacto, demostrando así que el nuevo virus se diseminaba con facilidad a través del aire.
Un análisis posterior determinó que la última mutación adquirida por la HA le confería al virus una mayor estabilidad en ambientes ácidos, los cuales son típicos de las mucosas del tracto respiratorio. Además, este nuevo virus mostró una mayor tolerancia a las altas temperaturas.
¿Pandemia?
Para este punto, Kawaoka y su equipo desarrollaron un virus de gripe aviar con cuatro mutaciones específicas en la Hemaglutinina —la proteína responsable del reconocimiento de las células— que les conferían la capacidad de reconocer las células de la tráquea humana y diseminarse fácilmente a través del aire, al menos en hurones.
Fue esto lo que asustó a los científicos y especialistas de la OMS y la NSABB y provocó la censura de este artículo. Conociendo las mutaciones que volvían a un virus mortal pero incapaz de infectar en humanos —salvo raras ocasiones— en uno más eficiente y de fácil transmisión, cualquier persona, incluso terroristas, podían desarrollarlo en un laboratorio que disponga de la tecnología.
Sin embargo, los investigadores creen que los miedos eran infundados y que era más peligroso no publicar los artículos que hacerlo, porque, el estudio demostraba los pocos cambios necesarios para desarrollar una cepa mucho más infecciosa, los cuales se pueden dar fácilmente en la naturaleza. Ahora, gracias a que conocemos esto, podemos estar mejor preparados ante cualquier variante de gripe aviar que se desarrolle más adelante.
Kawaoka y su equipo demostraron que su virus desarrollado en el laboratorio no era peor que el la H1N1 del 2009. Al comparar la sintomatología de estos dos virus, no se observó diferencias significativas entre ellos, es más, el H5N1 mutante era menos agresivo con la mucosa nasal. También demostraron que el suero obtenido de personas vacunadas contra la H5N1típica mostraban reactividad contra este nuevo virus H5N1, lo que indicaría que también estarían protegidos contra él. Asimismo, el oseltamivir —un fármaco usado contra la H1N1— también mostró ser eficiente contra el H5N1 mutante. Y por su fuera poco, ninguno de los hurones que participaron de los experimentos murieron a causa del virus.
Todos estos resultados apuntan a lo mismo: en caso que alguien desarrolle un arma biológica en base a este mismo virus o el virus logre escapar de su confinamiento en el laboratorio de Dr. Kawaoka, la población humana no se verá amenazada.
Finalmente, el virus seguirá evolucionando fuera de los laboratorios, ya tal vez algún día surja una variante que realmente ponga bajo amenaza a la humanidad, pero gracias a estos trabajos podemos estar más tranquilos porque no nos agarrará desprevenidos.
Ahora solo queda esperar la publicación del trabajo de Fouchier en Science, el cual es más controvertido que este porque el virus que ellos desarrollaron era mucho más patogénico, varios hurones murieron durante los experimentos. Además, la votación que hicieron los especialistas del NSABB para dar luz verde a su publicación no fue unánime, como si lo fue en el trabajo de Kawaoka.
Referencia:
 Imai, M., Watanabe, T., Hatta, M., Das, S., Ozawa, M., Shinya, K., Zhong, G., Hanson, A., Katsura, H., Watanabe, S., Li, C., Kawakami, E., Yamada, S., Kiso, M., Suzuki, Y., Maher, E., Neumann, G., & Kawaoka, Y. (2012). Experimental adaptation of an influenza H5 HA confers respiratory droplet transmission to a reassortant H5 HA/H1N1 virus in ferrets Nature DOI: 10.1038/nature10831
Imai, M., Watanabe, T., Hatta, M., Das, S., Ozawa, M., Shinya, K., Zhong, G., Hanson, A., Katsura, H., Watanabe, S., Li, C., Kawakami, E., Yamada, S., Kiso, M., Suzuki, Y., Maher, E., Neumann, G., & Kawaoka, Y. (2012). Experimental adaptation of an influenza H5 HA confers respiratory droplet transmission to a reassortant H5 HA/H1N1 virus in ferrets Nature DOI: 10.1038/nature10831
Imágenes | Nature 485,13–14 doi:10.1038/485013a