Le voy a comentar una interesante hipótesis que salió el jueves en Nature. Antes de empezar deben tener muy en claro que los procariotas son aquellos organismos simples, sin organelos especializados como mitocondrias, cloroplastos, lisosomas, retículo endoplasmático, etc. El grupo de las procariotas están formado por las bacterias y las arqueas (microorganismos que suelen vivir en ambientes extremos). Los eucariotas, son organismos más complejos y altamente estructurados, con la capacidad de agregarse para formar tejidos y órganos con funciones complejas. Al grupo de las eucariotas pertenecen desde las levaduras y hongos hasta las plantas y animales. Los eucariotas más sencillos, después de las levaduras, son los protozoos (Ej. amebas y paramecios).
Si pensamos por un momento cómo se originó la vida, los primeros en formarse de seguro fueron los procariotas, por ser más sencillos. ¿Donde? En los océanos primitivos. ¿Cuando? Posiblemente, hace unos 3500 millones de años. Los eucariotas aparecieron posiblemente hace 2000 millones de años. Entonces, ¿por qué los procariotas no se han vuelto más complejos que los eucariotas, si tienen más de 1500 millones de años de ventaja en tiempo evolutivo? Si bien muchos procariotas tienen, virtualmente, varias características de las eucariotas como: cromosomas lineares, membranas internas, sistemas de señalización (quorum sensing), poliploidía (repetición del material genético), tamaños gigantes, hasta endosimbiontes. Entonces, ¿por qué no se volvieron tan complejos como los eucariotas?
 A todos salta a la vista que la diferencia más importante entre las eucariotas y las procariotas fue la adquisición de mitocondrias. Hay muchas teorías que explican el origen de las eucariotas. La más aceptada es la endosimbiosis, donde una bacteria primitiva con la capacidad de producir energía fue engullida por otra más grande, y al final, la más pequeña pudo vivir dentro de la grande (se pudo dividir, producir energía, etc.) y formar la mitocondria. Así que el origen de las mitocondrias y de las células eucariotas pudo haber sido el mismo evento.
A todos salta a la vista que la diferencia más importante entre las eucariotas y las procariotas fue la adquisición de mitocondrias. Hay muchas teorías que explican el origen de las eucariotas. La más aceptada es la endosimbiosis, donde una bacteria primitiva con la capacidad de producir energía fue engullida por otra más grande, y al final, la más pequeña pudo vivir dentro de la grande (se pudo dividir, producir energía, etc.) y formar la mitocondria. Así que el origen de las mitocondrias y de las células eucariotas pudo haber sido el mismo evento.
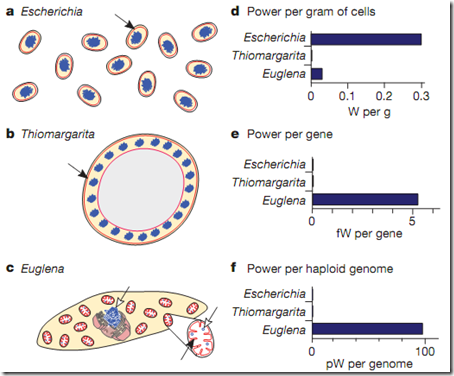
Entonces, ¿fue la adquisición de las mitocondrias un evento clave en la complejidad de las eucariotas? Y si fuera así… ¿Que ventaja le daba tener mitocondrias con respecto a las procariotas? No fue la respiración aeróbica, ya que las mitocondrias no incrementaron la tasa respiratoria de las eucariotas. Gramo por gramo, las procariotas respiran más rápido que las eucariotas. Entonces, ¿fue la compartamelización de la respiración dentro de la célula? Tampoco, ya que las procariotas pueden invaginar su membrana celular y también formar compartimentos. Entonces, las procariotas, con mayor tasa de respiración y compartamelización, parecieran tener una ventaja energética con respecto a las eucariotas, aún así, ¿por qué no se volvieron más complejas? Lane & Martin hipotetizan que la clave de todo fue la energía por gen expresado.
Un organismo es más complejo cuando su genoma es más complejo. Un genoma más complejo no quiere decir un genoma grande. Por ejemplo, las amebas tienen genomas más grandes que el genoma humano, pero no son tan complejos como nosotros.
Ahora vamos con un poco de matemáticas… (Usaremos como un eucariota modelo a un protozoo).
En procariotas, el 75% de la energía del organismo es invertida en la síntesis de proteínas y solo 2% en la replicación del ADN. Una bacteria promedio tiene unas 4400 proteínas diferentes, así que, invertirá 0.017% de su energía por cada proteína. Si queremos que nuestra bacteria sea más compleja, le aumentamos unas 10 veces su número de proteínas, así que ahora tendrá 44000. Pero, como la cantidad de energía de la bacteria sigue siendo la misma, debe redistribuir el 75% de energía que usa, en las nuevas proteínas (35% para las pre-existentes y 35% para las nuevas). Entonces, la bacteria producirá la mitad de las proteínas pre-existentes y sólo invertirá el 0.0009% de energía por cada una de las 40000 nuevas proteínas (la 1/20 parte). Si queremos más proteínas, la energía invertida por la bacteria debe ser mayor. Ya vemos que hay un pequeño problema energético.
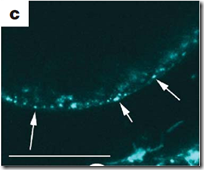 Las eucariotas, por ser más grandes, poseen cientos de proteínas estructurales. Pero, ser más grandes no significa ser más complejas. Por ejemplo. La bacteria Thiomargarita namibiensis – la más grande de todas – mide casi 1 milímetro de diámetro (de 5 a 20 veces más grande que una eucariota!), sin embargo, sigue careciendo de complejidad, pero, es altamente poliploide (su genoma esta altamente repetido). Como las bacterias no tienen núcleo, su genoma se organiza en unas estructuras denominadas nucleoides. Los nucleoides (puntitos azules en la imagen) están asociados a la membrana celular de la bacteria que es ahí donde se produce la energía. Aquí una clave importante en esta hipótesis. La complejidad del genoma tiene que ver directamente con la producción de energía. En eucariotas, la producción de energía se da principalmente en las mitocondrias, las cuales tiene su propio genoma, el ADN mitocondrial. Las cuales también están asociadas a sus membranas mitocondriales que es donde se produce la energía.
Las eucariotas, por ser más grandes, poseen cientos de proteínas estructurales. Pero, ser más grandes no significa ser más complejas. Por ejemplo. La bacteria Thiomargarita namibiensis – la más grande de todas – mide casi 1 milímetro de diámetro (de 5 a 20 veces más grande que una eucariota!), sin embargo, sigue careciendo de complejidad, pero, es altamente poliploide (su genoma esta altamente repetido). Como las bacterias no tienen núcleo, su genoma se organiza en unas estructuras denominadas nucleoides. Los nucleoides (puntitos azules en la imagen) están asociados a la membrana celular de la bacteria que es ahí donde se produce la energía. Aquí una clave importante en esta hipótesis. La complejidad del genoma tiene que ver directamente con la producción de energía. En eucariotas, la producción de energía se da principalmente en las mitocondrias, las cuales tiene su propio genoma, el ADN mitocondrial. Las cuales también están asociadas a sus membranas mitocondriales que es donde se produce la energía.
Más matemáticas…
La tasa metabólica promedio de las bacterias es 0.19±0.5 Watts por gramo de bacterias (W/g), pero cada bacteria pesa sólo 2.6x10-12g, entonces, la tasa metabólica por cada bacteria será de 0.49pW (…)
1W=1000mW; 1mW=1000uW; 1uW=1000nW; 1nW=1000pW y 1pW=1000fW; entonces…
1W=1’000,000’000,000pW ó 1pW=0.000000000001W (sumamente pequeña).
(…)
y la tasa metabólica promedio de las eucariotas (un protozoo) es 0.06±0.1W/g, pero su masa es 40100x10-12g (casi 20 mil veces más que las bacterias), entonces, la tasa metabólica por cada célula eucariota será 2286pW. Las eucariotas tienen 5000 veces más poder metabólico que las procariotas, o sea, más energía para mantener un genoma más complejo.
Ahora vamos a hacer los cálculos en función del tamaño del genoma. Más matemáticas… (la tasa metabólica lo expresaremos, simplemente, como la energía del organismo)
Una bacteria promedio tiene un genoma de un tamaño promedio de 6Mb (6000000 pares de base). Entonces, si dividimos su tasa metabólica (0.49pW) por el tamaño de su genoma, obtendremos una tasa metabólica por genoma de 0.08pw/Mb. Un protozoo promedio tiene un genoma de 3000Mb, así que su tasa metabólica por genoma será 0.76pW/Mb. Claramente vemos que los eucariotas tienen más energía por Mb de ADN. Así la bacteria tenga un genoma más pequeño y el eucariota uno más grande, la energía por MB de ADN siempre será mayor en el eucariota.
Pero, no nos olvidemos del ADN mitocondrial…
En promedio, el ADN mitocondrial (ADNmit) mide 30Kb (30000 pares de base) y en promedio un protozoo tiene 200 mil copias por célula. Así que el contenido de ADNmit será de 6000Mb por célula! (Más del doble del tamaño del ADN nuclear). En total, un protozoo tendrá 9000Mb por célula. Así que la energía por genoma será ahora 0.25pW/Mb. Aún así superior a la tasa de las bacterias (0.08pW/Mb).
Que pasaría para aumentar la complejidad genómica de la bacteria aumentamos el tamaño de su genoma a 3000Mb – como el de un eucariota; pero, manteniendo constante su tasa metabólica?
Simplemente, la energía por genoma caería a 0.16fW/Mb (0.00016pW/Mb), unas 4600 veces menor al de un protozoo!.
Ahora, para que no se cansen con tantos cálculos – que no son más que simples divisiones – calcularemos la energía por gen, que es lo que realmente interesa.
Una bacteria tiene en promedio 5000 genes y su tasa metabólica por célula era 0.49pW, así que la energía será de 0.0001pW/gen. Los protozoos tiene en promedio 20000 genes y su tasa metabólica era 2268pW, así que su energía será de 0.115pW/gen (1200 veces superior al de las bacterias!). Si la bacteria tendría el mismo número que los protozoos, su energía caería a 0.00003pW/gen, unas 4600 veces menos que en las eucariotas.
Ya vemos, que la clave de todo es la energía por gen! Pero, aún así no podemos descartar el tamaño superior de las células eucariotas. ¿Por qué las favorecen más y no lo hacen con las procariotas?. Las células producen energía a través de sus membranas plasmáticas, para producir más energía se debe aumentar el área superficial de la membrana, pero para aumentar el área de la membrana, también debe aumentar el volumen, y cuanto mayor es el volumen, mayor será el requerimiento de proteínas, ¿me dejo entender?. Si una bacteria la llevamos al mismo volumen, forma y tamaño de genoma que un protozoo, su energía por genoma caerá estrepitosamente a 0.003fW/Mb, unas 250 mil veces menor al de los protozoos! Y si lo llevamos a energía por gen, esta caerá a 0.0005fW/gen, 230 mil veces menor al de los protozoos!.
En resumen, un gen de un eucariota tendrá disponible 200 mil veces más energía que un gen procariota, así que podrá soportar un genoma 200 mil veces más grande. Esta gran energía requerida por un genoma eucariota es provista por las mitocondrias. En otras palabras, las mitocondrias permitieron superar la barrera energética a los eucariotas primitivos, les permitió explorar, evolucionar y expresar 200 mil genes más, sin una penalidad energética a la que están sometidas las procariotas. Pero, otra clave fue que los genes innecesarios no fueron expresados y fueron eliminados, es por esta razón que las eucariotas cargan sólo 12 genes por cada millón de pares de base (Mb), a diferencia de las bacterias que cargan 1000 genes por cada Mb.
Si la densidad genética de las bacterias fuera como la de los eucariotas (12genes/Mb), un genoma bacteriano promedio de 6Mb sólo cargaría a lo mucho 100 genes. Además, con solo una energía de 0.08pW/Mb, no podría mantener el ADN regulador (siARN, miARN) ni el ADN no codificante. Por esta razón, las bacterias tienen una alta densidad genética, además han eliminado las regiones inter e intragenéticas y han organizado sus genes en operones, para que una sola proteína pueda regular la expresión de muchos genes. En cambio, con 0.76pW/Mb y 12genes/Mb de las eucariotas, en principio podrían sostener 350 mil genes, permitiéndoles evolucionar, expresar y explorar nuevas familias genéticas, que expresarán nuevas proteínas, así como también, ADN no codificantes y reguladores.
Finalmente, las mitocondrias, al tener su propio genoma, le permiten responder de manera independiente a los cambios en el potencial de las membranas, de manera rápida y directa. Así que la cantidad de ADNmit esta relacionada con la capacidad respiratoria y la disponibilidad de ATP. En procariotas, la manera de hacer esto es tener muchas copias de su genoma y varios nucleosomas asociados a la membrana plasmática, pero este no es especializado en generar energía a diferencia del ADNmit. El ADN mitocondrial permitió redistribuir la relación bioenergética del ADN, permitiendo que el genoma nuclear se especialice en nuevas funciones.
Así que sin las mitocondrias, las eucariotas no hubieran podido pagar la penalidad energética de tener un genoma tan complejo y es también esta una posible explicación de por qué no se puede encontrar un intermediario evolutivo entre los procariotas y eucariotas. Las mitocondrias permitieron incrementar la superficie de las membranas bioenergéticamente importantes. Así que las células eucariotas empezaron evolucionar a partir de la adquisición de las mitocondrias y no fue la complejidad un pre-requisito para la evolución de las mitocondrias, como normalmente se cree.
Referencia:
 Lane, N., & Martin, W. (2010). The energetics of genome complexity Nature, 467 (7318), 929-934 DOI: 10.1038/nature09486
Lane, N., & Martin, W. (2010). The energetics of genome complexity Nature, 467 (7318), 929-934 DOI: 10.1038/nature09486
El Ing. Walter Pachecho, empezó su aventura con un carrito que funcionaba con energía solar, así que pensó ¿por qué no extrapolarlo y hacerlo con este famoso medio de transporte nacional?. Sin dudas ha sido una de las noticias más citadas durante la semana (pueden leerla aquí, aquí, aquí, y aquí).