31 enero, 2011
28 enero, 2011
¿De dónde venimos los humanos modernos?
En los últimos meses se han publicado una serie de artículos que han cambiado la forma de ver nuestra evolución y la forma como se diseminaron por el mundo las primeras poblaciones de humanos modernos. Hay datos que apuntan que hubo varias olas de migraciones de humanos arcaicos, de los cuales surgieron el Neandertal, el Denisova, entre otros. Sin embargo, lo que la mayoría de teorías dice es que fueron los humanos modernos los que abandonaron África hace unos 60,000 años y empezaron a diseminarse por todo el mundo.
Sin embargo, un estudio publicado hoy en Science muestra evidencias que los humanos modernos migraron de África, mucho más antes de lo que nos imaginábamos. Un grupo internacional de científicos liderados por el Dr. Simon J. Armitage de la Universidad de London, descubrieron herramientas hechas por humanos modernos en una región llamada Jebel Faya, ubicada al sudeste de la península arábica.
Lo interesante de este descubrimiento es que estas herramientas datan de hace unos 125,000 años, más 60,000 años antes de que los humanos modernos abandonaran África, según dicen las teorías actuales. Las teorías que hoy manejamos se basan en evidencias genéticas, ya que nuestros ancestros no africanos (asíáticos, australianos, americanos) no aparecieron antes de esa época. Entonces, estas herramientas pudieron haber sido hechas por humanos arcaicos.
Actualmente, la región de Jebel Faya pertenece a los Emiratos árabes Unidos. Si los humanos llegaron a esta región hace 125,000 años, debieron tomar un atajo y atravesar el mar Rojo a través del estrecho de Bab el-Mandeb (indicado con una flechita en la figura). Y si las condiciones de esta región de la península arábica son similares a las actuales, dichas poblaciones humanas debieron haber caminado por más de 2,000 Km de inhóspitos desiertos hasta llegar a Jebel Faya.
Pero, según los datos geológicos, hace unos 130,000 años se daba fin a una era de hielo, el nivel del mar rojo pudo haber sido tan bajo que los humanos lo pudieron haber atravesado sin problemas y la península arábica, en vez de ser un gran desierto, se hubiera parecido más a la sabana africana. Todas estas condiciones podrían haber favorecido la migración de poblaciones humanas a través de esta ruta.
Sin dudas estos resultados han sido bastante controvertidos. Primero, porque las evidencias genéticas no indican la presencia de humanos modernos fuera de África, por lo menos hasta hace unos 65,000 años; y segundo, porque poblaciones de humanos arcaicos también pudieron haber producido dichas herramientas, ya que se han encontrado restos similares en Asia y Europa, que datan aproximadamente de las mismas fechas.
Por ahora, el panorama sobre nuestros orígenes y evolución no es muy claro. Un reporte del mes pasado realizado por científicos israelís muestra la presencia de fósiles de homínidos con una antigüedad de 200,000 – 400,000 años, siendo casi tan antiguos como los encontrados en África, que es donde se cree se originaron los homínidos.
Por otro lado, si vamos al análisis genético, gracias a los primeros borradores del genoma del Neandertal y del Denisova vemos que por lo menos hubieron dos mezclas entre los humanos modernos y los humanos arcaicos. Las poblaciones actuales de humanos asiáticos y europeos tienen al menos entre el 1 y 4% de ADN de Neandertal, pero no hay rastros de dicho ADN en poblaciones africanas, lo que indica que los Neandertales se desarrollaron después de que los humanos arcaicos abandonaran África y se mezclaron con las poblaciones de humanos modernos hace 50,000 a 80,000 años, al este del Oriente Medio o en Arabia. Una vez mezclados, los humanos modernos empezaron a expandirse por toda Europa y Asia.
Sin embargo, al comparar los genomas del Neandertal, Denisova y del los humanos modernos sale a la luz que las poblaciones de Neandertales y los humanos modernos debieron haberse mezclado hace 100,000 años —20,000 años más antes de lo imaginado. Esto explicaría porque los actuales melanesios poseen tanto ADN de Neandertal como ADN de Denisova (casi el 8% de su genoma corresponde a estos dos homínidos). Los Neandertales se mezclaron con los humanos modernos, luego los humanos modernos migraron hacia el este asiático y en el trayecto se encontraron con los Denisovas, luego ellos también se mezclaron, pero estas poblaciones migraron hacia el sur, hacia la Melanesia.
¿Es bastante confuso cierto? Esto se debe a que hay un problema en catalogar a estos tres homínidos como si fueran o no especies diferentes. Si citamos el concepto de especie de Darwin, este dice: “No habrá una definición que mantenga contento a todos los naturalistas”, claro, tiene mucha razón. Pero, nosotros consideramos a una población como especie según el concepto propuesto por Ernst Mayr: “ un grupo o población natural de individuos que pueden cruzarse entre sí, pero que están aislados reproductivamente de otros grupos afines”.
Entonces, si los humanos modernos y los Denisovas y Neandertales se pudieron cruzar y dejar descendencia fértil, por qué habría que considerarlos como especies diferentes, si no encajan dentro de la definición de Ernst Mayr. Es por esta razón que muchos investigadores y expertos en el tema como el paleogenetista Svante Pääbo consideran a estos grupos de homínidos como poblaciones y no como especies, ya que si nos aferramos a la definición de especie, los tres serían Hommo sapiens.
Sin embargo, en base a las características morfológicas si hay muchas diferencias entre un Neandertal y un humano moderno, ni que hablar de las capacidades cognitivas. Pero, estas diferencias, que sólo se ven a nivel fenotípico más no genotípico ¿son suficientes como para considerarlos como especies diferentes? Les agrego un dato mas: Las diferencias genéticas entre el Denisova y el Neandertal sólo son un 9% más grandes que las diferencias que hay entre un francés y un bosquimano actuales.
Finalmente, para concluir, les dejo un gráfico donde muestran las posibles formas de como se pudieron haber originado los humanos modernos, en base a todo lo que les acabo de mencionar. Cuál de los siguientes modelos creen que es el más indicado? Para mi, es aquel donde los humanos arcaicos migraron fuera del África mucho más antes de lo imaginado, y que las poblaciones que se fueron asentando en Europa y Asia, siguieron mezclándose. Esto explicaría por que encontramos fósiles de humanos tan antiguos como los encontrados en África en otras partes del mundo.
Referencias:
Armitage, S., Jasim, S., Marks, A., Parker, A., Usik, V., & Uerpmann, H. (2011). The Southern Route "Out of Africa": Evidence for an Early Expansion of Modern Humans into Arabia Science, 331 (6016), 453-456 DOI: 10.1126/science.1199113
Gibbons, A. (2011). A New View Of the Birth of Homo sapiens Science, 331 (6016), 392-394 DOI: 10.1126/science.331.6016.392
27 enero, 2011
Una hormona de crecimiento potencia la memoria en ratas
Referencia:
Chen, DY., et al.A critical role for IGF-II in memory consolidation and enhancement. Nature 469,491–4977 doi:10.1038/nature09667
26 enero, 2011
Se secuencia el genoma del orangután
Hoy se publicó en Nature el primer borrador del genoma del orangután, quien pertenece al grupo de los grandes primates y que es el menos relacionado con el hombre, con un 97% de similaridad genética. Este primer borrador estuvo lleno de sorpresas, entre ellas que la variabilidad genética de las poblaciones de las especies de Borneo (Pongo pygmaeus) y de Sumatra (Pongo abelii) es más grande que la variabilidad genética encontrada en las poblaciones humanas. Además, el genoma del orangután ha evolucionado de manera más lenta con respecto a los otros primates. La cantidad de cambios genéticos encontrados en más de 12 millones de años de evolución han sido menores a la cantidad de cambios encontrados en el hombre o los chimpancés quienes tienen menos de 6 millones de años de haber evolucionado. Entonces vamos por partes.
El orangután (orang-utan: ‘hombre de los bosques’) es el más grande de los primates con una vida arbórea, pasando más del 95% del tiempo trepado en ellos. Están divididos en dos especies: los que viven en Borneo (P. pygmaeus) y los que viven en Sumatra (P. abelii). Pero, debido a la deforestación y el deterioro de sus hábitats —por las excesivas plantaciones de la palma aceitera— sus poblaciones se han visto reducidas considerablemente. En el año 2004, se estimaron que habían entre 7,000 – 7,500 P. pygmaeus y entre 40,000 – 50,000 P. abelii; ambos considerados en como especies amenazadas por la Unión internacional para la Conservación de la Naturaleza. Conocerlos a nivel genético es de vital importancia para poder desarrollar nuevas estrategias de conservación y repoblación.
Un consorcio internacional de científicos, liderados por el Dr. Devin P. Locke de la Universidad de Washington, secuenciaron el genoma completo de una orangután llamada “Susie” y otros 10 genomas de manera parcial: cinco de Borneo y cinco de Sumatra. Este primer borrador abarca el 98.5% del genoma (3.05Gb) y la cobertura del secuenciamiento fue de 5.5X (~18Gb de información).
Al comparar el genoma del orangután con el de los otros primates observaron que la cantidad de re-arreglos genómicos era mucho menor al encontrado en los humanos o en los chimpancés. Esto quiere decir que las inversiones, translocaciones, deleciones, inserciones o duplicaciones de regiones de ADN han sido menos dinámicos. Un claro ejemplo es que sólo el 3.8% de su genoma está duplicado, mientras que en los humanos y chimpancés es mayor al 5%. Los investigadores también encontraron que la tasa de ganancia o pérdida de genes así como la tasa de mutaciones del orangután fue la mitad a la encontrada en humanos y chimpancés. Todo esto indica que los orangutanes han evolucionado muy lentamente.
Además, hay otros factores que también contribuyeron a la estabilidad del genoma del orangután. Por ejemplo, la cantidad de copias de los elementos Alu —pequeñas regiones de ADN capaces de ‘saltar’ de un lugar a otro, modificando ciertos genes, inactivándolos o creando otros nuevos— que fueron encontrados en el genoma del orangután, fue mucho menor al encontrado en los chimpancés y en los humanos. Esta sería otra de las causas de por qué la estructura del genoma del orangután ha cambiado tan poco desde que divergieron hace unos 12 – 16 millones de años.
Otra cosa extraña encontrada en el genoma del orangután es que las dos especies tienen un neocentrómero en el cromosoma #12, algo que no se encuentra en ningún otro primate. Si recordamos como es un cromosoma se nos vendrá automáticamente a la cabeza una ‘X’, el centrómero es donde se cruzan las dos líneas que forman la X, que puede estar ubicado en el centro o hacia un extremo. Por otro lado, los neocentrómeros son regiones cromosómicas diferentes a los centrómeros, tanto en estructura, secuencia y localización, sin embargo, pueden llegar a cumplir con su función y unirse a los microtúbulos durante la metafase para segregarse a cada uno de los polos en la división celular.
Al comparar aproximadamente 14,000 genes encontrados tanto en orangutanes como en humanos, chimpancés, macacos y perros, observaron que dos categorías de genes estaban sujetos a la selección natural en los orangutanes. Estos correspondían a genes envueltos en la percepción visual y en el metabolismo de los glicolípidos. En humanos, errores en los genes que están involucrados en la síntesis de glicolípidos pueden generar ciertas enfermedades neurodegenerativas. Entonces, cambios en esta vía metabólica pudieron haber sido importantes en nuestra evolución neurológica, así como en la diversidad de dietas y estrategias reproductivas.
Al analizar y comparar las secuencias de los genomas parciales de los 10 individuos tomados para el estudio (cinco de cada especie), los científicos encontraron un poco más de 13 millones de pequeñas mutaciones individuales (SNPs): 6.69 millones de SNPs dentro de los orangutanes de Borneo y 8.96 millones de SNPs en los orangutanes de Sumatra. En otras palabras, hay una mayor diversidad genética dentro de P. abelii. Esto puede ser lógico ya que su número poblacional es mayor.
En base a estos datos, los científicos pudieron calcular hace cuanto divergieron los orangutanes de Sumatra y de Borneo asumiendo una tasa de mutación de 2x10-8 y un tiempo generacional de 20 años. También hallaron el número efectivo (número de individuos que pudieron dar origen a cada una de las especies). Los datos indican que las dos especies divergieron hace unos 400,000 años y que el número poblacional del ancestro común fue de 17,000 individuos.
Es bueno tener una gran diversidad genética dentro de las poblaciones de cada una de las especies, porque esto permite que puedan soportar mejor las presiones evolutivas a las que están siendo sometidas debido a la degradación de sus hábitats naturales. Además, gracias a esta diversidad se pueden establecer estrategias de repoblación de estas especies si correr el riesgo de perder el vigor genético de los individuos. Sin embargo, los científicos no están seguros si esta diversidad genética se seguirá manteniendo si no se hace algo por mantener sus hábitats y evitar la fragmentación de sus poblaciones. A esto hay que sumar su estilo de vida arbóreo y sus tasas de reproducción lentas (8 años entre una cría y otra), convirtiéndolos en especies sumamente vulnerables.
Referencia:
Locke, D., Hillier, L., Warren, W., Worley, K., Nazareth, L., Muzny, D., Yang, S., Wang, Z., Chinwalla, A., Minx, P., Mitreva, M., Cook, L., Delehaunty, K., Fronick, C., Schmidt, H., Fulton, L., Fulton, R., Nelson, J., Magrini, V., Pohl, C., Graves, T., Markovic, C., Cree, A., Dinh, H., Hume, J., Kovar, C., Fowler, G., Lunter, G., Meader, S., Heger, A., Ponting, C., Marques-Bonet, T., Alkan, C., Chen, L., Cheng, Z., Kidd, J., Eichler, E., White, S., Searle, S., Vilella, A., Chen, Y., Flicek, P., Ma, J., Raney, B., Suh, B., Burhans, R., Herrero, J., Haussler, D., Faria, R., Fernando, O., Darré, F., Farré, D., Gazave, E., Oliva, M., Navarro, A., Roberto, R., Capozzi, O., Archidiacono, N., Valle, G., Purgato, S., Rocchi, M., Konkel, M., Walker, J., Ullmer, B., Batzer, M., Smit, A., Hubley, R., Casola, C., Schrider, D., Hahn, M., Quesada, V., Puente, X., Ordoñez, G., López-Otín, C., Vinar, T., Brejova, B., Ratan, A., Harris, R., Miller, W., Kosiol, C., Lawson, H., Taliwal, V., Martins, A., Siepel, A., RoyChoudhury, A., Ma, X., Degenhardt, J., Bustamante, C., Gutenkunst, R., Mailund, T., Dutheil, J., Hobolth, A., Schierup, M., Ryder, O., Yoshinaga, Y., de Jong, P., Weinstock, G., Rogers, J., Mardis, E., Gibbs, R., & Wilson, R. (2011). Comparative and demographic analysis of orang-utan genomes Nature, 469 (7331), 529-533 DOI: 10.1038/nature09687
Los mejores amigos: una planta carnívora y un murciélago
En el archipiélago de Borneo es común encontrar a una de las plantas carnívoras más fascinantes, las del género Nepenthes. Estas plantas poseen una hoja modificada en forma de un vaso profundo y alargado, el cual contiene una mezcla de enzimas digestivas en su interior. Los insectos —que son atraídos por sus deliciosos aromas y llamativos colores, muchos de ellos a longitudes de onda imperceptibles para el ojo humano— curiosos entran en la boca del jarro y resbalan hacia la solución digestiva, gracias a una sustancia resbalosa que es secretada por las paredes de la hoja. La presa no puede escapar y termina por digerirse al cabo de unos días, liberando los nutrientes necesarios para que la planta viva feliz.
Las nepentes pueden ser tan grandes que no sólo capturan insectos. Por ejemplo, la Nepenthes truncata puede capturar hasta pequeños roedores como los ratones.
Sin embargo, en el año 1980, un ecólogo llamado Jonathan Moran hizo una extraña observación en una variedad de la especie Nepenthes rafflesiana llamada 'elongata'. Su hoja modificada tenía la forma de un jarro más largo y estrecho que la de sus parientes más cercanas. Además carecía de aromas y colores llamativos que pudieran atraer pequeñas presas. Todo esto provocaba que la N. r. elongata capturara siete veces menos insectos que las variedades comunes. Entonces, ¿cómo hacía para sobrevivir un predador que no caza?
Más de 20 años después de la observación de Moran, otro ecólogo llamado Ulmar Grafe de la Universidad de Brunéi exploraba junto a sus colegas el bosque húedo de Borneo en busca de renacuajos dentro de las nepentes, cuando observó a un murciélago posarse dentro de una de ellas. En ese momento, el acontencimiento pasó desapercibido, pero fue meses después, cuando Grafe leyó el trabajo publicado por Moran sobre el misterio de las nepentes que a pesar de ser malas cazadoras, podían sobrevivir normalmente. Fue ahí donde supo a qué se debía.
Así que Grafe se enrumbó en otra exploración que duró 7 semanas, en la cual observaron el comportamiento de los murciélagos y su relación con las nepentes. Grafe y su equipo encontraron pequeños murciélagos de 4cm de longitud (Kerivoula hardwickii) en la cuarta parte de las N. r. elongata estudiadas.
Resulta que estos pequeños murciélagos usaban a las N. r. elongata como hogares ya que al ponerles unos radiotransmisores para estudiar su comportamiento y desplazamiento durante el día y la noche, vieron que los murciélagos dormían dentro de las plantas carnívoras y sólo lo hacían en la variedad elongata, no en otra variedad o especie. Los murciélagos eran bien selectivos con su vivienda y hasta se encontraron madres acurrucados con sus hijos murciélagos dentro de las plantas.
Esto se debe a que la variedad elongata esta diseñada de tal manera que esta especie de murciélago cabe sin problemas. El espacio ubicado encima del fluído digestivo es suficiente como para albergar hasta dos murciélagos, y el tubo es tan estrecho que el mamífero calza exacto y no termina resbalando por las parades.
Entonces, la nepente ha creado una relación simbiótica con el murciélago, dándole un lugar acogedor donde pasar la noche y recibiendo a cambio las heces del animal que son ricas en compuestos nitrogenados, un elemento vital para el desarrollo de las plantas. Por ejemplo, las hojas adyacentes a las plantas con murciélagos tienen 13% más nitrógeno que las que están adyacentes a las plantas que no tienen murciélagos. Los científicos calcularon que la N. r. elongata obtiene la tercera parte de su nitrógeno directamente del guano del murciélago. Esto lo determinaron a través de las proporciones isotópicas del nitrógeno, cuyos valores son diferentes en el guano con respecto a otra fuentes de nitrógeno.
Es posible que haya más de este tipo de relaciones entre otros animales con las plantas carnívoras y tal vez la nepente no sólo le de un lugar donde pasar la noche, sino otros beneficios más que aún estarían por descubrir. Las nepentes son plantas extremadamente exóticas, ya vimos a una que comía ratones, ahora una que sirve de hotel, que cosas más no llegaremos a encontrar, ya que hay más de 120 variedades extrañas de nepentes en Borneo. Sin dudas, la naturaleza cada día nos asombra más.
Referencia:
Grafe, TU., et al. A novel resource–service mutualism between bats and pitcher plants. Biol. Lett. (published online before print) doi:10.1098/rsbl.2010.1141 (2011)
Vía Science News. Galería de fotos: NERS.
24 enero, 2011
Un documental acerca de la biología sintética
Me llegó un correo con información referente a un proyecto independiente bastante interesante, y como todo proyecto independiente, necesita de apoyo económico para poder realizarlo. La importancia de ser un proyecto independiente es que aborda el tema —en este caso la biología sintética— desde un punto de vista imparcial. Pero, antes de comentarles más al respecto, vamos por partes…
¿Qué es la biología sintética? Aún este término no está bien definido. En mayo del 2010, Craig Venter y sus colaboradores crearon el primer genoma sintético y lo insertaron en un protoplasto bacteriano, específicamente de un Mycoplasma, y así pudo obtener un ser vivo capaz de desarrollar sus funciones normalmente usando un genoma hecho de forma artificial. ¿Esto puede ser considerado como biología sintética?, Sí, por qué no. Sin embargo, el genoma que sintetizó Venter y sus colaboradores no es nada novedoso, sino la copia de uno ya conocido.
Para muchos científicos, la biología sintética es crear nuevas funciones celulares, nuevas enzimas capaces de usar nuevos sustratos y convertirlos en nuevos productos, desarrollar nuevas vías metabólicas que no pueden ser encontrados de forma natural para poder desarrollar nuevas fuentes de energía, metabolizar nuestros contaminantes para evitar perjudicar nuestro ambiente o crear nuevos fármacos para hacer frente a las enfermedades más devastadoras.
En otras palabras, los científicos ven al ADN como un código programable, como un lenguaje de programación, tal como el que usan las computadoras, en este caso en vez de 1 y 0, son cuatro letras (A, T, C y G). De esta manera, los científicos se transforman en ingenieros… ingenieros genéticos… capaces de diseñar genes los cuales serán transformados en proteínas novedosas, con funciones especiales y valiosas para la humanidad.
Como toda nueva tecnología es bueno ver sus pros y sus contras desde un punto de vista imparcial. Por obvias razones, la biología sintética tiene un gran potencial, sobre todo económico. Aquella compañía que sea capaz de manejar el código genético a su antojo, capaz de diseñar y producir genes, proteínas y, por que no, seres vivos, tendrá un gran poder.
El documental “The Synthetic Bio” nos dará una visión clara y sencilla sobre la biología sintética, dándonos a conocer todas sus potencialidades y la forma como mejorarán la calidad de vida de las personas. Este documental hará un recorrido de los últimos 10 años, que es en los cuales se han dado los cimientos para la biología sintética, gracias al vertiginoso avance de la biología molecular. También tocará la parte ética de esta tecnología, la cual siempre generará controversia.
Este documental viene siendo desarrollado por una productora independiente llamada Field Test la cual se ha especializado en desarrollar documentales cinematográficos acerca de la ciencia moderna. Para desarrollar este documental sobre la biología sintética necesitan de $30,000 para poder terminarlo a tiempo y mandarlo al Festival de Cine de Sundance, a fines de agosto.
Para lograr este objetivo, han puesto en marcha una campaña a través de KickStarter, una plataforma virtual para conseguir donaciones y financiamientos. Tienen 45 días (desde el 19 de Enero hasta el 4 de Marzo) para conseguir los $30,000. Las donaciones van desde $1, no hay un límite máximo. Según la donación que hagas recibirás material referente al documental.
Pero no te preocupes si no tienes dinero para hacer tales donaciones o una tarjeta de crédito para hacerlo, basta con que compartas esta información y la difundas para que por ahí alguien si se anime a hacerlo. Hasta ahora van casi $4,000… falta mucho aún.
Link: http://www.kickstarter.com/projects/637230479/a-documentary-film-about-synthetic-biology
Imagen: Flickr @z33be
¿A qué se debió la extinción masiva del Pérmico?
La extinción masiva del Pérmico, que pone fin a la era geológica del Pérmico para dar paso al Triásico, fue la más devastadora de todas ya que se perdió el 96% de las especies marinas y más del 70% de las especies terrestres. Esta extinción se dio hace aproximadamente 250 millones de años, pero hasta ahora sigue siendo un misterio las causas que la dieron origen.
Las hipótesis fueron muchas, desde la colisión de un asteroide hasta la liberación abrupta del metano contenido en los hielos de los casquetes polares. Sin embargo, la teoría que tiene más fuerza es de la erupción de las escaleras siberianas que produjeron enormes flujos de basalto que liberaron cuantiosas cantidades de CO2 al ambiente durante unos 750,000 años, cambiando el clima del planeta y afectando el ciclo de carbono. Esta hipótesis ahora tiene más fuerza porque científicos Comisión Geológica de Canadá (GSC) encontraron tres distintas capas de cenizas volantes de carbono en sedimentadas de las islas árticas canadienses, cuyas rocas se habrían formado en al norte del súper-continente Pangea, justo antes de la extinción del Pérmico-Jurásico. Los resultados de sus estudios fueron publicados ayer en Nature Geoscience.
Cuando el geoquímico Stephen Grasby y sus colaboradores del GSC analizaron estas rocas que se encontraban justo en las capas anteriores a las que corresponden al fin de la era Permiana, observaron que tenían partículas inusuales, las cuales eran similares a las encontradas actualmente cerca a las plantas de energía de carbón. Estas partículas llamadas cenosferas que contenían las cenizas volantes de carbono, sólo son producidas por la combustión de este elemento. Entonces, la erupción de las escaleras siberianas liberaron estas cenizas al ambiente debido al carbón fundido que había en el magma. Fue tal la magnitud de la erupción que se liberó más de un trillón de metros cúbicos de roca fundida.
Debido al tamaño y peso de estas cenosferas, que es similar al de las cenizas volcánicas, se pudieron elevar hasta 20Km de altura y a partir de ahí propagarse por todo el mundo. Además, estas cenosferas no estaban compuesto sólo de carbono, sino también de otros metales pesados tóxicos como el cromo y el arsénico. Así que cuando estas cenosferas precipitaron y cayeron a los océanos se cree que crearon un caldo sumamente tóxico que empezó a acabar prácticamente con toda la vida que ahí existía. Además, toda la materia orgánica que se empezó a descomponer empezó a capturar el oxígeno presente en los mares, bajando peligrosamente sus niveles, y comprometiendo a los pocos sobrevivientes que quedaban.
Sin dudas es un gran descubrimiento que ayuda a entender como pudo haber ocurrido uno de los más grandes misterios de la vida: la desaparición repentina, en tan corto tiempo —sólo 1 millón de años— de más del 90% de las especies marinas y 70% de las especies terrestres. Sin dudas, los más afectados fueron los que vivían en los mares ya que no había forma de escapar a la toxicidad de su ambiente. Si bien no es la palabra final en este misterio, si nos brinda una teoría bastante plausible.
Referencia:
Grasby, SE., et al. Nature Geoscience. DOI: doi:10.1038/ngeo1069 (2011)
Vía Science News.
23 enero, 2011
EteRNA, jugando con el ARN
Aquí les presento otro juego bastante interesante y adictivo, que a parte de divertirte un rato, aprenderás mucho sobre el ARN y sus complejas formas, estructuras y funciones. El juego es libre y se juega directamente desde tu navegador.
El concepto de EteRNA es bastante similar al de Foldit: en vez de plegar proteínas, vamos a plegar moléculas de ARN. ¿Plegar moléculas de ARN?… Sí. Tal vez la visión que la mayoría tiene sobre las moléculas de ARN es de una larga cadena de nucleótidos distribuidos linealmente. Sin embargo, a pesar de ser una cadena de hebra simple, puede formar interacciones entre los nucleótidos complementarios (Adenina con Uracilo y Guanina con Timina). Así que estas interacciones doblarán y formaran horquillas, bucles, y una gran diversidad de formas complejas, tan complejas como las formas que adquieren las proteínas. Es así como se forman los ARN ribosomales o los ARN de transferencia.
Por ejemplo, los ARN ribosomales tienen una estructura compleja y es gracias a ella que los ribosomas tienen la capacidad de leer el ARN mensajero y traducirlo a proteína.
La idea del juego es diseñar estructuras complejas y estables de ARN. Para ello, tendrás la capacidad de cambiar los nucleótidos que se te apetezcan. Para no complicar el juego, los nucleótidos están representados por colores. De esta manera, el rojo (G) se complementará con el verde (C) y el amarillo (A) se complementará con el celeste (U). Cada tipo de apareamiento tendrá su valor energético que te permitirá ver si tu estructura es estable o termodinámicamente posible. Así que, el enlace rojo-verde, será más fuerte porque la Guanina y la Citosina forman tres puentes de hidrógeno, mientras que la adenina y uracilo sólo dos.
Lo más interesante del juego es que antes de desarrollar cada puzle te dan una pequeña introducción respecto a la estructura que formarás, la función biológica que cumple, las interacciones con otras proteínas, o si es un ARN interferencia, te indican que genes pueden ser silenciados por esa estructura.
Para hacer más adictivo el juego, cada vez que resuelvas un puzle te darán dinero, el cual te permitirá subir dentro del ranking del juego. También cuenta con un pequeño chat para que conozcas a gente de otros lugares y tal vez por ahí conozcas al director de un centro de investigación importante y te jale para su laboratorio.
¿Y cuál es la misión del juego? No, no es sólo entretener a los biólogos y otros geeks. Tras esta inocente interfaz hay un plan macabro… Desarrollar la primera librería de estructuras de ARN sintéticos a gran escala. Gracias a las estructuras que diseñemos a través del juego, los investigadores que están tras este proyecto podrán determinar si sus modelos se pliegan adecuadamente y así saber si son biológicamente activos.
Además, los jugadores —sin darse cuenta— crearán nuevas moléculas de ARN, con estructuras que tal vez no encontremos de manera natural, pero que podrían llegar a tener una función dentro de las células. Este interés viene debido a que en los últimos años se ha estudiado mucho acerca de la capacidad de estas moléculas de ARN no codificante (microARNs, ARN pequeño nuclear, ARN largo no codificante, ARN de pequeña horquilla, etc.) de regular la expresión de muchos genes, entre ellos oncogenes, factores de supresión de tumores o genes virales; los cuales podrían ser usados para desarrollar agentes terapéuticos para tratar el cáncer y otras enfermedades como el VIH.
Así que a matar el tiempo colaborando con la ciencia.
Link: http://eterna.cmu.edu/
22 enero, 2011
¿Cuál es la molécula sintética más grande que existe?
Uhm… difícil pregunta. Bueno, en la naturaleza existen muchas moléculas de gran tamaño, un claro ejemplo son las proteínas o el ADN, y son grandes debido a que están formados por la unión de muchas moléculas más pequeñas. Las proteínas están formadas por la unión de aminoácidos, y el ADN por la unión de nucleótidos.
Gracias a la química sintética se han podido crear también moléculas sumamente grandes y complejas, muchas de ellas son usadas actualmente como agentes terapéuticos, insecticidas, antibióticos, etc. Pero es muy difícil construir una molécula que sea grande y estable a la vez. Una forma de hacerlo es a través de la polimerización, que es la unión consecutiva de pequeñas moléculas hasta formar una cadena. Es así que se construyó la molécula sintética más grande conocida: el poliestireno, la cual tiene la masa de 40 millones de átomos de Hidrógeno.
 Recientemente Dieter Schlüter y colaboradores del Instituto Tecnológico de Zúrich, crearon la molécula sintética más grande conocida hasta ahora, desplazando de su trono al poliestireno. Su nombre es PG5, tiene la masa de 200 millones de átomos de Hidrógeno y mide aproximadamente 10 nm (0.00000001m). Bueno, sé que 10 nanómetros no es algo grande, pero a nivel molecular, lo es. En otras palabras, es tan grande como la cápside de un virus. En la figura podemos apreciar una comparación entre los tamaños del virus del mosaico del tabaco con la PG5. A diferencia de PG5, un virus está formado por miles de moléculas, como las glicoproteínas y ácidos nucleicos.
Recientemente Dieter Schlüter y colaboradores del Instituto Tecnológico de Zúrich, crearon la molécula sintética más grande conocida hasta ahora, desplazando de su trono al poliestireno. Su nombre es PG5, tiene la masa de 200 millones de átomos de Hidrógeno y mide aproximadamente 10 nm (0.00000001m). Bueno, sé que 10 nanómetros no es algo grande, pero a nivel molecular, lo es. En otras palabras, es tan grande como la cápside de un virus. En la figura podemos apreciar una comparación entre los tamaños del virus del mosaico del tabaco con la PG5. A diferencia de PG5, un virus está formado por miles de moléculas, como las glicoproteínas y ácidos nucleicos.
Los investigadores del Instituto Tecnológico de Zúrich llevaron la síntesis química al máximo, con varias rondas de polimerización para formar el esqueleto principal hecho a base de Carbono e Hidrógeno. Luego se le añadieron las demás moléculas a manera de ramas, entre ellas anillos bencénicos, compuestos nitrogenados y otros hidrocarburos. Y después se le añadió otras pequeñas moléculas a manera de sub-ramas. Finalmente se obtuvo una estructura similar a un árbol en otoño la cual requirió la formación de 170,000 enlaces químicos.
Esta molécula es tan grande, compleja y estable que puede cargar drogas en sus pliegues y formar enlaces químicos con muchos tipos de moléculas diferentes, lo cual la hace una buena candidata para el trasporte de nuevos agentes terapéuticos con una mayor especificidad.
Referencia:
Zhang, B., Wepf, R., Fischer, K., Schmidt, M., Besse, S., Lindner, P., King, B. T., Sigel, R., Schurtenberger, P., Talmon, Y., Ding, Y., Kröger, M., Halperin, A. and Schlüter, A. D. (2011), The Largest Synthetic Structure with Molecular Precision: Towards a Molecular Object. Angewandte Chemie. International Edition, 50: 737–740. doi: 10.1002/anie.201005164
Vía New Scientist.
21 enero, 2011
Si escribes lo que has estudiado, aprenderás mejor
Tal vez este es el estudio que demuestra por qué los que hacen su “plaje” salen mejor en los exámenes que los que estudian a conciencia. Claro, muchos dirán que salen mejor porque hacen trampa y usan sus plajes durante el examen. Pero, se han visto casos —y tal vez les ha pasado a algunos— en que uno logra aprender lo que ha escrito en ese pequeño papelito de autoayuda que ya no lo llegan a utilizar durante el examen.
Para los no conocedores: En Perú y algunos países de Latinoamérica, “Plajear” es uno verbo que no esta definido por la RAE pero que deriva del verbo Plagiar.
Plajear suele ser el acto de copiar durante un examen, ya sea de otra persona o de alguna superficie sólida que sirve como soporte para la escritura de fórmulas, teorías, conceptos o definiciones. Entre las superficies sólidas más comunes tenemos: las hojas de papel de tamaños reducidos, el reverso de las calculadoras, las paredes, el antebrazo, etc.
Plajero, es la persona que plajea.
Un par de científicos de la Universidad de Purdue, demostraron que aquellos estudiantes que se tomaron unos minutos para hacer un mapa conceptual o escribir un pequeño ensayo de lo que habían leído, una semana después, rindieron un mejor examen. Los resultados de esta investigación fueron publicados ayer en la versión online de Science.
Los participantes fueron 200 estudiantes de diferentes colegios a los cuales se les dividió en cuatro grupos. A cada grupo se le pidió que usaran una estrategia diferente de estudio. Las estrategias eran: el estudio simple (leer el artículo una vez), el estudio repetitivo (leer el artículo repetidas veces), hacer un mapa conceptual después de leer el artículo, y escribir un pequeño ensayo sobre el artículo que acababan de leer.
Los últimos fueron los que obtuvieron los mejores resultados, tanto en los exámenes con preguntas concretas o literales, como en los exámenes con preguntas para pensar o inferir. Sin embargo, en los exámenes que tenían preguntas metacognitivas, los que hicieron el estudio repetitivo fueron los que mejor salieron.
Entonces, los plajeros, de alguna manera y sin darse cuenta están aprendiendo a través de la práctica de la recuperación de la memoria. O sea, estudiar no significa meter los conocimientos al cerebro (tampoco en un papel de 10x10cm), sino tener la capacidad de recuperar esos conocimientos en el momento oportuno. Para ello, cuando uno estudia, es bueno escribir todo lo que recuerde y centrarse en eso, dejando a un lado el libro y los cuadernos por un momento.
Esta práctica ejercita al cerebro para que recuerde y recupere lo leído minutos antes, y cuando escribimos lo aprendido, estamos haciendo una especie de retroalimentación (feedback), que favorece el aprendizaje. Pero, ¿por qué es mejor escribir que hacer un mapa conceptual? No se sabe, tal vez la síntesis que uno busca al elaborar un mapa conceptual hace que se pierda los conectores claves para darle sentido a la idea que intentamos plasmar, y por eso tendemos a olvidarlo más fácilmente.
Así que si quieren salir bien en sus próximos exámenes, estudien, hagan su plaje y escriban sus miedos minutos antes del examen. Y si no recuerdan alguna fórmula o concepto, usen su plaje. Pero sólo en casos de emergencia.
Referencia:
Jeffrey D. Karpicke & Janell R. Blunt. Retrieval Practice Produces More Learning than Elaborative Studying with Concept Mapping. Science. DOI: 10.1126/science.1199327. Published Online 20 January 2011.
¿Cómo entra el parásito de la malaria a los glóbulos rojos?
 Fue así que investigadores australianos liderados por el Dr. David T. Riglar usaron los ensayos de inmunofluorescencia e iluminación estructurada 3D para relacionar estos eventos estructurales con los eventos moleculares. De esta manera pudieron registrar y entender cómo el mecanismo de ingreso de P. falciparum a los glóbulos rojos a nivel molecular. El trabajo fue publicado ayer en la revista Cell Host & Microbe.
Fue así que investigadores australianos liderados por el Dr. David T. Riglar usaron los ensayos de inmunofluorescencia e iluminación estructurada 3D para relacionar estos eventos estructurales con los eventos moleculares. De esta manera pudieron registrar y entender cómo el mecanismo de ingreso de P. falciparum a los glóbulos rojos a nivel molecular. El trabajo fue publicado ayer en la revista Cell Host & Microbe.Como se puede ver en la imagen, la proteína PfRON4 de P. falciparum es la responsable de formar las uniones oclusivas, luego forma una especie de anillo gracias a las enzimas secretadas por las roptrias, y por ahí penetra el merozoito, que es visualizado de color azul, que es una molécula fluorescente que se une al ADN. Con estas imágenes hicieron una reconstrucción en 3D.
 |
| [Click para agrandar] |
Referencia:
20 enero, 2011
Una ameba es el agricultor más primitivo
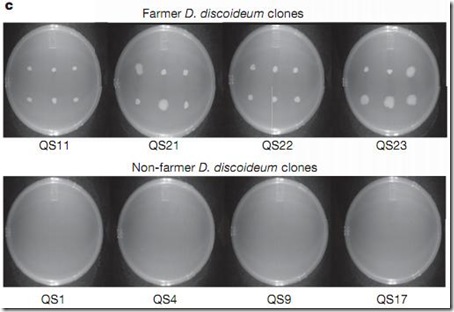
Pero, ¿cómo estos simples organismos pueden llegar a ser agricultores? Hasta ahora se conocen solo unas cuantas especies que han desarrollado la agricultura. Por ejemplo, las hormigas cultivadoras de hongos, quienes se encargan de dispersar y sembrar las semillas —que vienen a ser las esporas de los hongos— y luego las cultivan alimentándolas con las hojas que recolectan del bosque, así tienen una constante fuente de alimento.
Pero, sólo el 36% de sus clones recolectados tenían esta capacidad. Así que Brock analizó sus secuencias de ADN mitocondrial y ribosomal para determinar si sus clones eran de la misma especie o no, los resultados arrojaron que no habían diferencias entre ellas, correspondiendo a la misma especie. Por otro lado, al analizar el ADN ribosomal de las bacterias presentes en los soros vieron que ellas si pertenecían a diferentes especies, y muchas de ellas formaban parte de la dieta principal de Dicty.
19 enero, 2011
Desde el blog de Yana – Ahorro de energía eléctrica
Miguel Figueroa, más conocido como “el Yana”, es biólogo de la Universidad Nacional Agraria La Molina especializado en agrobiodiversidad, y quien posee un magistral talento para contar las historias más alocadas las cuales cargan un mensaje subliminal. Esta habilidad ha sido muy bien aprovechada para fomentar la educación ambiental en nuestro país, a través de una serie de “mini-programas” que tratan de explicar —de manera sencilla y entretenida— las formas como podemos contribuir con el cambio.
Para su sexta entrega, Yana nos da algunas recomendaciones para ahorrar energía, explicando por qué ese foco Phillips amarillo de 100W contribuye con la contaminación del ambiente, al igual que no saber organizar la ropa para al momento de plancharla.
Saque su cuenta no más cuanta energía es desperdiciada en forma de calor por culpa de estos focos. Un foco debe cumplir una única función que es de iluminar, el calor generado es energía perdida. Por otro lado, eviten planchar la ropa por las noches, ya que es la hora en que más energía se está consumiendo. Háganlo durante el día.
Puedes ver sus anteriores programas en: http://elblogdeyana.blogspot.com/
Ataque doble contra el VIH

Lo que hicieron los científicos del Instituto de Investigación Beckman, liderados por el Dr. John Rissi, fue construir una molécula de ARN que puede atacar al VIH mediante dos mecanismos: reprimiendo la expresión de genes necesarios para su multiplicación y bloqueando las proteínas específicas de membrana. Los resultado son favorables, al menos en ratones, según reportaron los investigadores en Science Translational Medicine.
¿Cómo funciona? Esta molécula de ARN fue construida a partir de dos tipos de ARN: uno es un pequeño ARN de interferencia (ARNi), el cual bloquea la expresión de los genes que el virus necesita para multiplicarse llamados tat y rev; mientras que el otro es ARN conocido como aptámero que se une a una proteína encontrada en la superficie de los virus y de las células infectadas llamada gp120. Éste aptámero tiene dos funciones: depositar el ARNi en la célula infectada y neutralizar los virus que flotan en la sangre a través de la ligación con las proteínas gp120.
Este ARN quimera (una mezcla de dos tipos de ARN completamente diferentes) fue insertado en un ratón sensible al VIH. Los investigadores observaron que la carga viral en sangre disminuyó considerablemente, lo que indica que el aptámero estaba cumpliendo bien su función de neutralizar a los virus e impedir que sigan infectando otras células. Luego, cuando midieron los niveles de expresión de los genes virales tat y rev en los linfocitos de los ratones infectados, vieron que éste se redujo entre el 75 y 90% con respecto a los ratones que no recibieron el tratamiento. Esto indica que el ARNi logró entrar a las células y estaba cumpliendo con la función para la cual fue programada. Por lo tanto, la quimera tuvo un efecto más potente comparado con el uso del aptámero o el ARNi individualmente.
Sin embargo, a pesar que el diseño de esta molécula sea hermoso, la acción del ARNi no es muy importante. La carga viral no se redujo significativamente cuando compararon el ARN quimera con el aptámero solo. La idea no es que el virus deje de replicarse —aunque es importante para controlar la diseminación del virus— sino eliminar todas las partículas virales de la sangre, algo que hasta ahora no ha podido ser obtenido por ningún nuevo tratamiento en desarrollo.
Pero si se combina el uso del ARN quimera con los retrovirales que se usan actualmente, podría prevenir la emergencia de cepas resistentes a los tratamientos ya que los virus, a pesar que adquieran mutaciones que le confieran resistencia, no podrán diseminarse porque la replicación de su ADN ha sido bloqueada.
De todas maneras es una buena estrategia, porque se podría crear un ARN quimera que, a parte de cumplir con estas dos funciones, induzca la muerte celular (apoptosis) en las células infectadas, de esta manera, se podría librar al organismo de las partículas virales y finalmente curar al paciente. Aún falta mucho por investigar, pero es interesante avance.
Vía Nature News.
Imagen: Flickr @spoffy.