Esta sin dudas ha sido la noticia de la semana, aunque me da pena decirlo, en muchos de los medios de comunicación la información está completamente alejada de la realidad. En primer lugar, no se ha creado una forma de vida, ni se ha creado la primera bacteria artificial. Lo que Venter et al. hicieron fue crear el primer organismo vivo—en este caso una bacteria— controlado completamente por un genoma artificial. A grandes rasgos, lo que hicieron fue sintetizar químicamente un genoma (ADN) de 1.08 millones de pares de base (pb) e insertarlo dentro de una bacteria (Mycoplasma) para que este genoma artificial pase a controlar todo el funcionamiento del microorganismo, como lo haría un genoma natural. Este trabajo fue publicado en la versión on-line de Science (Sciencexpress) y lo pueden descargar libremente.
Se ha avanzado mucho en la genómica desde que Sanger secuenció el primer genoma —del fago φX174— en 1977. Los fagos son virus que atacan a las bacterias y tienen genomas muy chiquitos. En 1995, Fleischmann et al. secuenciaron el genoma de Haemophilus influenzae, una bacteria con un genoma más grande y complejo que del fago φX174. Todas las secuencias generadas se empezaron a digitalizar; pero, ¿sería posible usar estas secuencias para sintetizar químicamente un ADN? De ser posible, ¿funcionaría como uno natural si es insertado en un ser vivo?. Estas preguntas ya tienen una respuesta.
Entonces, en forma general el trabajo consiste primero en hacer la síntesis química de un genoma chiquito, el más pequeño posible. Mycoplasma genitalium es la bacteria con uno de los genomas más pequeños conocidos hasta ahora, el cual tiene ~580000pb (580Kb) que codifica para unas 485 proteínas, siendo 100 de ellas prescindibles si son eliminadas de una a la vez. Luego, dividirlos en pequeños bloques (cassettes) para poder insertarlos en vectores que permitan meter estas secuencias en el microorganismo. Después, debemos ensamblar todos los cassettes hasta completar el genoma artificial, usando un organismo diferente que tenga la capacidad de hacer este trabajo. Finalmente, purificar el genoma artificial intacto y trasplantarlo a la célula receptora. Venter et al. tuvieron que diseñar mecanismos y protocolos para poder realizar este trabajo.
El primer tropezón que encontraron fue que M. genitalium tenía una tasa de crecimiento extremadamente baja. Así que tuvieron que optar por otros Mycoplasmas con tasas de crecimiento más rápidas. Los investigadores usaron a un M. mycoides como donador y a M. capricolum como receptor. Sin embargo, al hacer los primeros intentos de trasplante del genoma de M. mycoides —en forma de cromosoma bacteriano— fallaron debido a que el genoma del donador y del receptor se encuentran metilados, protegiéndolos de la acción de las enzimas de restricción que actúan como mecanismo de defensa, cortando y degradando el ADN extraño (sin metilar). Debido a que los cromosomas bacterianos los hacen crecer como plásmidos en las levaduras (porque son más fáciles de cultivar), el ADN no está metilado y cuando es trasplantado al receptor, cae víctima de las enzimas de restricción. Los investigadores usaron dos estrategias: metilar el ADN usando metilasas purificadas o romper el sistema de enzimas de restricción del receptor.
Una forma más fácil de explicar esto de la metilación… En una discoteca (Mycoplasma), las personas que están en la sección VIP tienen una pulsera que los identifica (genoma del Mycoplasma metilado); si alguien que no es de VIP entra (genoma de Mycoplasma no metilado desarrollado en la levadura), automáticamente el personal de seguridad (enzimas de restricción) lo identificarán porque no tiene la pulsera y lo sacarán a patadas. Así que para evitar esto, el que quiera entrar a VIP deberá pagar más y recibir su pulsera (meilación) o dopar a los elementos de seguridad para que ya no cuiden y puedan entrar a VIP sin miedo de que los boten a patadas.
Una vez que solucionaron estos inconvenientes diseñaron el genoma artificial a partir de las secuencias de dos cepas de M. mycoides. A este genoma artificial le llamaron JCVI-syn1.0 el cual se diferenciaba en sólo 19 nucleótidos del genoma de referencia y tenían 4 secuencias “marca de agua” que serán usados más adelante para la identificación del genoma artificial. Este genoma artificial tenía un tamaño de 1.08Mb (1080000pb). Como el genoma es demasiado grande como para ser sintetizado químicamente de un paso, se dividió en pequeñas porciones (cassettes) de 1.08Kb (1080pb) cada una. Cada cassette tenía 80pb repetidos en cada extremo, para identificar el orden en que serán pegados uno con otro. Además cada cassette tenía un sitio de corte para una enzima de restricción para poder ser clonado en un vector (plásmido usado para insertar genes) e introducido a una levadura. El ensamblaje de los ~1000 cassettes se hizo en 3 etapas:
-
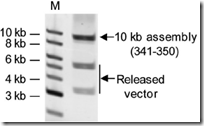
La primera etapa fue el primer ensamblaje de intermediarios sintéticos de 10Kb (~10 cassettes). Los cassettes fueron puestos en vectores y recombinados en las levaduras para formar fragmentos de 10Kb (10000pb). Luego, los vectores cargando los fragmentos de 10Kb fueron transferidos a
E. coli para ser purificados y analizados. Al menos uno de cada 10 levaduras clonadas tenían los fragmentos de 10Kb ensamblados.
-

La segunda etapa fue el ensamblaje de intermediarios sintéticos de 100Kb. Se repitió el paso anterior; sin embargo, cuando los fragmentos de 100Kb (100000pb) fueron transferidos a
E. coli no pudieron permanecer estables, así que se tuvieron que aislar y purificar directamente desde la levadura. Para determinar si los vectores contenían los fragmentos de 100Kb, se hizo una PCR multiple. Cada fragmento de 10Kb tenía una secuencia única de amplificación y al hacer la PCR se debían obtener 10 bandas. El 25% de las levaduras clonadas tenían el fragmento de ~100Kb. En total fueron 11 intermediarios de ~100Kb.
-
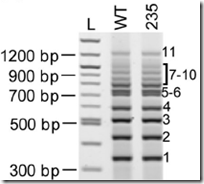
La tercera etapa era del ensamblaje final. Para esta etapa final, se purificaron los 11 intermediarios de ~100Kb, se capturaron en “plugs” de agarosa, sometieron a una enzima de restricción para liberarlos de sus vectores, se aislaron los fragmentos libres mediante una electroforesis y se insertaron en una levadura (transformación genética). Una vez dentro, los 11 fragmentos se autoensamblaron sin la necesidad de algún vector —como en los dos casos anteriores— y formaron un cromosoma único de 1.08Mb (cromosoma bacteriano de
M. mycoides sintético). Para seleccionar las levaduras que habían ensamblado bien los 11 fragmentos de 100Kb se hizo nuevamente una PCR multiple. Cada fragmento de 100Kb tenía una secuencia única de amplificación y al hacer la PCR se debían obtener 11 bandas. Además, para demostrar el correcto ensamblaje se identifico las secuencias “marca de agua” en el JCVI-syn1.0.

 Una vez ensamblado todo el genoma sintético se procedió a trasplantarlo dentro del receptor M. capricolum. Para seleccionar a las colonias que habían aceptado al genoma sintético se usaron medios de cultivo selectivos con antibióticos (tetraciclina) y colorantes (X-gal). Si en la placa petri aparecían colonias y eran de color azul, significaba ÉXITO, las colonias tenían el genoma sintético insertado y funcionando. Y así fue!!!… se logró obtener colonias de M. capricolum que contenían un genoma completamente artificial (sintetizado químicamente) y funcional, además, tenían la capacidad de replicarse. De ahí el término de ‘casi’ sintéticos.
Una vez ensamblado todo el genoma sintético se procedió a trasplantarlo dentro del receptor M. capricolum. Para seleccionar a las colonias que habían aceptado al genoma sintético se usaron medios de cultivo selectivos con antibióticos (tetraciclina) y colorantes (X-gal). Si en la placa petri aparecían colonias y eran de color azul, significaba ÉXITO, las colonias tenían el genoma sintético insertado y funcionando. Y así fue!!!… se logró obtener colonias de M. capricolum que contenían un genoma completamente artificial (sintetizado químicamente) y funcional, además, tenían la capacidad de replicarse. De ahí el término de ‘casi’ sintéticos.
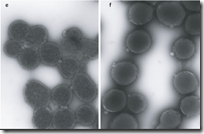 Pero este M. mycoides derivado de un genoma sintético, será fisiológica y morfológicamente similar al original o presentará diferencias significativas. Para esto se hizo un estudio proteómico y se observó que el patrón de puntos obtenidos en la electroforesis 2D (bidimensional) de la cepa silvestre, fue similar al de la cepa sintética. Y al tomar imágenes con un microscopio electrónico, la morfología de las dos cepas fueron idénticas. La única diferencia fue que la cepa sintética tenía una tasa de crecimiento ligeramente superior a la cepa silvestre, de ahí, ningún cambio significativo entre una y la otra.
Pero este M. mycoides derivado de un genoma sintético, será fisiológica y morfológicamente similar al original o presentará diferencias significativas. Para esto se hizo un estudio proteómico y se observó que el patrón de puntos obtenidos en la electroforesis 2D (bidimensional) de la cepa silvestre, fue similar al de la cepa sintética. Y al tomar imágenes con un microscopio electrónico, la morfología de las dos cepas fueron idénticas. La única diferencia fue que la cepa sintética tenía una tasa de crecimiento ligeramente superior a la cepa silvestre, de ahí, ningún cambio significativo entre una y la otra.
Para concluir, este ha sido uno de los más grandes avances de la ciencia en los últimos 10 años, comparado con lo que fue en su tiempo la síntesis de la urea (una sustancia orgánica) a partir de compuestos inorgánicos hecho por Wöhler en 1828. Este trabajo será considerado como un hito en la historia de la ciencia de los próximos años, y será el punto de partida para todo el desarrollo de la biología sintética. Imagínense diseñar desde cero un microorganismo capaz de degradar el petróleo y sus compuestos derivados, usando secuencias tomadas del GenBank, para sintetizar químicamente este genoma, insertarlo en un receptor y tener un ser vivo capaz de solucionar problemas tan graves como lo ocurrido en el Golfo de México.
Referencia:
 Gibson, D., Glass, J., Lartigue, C., Noskov, V., Chuang, R., Algire, M., Benders, G., Montague, M., Ma, L., Moodie, M., Merryman, C., Vashee, S., Krishnakumar, R., Assad-Garcia, N., Andrews-Pfannkoch, C., Denisova, E., Young, L., Qi, Z., Segall-Shapiro, T., Calvey, C., Parmar, P., Hutchison, C., Smith, H., & Venter, J. (2010). Creation of a Bacterial Cell Controlled by a Chemically Synthesized Genome Science DOI: 10.1126/science.1190719
Gibson, D., Glass, J., Lartigue, C., Noskov, V., Chuang, R., Algire, M., Benders, G., Montague, M., Ma, L., Moodie, M., Merryman, C., Vashee, S., Krishnakumar, R., Assad-Garcia, N., Andrews-Pfannkoch, C., Denisova, E., Young, L., Qi, Z., Segall-Shapiro, T., Calvey, C., Parmar, P., Hutchison, C., Smith, H., & Venter, J. (2010). Creation of a Bacterial Cell Controlled by a Chemically Synthesized Genome Science DOI: 10.1126/science.1190719