Simples mutaciones, que en principio son degenerativas, aumentan el grado de complejidad de las maquinarias moleculares.
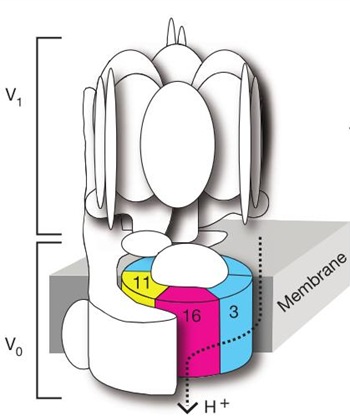
Esta maravilla que vemos en la figura superior es la ATPasa vacuolar (ATPasa-V), y no es más que una “máquina” que usa la energía provista por el ATP para bombear protones —átomos de hidrógeno sin su electrón (H+)— hacia el interior de ciertos organelos, con el fin de intercambiarlos después por otras sustancias, tales como: iones de calcio, neurotransmisores y hormonas; o también acidificar las cabezas de los espermatozoides (acrosomas) para facilitar la penetración del óvulo.
Al igual que muchas maquinarias celulares, la ATPasa-V está conformada por varias proteínas, lo que la convierte en una molécula sumamente compleja. La región V1 —compuesta por ocho proteínas— hidroliza el ATP y provee de energía al anillo V0 —conformado por seis proteínas— para que rote y mueva los protones hacia el interior de los organelos.
Pero, ¿cómo se alcanzó tal complejidad? Todo parte de un ensamblaje muy simple y primitivo que va adquiriendo nuevas partes durante su evolución, confiriéndole nuevas funciones con un grado de complejidad cada vez superior. El problema radica en cómo se adquieren dichas partes.
La evolución nos dice que este proceso empieza por la duplicación de un gen. Luego, las mutaciones tomarán un rol protagonista, modificando y combinando sus secuencias, por lo tanto, su función. Al final del camino, y si la selección natural se lo permite, estos nuevos genes originados del mismo gen ancestral (parálogos), codificarán proteínas que cumplirán con tareas específicas diferentes, aumentando la complejidad del sistema.
Sin embargo, explicarlo de esta manera es bastante abstracto. Lamentablemente, no podemos seguir la evolución de una maquinaria molecular compleja “paso a paso”, porque no disponemos de millones de años para hacerlo. Entonces, ¿habrá alguna manera de “ver la evolución” en el laboratorio? Pues sí.
Un grupo de investigadores del Instituto de Biología Molecular de la Universidad de Oregón, liderados por Gregory Finningan y Joe Thornton, resucitaron las proteínas ancestrales que formaron parte del anillo V0 primitivo y las expresaron en una levadura, observando que lograban ensamblarse correctamente para formar una ATPasa-V funcional. Según el artículo publicado el 9 de Enero en Nature, los investigadores también determinaron las mutaciones que condujeron al grado de complejidad observado en la actualidad.
Aunque la ATPasa-V está presente en todas las células eucariotas, el anillo V0 varía dependiendo del linaje. En los animales y la mayoría de eucariotas, el anillo V0 está compuesto por dos subunidades (una Vma16 y cinco Vma3); mientras que en los hongos, está compuesto por tres (una Vma16, una Vma11 y cuatro Vma3). Todas estas proteínas son codificadas por genes parálogos, que como explicamos hace algunos párrafos, se originaron a partir del mismo gen ancestral.
Desde 1990, los científicos han sido capaces de resucitar proteínas ancestrales. En principio, la metodología se basa en comparar la secuencia de aminoácidos de una determinada proteína en diferentes organismos —algunos serán más primitivos que otros, por lo tanto, sus secuencias también lo serán. Estas diferencias proveen a los investigadores de claves de cómo las secuencias cambian: habrán aminoácidos que serán sustituidos por otros con una mayor facilidad. A esto le llamamos, parsimonia.
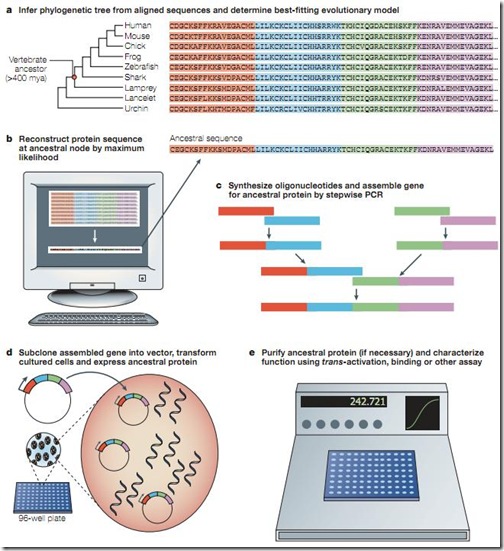
A inicios del siglo XXI, Thornton desarrolló un algoritmo computacional que le permitía reconstruir proteínas ancestrales de una manera más rápida. Lo primero que se hace es alinear y comparar las secuencias en base a su filogenia (a). Luego se analizan los cambios más probables y se reconstruye la proteína ancestral (b). Después se pasa a una secuencia de nucleótidos en base al código genético. Y, finalmente, se sintetiza el gen (c) para introducirlo y en un organismo modelo (d), quien se encargará de expresarlo para hacer los análisis respectivos (e).
Entonces, usando las secuencias de las proteínas Vma16 (fucsia), Vma3 (celeste) y Vma11 (amarillo), Finningan y sus colaboradores reconstruyeron los genes ancestrales. Al compararlas, los investigadores observaron que Vma3 y Vma11 son proteínas hermanas, que compartieron una proteína ancestral común hace más de 800 millones de años. A su vez, Vma3/Vma11 y Vma16 también se originaron a partir de un mismo ancestro común, tal vez a inicios del origen de los eucariotas.
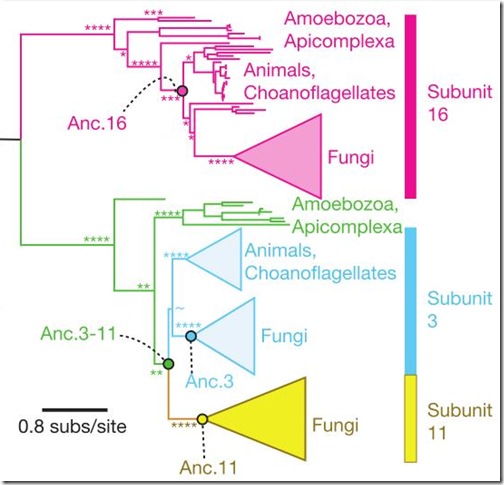
Usando el algoritmo computacional, los investigadores lograron reconstruir cuatro proteínas ancestrales: Anc.3 (ancestro de todas las Vma3 de hongos), Anc.11 (ancestro de todas las Vma11), Anc.16 (ancestro de todas las Vma16) y Anc.3-11 (ancestro de Anc.3 y Anc.11). Luego, insertó los genes correspondientes en diferentes levaduras.
Los resultados fueron asombrosos. Cuando las levaduras carecían de las proteínas Vma3, o la Vma11, o ambas a la vez, no podían desarrollarse ni formar colonias. Sin embargo, cuando se les insertó el gen que codifica para la proteína Anc.3-11, el crecimiento de estas levaduras fue restaurado: la Anc.3-11 tomó el papel de la Vma3 y Vma11. El mismo resultado se obtuvo cuando pusieron el gen Anc.16 en levaduras deficientes para la subunidad Vma16. Y, por si fuera poco, al poner las proteínas Anc.3-11 y Anc.16 en levaduras deficientes para las tres subunidades del anillo V0, éstas pudieron crecer con toda normalidad. Además, un análisis de acidificación demostró que la ATPasa-V “ancestral” era completamente funcional.
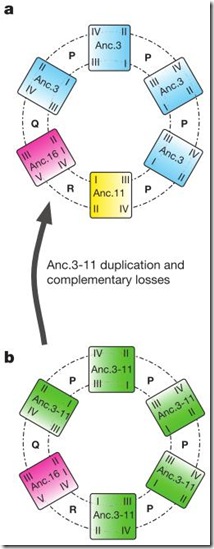 Con estos resultados Finningan y sus colegas pudieron revelar cómo evolucionó el anillo V0 para volverse cada vez más complejo. Al inicio, el anillo estaba formado por un sólo tipo de subunidad: la proteína más ancestral de todas (digamos la “Anc.3-11-16”). Luego, ésta se duplicó y cada una empezó a adquirir mutaciones que las diferenciaron mutuamente. Una pasó a formar la subunidad Anc.16 y la otra la subunidad Anc.3-11 (Figura b). Anc.3-11 tenía la capacidad de interactuar con otra Anc.3-11 (enlace P) y con la Anc-16, por cualquiera de los dos lados (enlace Q y R).
Con estos resultados Finningan y sus colegas pudieron revelar cómo evolucionó el anillo V0 para volverse cada vez más complejo. Al inicio, el anillo estaba formado por un sólo tipo de subunidad: la proteína más ancestral de todas (digamos la “Anc.3-11-16”). Luego, ésta se duplicó y cada una empezó a adquirir mutaciones que las diferenciaron mutuamente. Una pasó a formar la subunidad Anc.16 y la otra la subunidad Anc.3-11 (Figura b). Anc.3-11 tenía la capacidad de interactuar con otra Anc.3-11 (enlace P) y con la Anc-16, por cualquiera de los dos lados (enlace Q y R).
Y entonces ocurrió lo inevitable… Hace unos 800 millones de años (después que los animales y los hongos se separaran), Anc.3-11 se duplicó y cada una empezó a adquirir nuevas mutaciones. Uno de los genes perdió su capacidad de unirse con la Anc.16 mediante el enlace Q (Anc.11), y el otro mediante el enlace R (Anc.3), pero si conservaron la capacidad de unirse entre sí mediante el enlace P (Figura a).
Fue así como evolucionaron las tres subunidades que ahora observamos en el anillo V0 de la ATPasa-V de los hongos. Sin embargo, aún faltaba entender qué mutaciones condujeron a esta diferenciación y especialización de funciones.
Al comparar las secuencias de aminoácidos de Anc.3-11 con Anc.3 y Anc.11, los investigadores encontraron 25 y 31 sustituciones, respectivamente. Para determinar cuál de estos cambios fue el responsable de la divergencia de estas proteínas hermanas, Finningan y su equipo manipularon el gen Anc.11-3 e probaron cada una de las mutaciones.
Los resultados mostraron que sólo dos sustituciones de aminoácidos fueron los responsables de toda esta especialización y complejidad del anillo V0. El cambio de una valina por una fenilalanina en la posición 15 hacía que Anc.11-3 perdiera su capacidad de funcionar como Anc.3; mientras que el reemplazo de una metionia por una isoleucina en la posición 22 hacía que Anc.11-3 perdiera su función como Anc.11.
Este estudio es bastante interesante por dos cosas. Primero, porque nos permitió observar directamente cómo evolucionan las maquinarias moleculares complejas gracias a la reconstrucción de proteínas ancestrales. Y segundo, porque el trabajo demostró que la complejidad se puede adquirir fácilmente a través de simples procesos de duplicación genética y simples mutaciones degenerativas, que a la larga ofrecen algún tipo de ventaja adaptativa al organismo.
Sin embargo, los investigadores son bastante prudentes con sus conclusiones. “Somos conscientes de que no hay otro análisis similar de la trayectoria evolutiva de una maquinaria molecular, por lo que la generalidad de nuestras observaciones es desconocida”, advierte Thornton. Simplemente, el estudio nos da una respuesta sencilla y elegante de cómo estas maravillas de la naturaleza se hacen cada vez más complejas.
Referencia:
 Finnigan, G., Hanson-Smith, V., Stevens, T., & Thornton, J. (2012). Evolution of increased complexity in a molecular machine Nature DOI: 10.1038/nature10724
Finnigan, G., Hanson-Smith, V., Stevens, T., & Thornton, J. (2012). Evolution of increased complexity in a molecular machine Nature DOI: 10.1038/nature10724
Joseph W. Thornton. (2004). Resurrecting ancient genes: experimental analysis of extinct molecules Nat Rev Gen 5, 366-375 DOI: 10.1038/nrg1324
Esta entrada participa en el IX Carnaval de Biología celebrado en el blog La ciencia de la vida de mi colega Carlos Lobato.