Un día como hoy en 1953, tres breves artículos publicados en Nature: uno por Francis Crick y James Watson, otro por Maurice Wilkins y colaboradores y otro por Rosalind Franklin; revolucionó todo el campo de la biología porque significó nada menos que el inicio de la “era de la genómica”. La estructura del ADN era finalmente revelada siendo considerado como uno de los más grandes logros científicos del siglo XX.
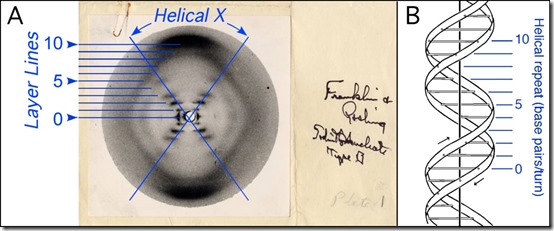 Imagen | Intech.
Imagen | Intech.
La historia de este notable descubrimiento es muy interesante y está lleno de drama y mucha controversia, donde la valiosa contribución de Rosalind Franklin inicialmente no fue reconocida como debió ser. Eran tiempos muy difíciles para las mujeres científicas.
Nominados al Nobel de Química
El 22 de enero de 1960, Watson y Crick fueron nominados por primera vez al Premio Nobel de Fisiología o Medicina de ese año por el virólogo británico Michael Stoker. A esta nominación les siguieron otras dos ese mismo año y tres más al año siguiente. Finalmente, en 1962, Watson, Crick y Wilkins recibieron el Nobel por su importante descubrimiento. Rosalind Franklin no lo recibió porque murió en 1958 y una de las reglas de este galardón es que no se entrega de manera póstuma.
Sin embargo, lo que pocos saben es que Watson, Crick y Wilkins también fueron nominados al Nobel de Química de 1962 según revela una carta firmada por Jacques Monod, Peter Campbell, William Stein, Harold Urey, John Cockroft y Stanford Moore, que fue dirigida al Comité Nobel y que fueron halladas en los archivos del Instituto Pasteur de Francia.
 Imagen | Nature.
Imagen | Nature.
El Nobel de Química de 1962 cayó finalmente en manos de Max Perutz y John Kendrew por la determinación de las estructuras de la hemoglobina y la mioglobina.
Los gloriosos años 70’s
Esta década fue de las más importantes para la biología molecular. Gracias al descubrimiento de la estructura del ADN veinte años atrás, se lograron muchos avances en el estudio de los genes a nivel individual: cómo almacenaban la información, cómo se expresaban, que proteínas codificaban, qué relación tenían con el fenotipo de un organismo, etc. Sin embargo, aún quedaban muchas interrogantes, entre ellas, la forma cómo eran encendidos o apagados, cómo estaba distribuidos en los cromosomas y a qué distancias se encontraban unos de otros, qué efectos tenían las mutaciones, cómo se daba la evolución éste nivel, etc.
A inicios de la década de 1970, los microbiólogos Hamilton Smith y Kent Wilcox aislaron y caracterizaron la primera enzima de restricción que son como unas tijeras biológicas que cortan el ADN en regiones con secuencias muy específicas. Gracias a esta enzima y el desarrollo una técnica que permitía separar por tamaño los fragmentos de ADN cortados (electroforesis), los científicos empezaron elaborar mapas de restricción (lugares del genoma donde actúa la enzima) y establecer la distancia física entre distintos genes. En otras palabras, los genes dejaron de ser algo abstracto. Ahora ya tenían una ubicación física dentro del genoma.

En 1975, Fred Sanger desarrolló una técnica que le permitía leer los genes letra por letra (nucleótido por nucleótido). Dos años después presentó por primera vez la secuencia completa de un pequeño genoma: el del fago φX174. Así nació la secuenciación del ADN. Esta técnica original era sumamente laboriosa, requería el uso de sondas radiactivas y muchas horas de trabajo para leer pequeñas secuencias de ADN. Sin embargo, el método de Sanger sentó las bases para los primeros equipos de secuenciación automáticos que ya permitían leer miles de pares de base en algunas horas a inicios de la década de 1990. Actualmente, los secuenciadores de última generación permiten leer millones de secuencias —incluso genomas completos— en pocas horas y por una ridícula fracción del costo que tenía hace 10 años.
En 1977, dos equipos de investigadores, uno liderado por Rich Roberts y el otro por Phil Sharp hicieron un extraño descubrimiento: las moléculas de ARN mensajero (ARNm) de un adenovirus eran más pequeñas que sus genes correspondientes. Esto indicaba que los genes tenían regiones que eran removidas durante la formación del ARNm. Al año siguiente, el Walter Gilbert escribió un artículo de opinión sobre este descubrimiento y propuso llamar a esas regiones “intrones” (por intragenic regions).
A pesar que no se conocían muchos genes en 1978, el Dr. Gilbert predijo que existía al menos 10 veces más intrones que exones (las regiones que llegan a expresarse en forma de proteínas) y que algunas porciones de los intrones podrían llegar a actuar como exones, lo que ahora se conoce como splicing alternativo. Este mecanismo permite ampliar la diversidad de proteínas y regular la actividad de un gen para que cumpla diferentes funciones según el tejido donde se exprese.
No codifica pero regula
En el 2003 se presentó el genoma humano. Había grandes expectativas en torno a él porque se creía que por fin encontraríamos las causas que provocan el cáncer, el envejecimiento celular, los desórdenes metabólicos, entre otras cosa. Sin embargo, ya pasaron más de 10 años y aún no tenemos respuestas.
Prácticamente, todo el genoma humano (~98%), y en general, de todos los organismos superiores no son codificantes, en otras palabras, nunca llegan a formar proteínas, ni siquiera péptidos. Por algunos años lo consideraron como “ADN basura”, un mal nombre porque el hecho de no saber para que sirve no significa que no sirve para nada. Entonces, ¿qué hace ahí? Para resolver esta interrogante, en el 2003 se formó un consorcio internacional de investigadores que se pusieron a trabajar en un proyecto llamado ENCODE cuya finalidad era encontrar todos los elementos funcionales en el genoma humano.
Los resultados que aparecieron publicados en 30 artículos científicos a fines de 2012 fueron muy interesantes. En resumen se determinó que el 20% del ADN no codificante es funcional y que un 60% adicional llega a ser transcrito hasta ARN pero se desconoce cuál es su función real. La mayor parte de ese 20% se encarga de regular la expresión de genes codificantes, incluso actuando a largas distancias y de manera coordinada con otros factores. Incluso estas regiones no codificantes presentan mutaciones que podrían estar asociadas con el desarrollo de ciertas enfermedades.
Como podemos ver, el descubrimiento de la estructura del ADN hace 60 años nos ha abierto una verdadera caja de pandora. Se han generado muchas más preguntas que respuestas, similar a lo que fue el descubrimiento de la expansión acelerada del universo para la cosmología. El camino iniciado en el siglo XIX por Mendel, Darwin y Wallace no parece tener aún un destino a la vista, el viaje continua y eso es bueno. Es lo bello que tiene la ciencia.