Hace unos seis meses hablamos en el blog del controvertido descubrimiento hecho por investigadores del Instituto de Astrobiología de la NASA, liderados por la Dra. Felisa Wolfe-Simon, en el cual sugerían que unas bacterias aisladas del Lago Mono en California tenían la capacidad de reemplazar al fósforo —uno de los seis elementos químicos fundamentales de los organismos vivos— por el arsénico, en muchas biomoléculas, incluso en el ADN.
![[bacterias_arsenato[4].jpg]](//lh4.ggpht.com/_4607aWHTvKI/TPhy20XkS9I/AAAAAAAADAA/WQLbtTMYrxs/s1600/bacterias_arsenato%5B4%5D.jpg)
Las críticas empezaron a invadir la web a tan sólo a horas de haber sido anunciado el descubrimiento en una conferencia de prensa donde la gente esperaba que la NASA anuncie algo que en realidad cambiaría la forma como estudiamos la astrobiología, tal vez, el descubrimiento de algún marcianito por ahí. Pero, lo que Wolfe-Simon et al. publicaron en Science, fue algo más controvertido para el mundo biológico, ya que cambiaba algo que se daba por sentado para que la vida pueda ser como hoy la conocemos. Por esta razón, muchos investigadores se mostraron su escepticismo abiertamente y empezaron a criticar el artículo. Aquí haremos un recuento de las principales críticas.
Niveles de fósforo requeridos por la bacteria para vivir
Wolfe-Simon et al. demostraron que sus bacterias (GFAJ-1) usaban el As en vez del P cuando vieron que el crecimiento de las bacterias fue significativamente superior en un medio de cultivo con arsénico y sin fósforo (As+/P-) que en un medio sin ninguno de los dos componentes (As-/P-).
Según James B. Cotner de la Universidad de Minnesota y Edward K. Hall de la Universidad de Viena, Wolfe-Simon y sus colaboradores asumieron que el contenido de P encontrado en las bacterias que crecieron en el medio de cultivo As+/P- (0.02% ± 0.01%) está por debajo del 1% – 3% de P requerido típicamente por una bacteria heterotrófica como lo es GFAJ-1. Pero, estos valores no serían buenos referentes ya que representan los niveles de P obtenidos en bacterias intestinales (E. coli) que fueron cultivados en medios ricos en P (de 0.013 a 1.3 mM), las cuales, además, tienen tasas de crecimiento entre 80 a 280 veces más rápidas que las GFAJ-1, por lo tanto requerirán de mayor cantidad de P.
Por otro lado, Cortner & Hall también cuantificaron los niveles de P requeridos por bacterias aisladas de ecosistemas acuáticos, encontrando que la mayoría tenía un 0.5% de P, y hasta se pudo encontrar algunas bacterias que tenían tan sólo 0.03% de P. Concluyendo que si bien los valores obtenidos por Wolfe-Simon son bajos, caen dentro del rango encontrado en bacterias de ecosistemas acuáticos, sobre todo en aquellas que viven en ambientes donde los niveles de P son limitantes.
La microbióloga Rosie Redfield de la Universidad de la Columbia Británica, la principal detractora tanto científica como personal de Wolfe-Simon, cree que los niveles de P encontrados en los medios de cultivo As+/P- debido a las impurezas de los reactivos usados para prepararlos (~3.1uM), pueden ser más que suficientes para promover el crecimiento de las GFAJ-1 y no necesariamente es el As el responsable de dicho crecimiento.
Schoepp-Cothenet et al. también hacen la misma observación, y es que a pesar que hay 10,000 veces más As que P en el medio de cultivo, la bacteria sólo tiene en su interior 3 veces más As que P. Así que ellos creen que la bacteria usa las trazas de P del medio para poder crecer, esto sumado a que su división celular es lenta (cada 2 días), permitiéndole tener un tiempo más que suficiente para poder capturar las pequeñas cantidades de P y usarlo para su crecimiento.
Los investigadores húngaros István Csabai y Eörs Szathmáry también critican que Wolfe-Simon et al. no hayan calculado los niveles mínimos de P requeridos por GFAJ-1 para poder vivir, tal vez el 0.019% encontrado en las que crecieron en el medio As+/P- era suficiente para ello.
Además, la Dra, Patricia L. Foster de la Universidad de Indiana explica que E. coli y muchas otras bacterias tienen dos formas principales de asimilar el P: a través del sistema Pst (transportador específico de fosfato), el cual asimila el P cuando los niveles de fosfato son bajos; y a través del sistema Pit (transportador de fosfato inorgánico), el cual tiene una baja afinidad por el P, pero está activo todo el tiempo. Cuando se cultiva a E. coli en As, se incrementa la asimilación de P vía Pst, aunque el sistema Pit empieza a meter As al interior de la célula, envenenándola. Tal vez lo que Wolfe-Simon et al. hicieron al someter a sus bacterias al medio As+/P- fue seleccionar mutantes que perdieron el sistema Pit e incrementaron la capacidad del sistema Pst, permitiéndole asimilar más P, aprovechando las trazas que habían en el medio As+/P-.
Problemas con el ADN y su posible composición en base al As
Algo que a mi también me sorprendió cuando leí el artículo fue que Wolfe-Simon et al. no usaron kits de extracción y purificación de ADN comerciales, sino que usaron métodos fisicoquímicos tradicionales que son usados principalmente en laboratorios con bajos presupuestos (extracción por fenol:cloroformo). El problema con este método, según comenta Redfield, es que al precipitar el ADN con etanol —en la última fase del proceso— el precipitado (pelet) queda contaminado con impurezas de ARN y otras moléculas insolubles en etanol.
Sin embargo, para que las impurezas que podrían estar presentes en las muestras de ADN no afecten los estudios posteriores, Wolfe-Simon et al. limpiaron el ADN usando la electroforesis. Cargaron las muestras de ADN en un gel de agarosa y le aplicaron una corriente eléctrica. El ARN por ser más pequeño, migrará mucho más rápido que el ADN. Las proteínas, por ser más grandes, y otras moléculas insolubles no migrarán por el gel y se quedarán en el mismo donde fueron sembrados, de esta manera, el ADN se separará de todos sus contaminantes.
 Pero, otro problema fue que Wolfe-Simon no purificó el ADN de los geles de agarosa —un procedimiento que no tarda ni 10 minutos con un kit comercial. Lo que hicieron fue secar el gel y analizar el ADN con las técnicas espectrométricas. Por esta razón no se sabe exactamente cuanto ADN puede haber en el pedazo de agarosa analizado y, tal vez, la cantidad de carbono obtenido con el cual hicieron algunos de los cálculos, correspondía en realidad a la agarosa y no al ADN.
Pero, otro problema fue que Wolfe-Simon no purificó el ADN de los geles de agarosa —un procedimiento que no tarda ni 10 minutos con un kit comercial. Lo que hicieron fue secar el gel y analizar el ADN con las técnicas espectrométricas. Por esta razón no se sabe exactamente cuanto ADN puede haber en el pedazo de agarosa analizado y, tal vez, la cantidad de carbono obtenido con el cual hicieron algunos de los cálculos, correspondía en realidad a la agarosa y no al ADN.
Por otro lado, Stefan Oehler del Centro de Investigación de Biociencias Médicas Alexander Flemming de Grecia, cree que si Wolfe-Simon et al. hubieran hecho una centrifugación de gradiente de densidad, técnica que permitiría diferenciar al ADN con As del ADN con P,en base a su densidad (el ADN con As sería más pesado, por lo tanto, más denso que el ADN con P), tal vez le hubiera dado mayor contundencia a sus resultados.
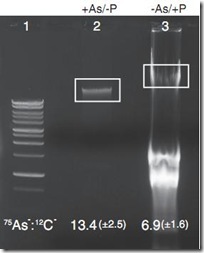
David W. Borhani, estudiante del MIT, hace una interesante observación del gel donde se corrió el ADN. Como pueden ver en la figura, el ADN extraído de la bacteria que creció en el medio As+/P- tiene un tamaño diferente y esta más limpio que el ADN de la bacteria que creció en el medio con As-/P+, el cual está degradado. Si el As es más inestable que el P, sería más probable observar al ADN de la bacteria del arsénico degradado. Además, las proporciones de As:C en las bacterias que crecieron en As+/P- es sólo el doble —y la 1/64 parte de la proporción de P:C— de las que crecieron en el medio As-/P+. Si el ADN de estas bacterias estuviera compuesta de As, las proporciones deberían ser más cercanas.
Inestabilidad del As
La forma biológicamente activa del As es el arsenato, tal como la forma activa del P es el fosfato. En forma de arsenato (As+5), el As puede fácilmente reemplazar al P en muchos aspectos bioquímicos y es ahí donde radica su toxicidad. Sin embargo, las condiciones fisiológicas del citoplasma de las bacterias (potenciales redox por debajo de 0mV/SHE) hacen que el As se reduzca y pase a su forma química de arsenito (As+3), la cual es biológicamente diferente al fosfato y no pasaría a integrar las principales biomoléculas del organismo. En cambio, el fosfato también puede pasar a fosfito, pero solo a partir de potenciales redox de –700mV/SHE, el cual está fuera del rango de las condiciones fisiológicas de las bacterias. Es por estas razones que Schoepp-Cothenet et al. creen que lo que Wolfe-Simon encontró fueron bacterias que pueden vivir con altas concentraciones de As en su interior (en forma de arsenito) y tal vez sean estas impurezas las que se apreciaron en los datos espectrométricos.
Steven A. Benner del Instituto Westheimer para la Ciencia y Tecnología calcula que el tiempo de vida media promedio de los enlaces arsenato-ADN al pH del interior de las bacterias es de aproximadamente un minuto, a diferencia de los ~30 millones de años de los enlaces fosfato-ADN. Para poder tener un ADN hecho a base de As se deben superar muchas barreras bioquímicas, principalmente, la cinética de la reacción de hidrólisis de los enlaces éster del arsenato con los nucleótidos, para ello deberían haber compuestos que estabilicen estos enlaces ya que el proceso de extracción de ADN y electroforesis es largo y estresante, sobre todo, los tratamientos fisicoquímicos (centrifugaciones, extracciones con solventes orgánicos, potenciales eléctricos, etc.).
Además, debemos recordar que el As no entra así por así, de manera directa, a reemplazar al P. Para que el As esté presente en el ADN, este debe ser introducido desde el momento en que se forman los nucleótidos (la unión de los grupos arsenato a las pentosas unidas a las bases nitrogenadas), pero, como el arsenato es sumamente inestable, sería casi imposible que llegue a soportar todo el proceso de síntesis. Para que pueda funcionar la hipótesis, el arsenato debería ser introducido a los nucleótidos a través de un mecanismo alternativo directo o con la ayuda de moléculas estabilizadoras.
Errores de cálculos y estadísticos
Csabai & Szathmáry también hacen un interesante comentario acerca de las posibles fallas al momento de hacer los cálculos. Wolfe-Simon dicen haber encontrado una tasa de As:P de 7.3 en el medio As+/P- y 0.002 en As-/P+, pero estos valores fueron obtenidos a partir de tasas promedio. Cuando Csabai & Szathmáry hicieron los cálculos en sentido inverso, o sea, calcularon la tasa de As:P a partir de la inversa de la tasa P:As [ya que matemáticamente As:P = 1/(P:As) = (P:As)-1], la tasa de As:P ya no fue de 7.3, sino de 1.59.
Por otro lado, en los resultados publicados por Wolfe-Simon, muchas de las desviaciones estándar eran mayores al mismo valor promedio obtenido, por ejemplo, en la proporción de As en el peso seco de las bacterias, la cual era equivalente a 0.19%±0.25%. Esto quiere decir, que en algunas muestras no había nada de As. Y en cuanto a los análisis espectrométricos, las líneas bases de P son muy altas con respecto a la cantidad de P cuantificado en las muestras de ADN, lo que podría generar datos inexactos, ya sea subestimados o sobrestimados.
Conclusión
Como pueden, son muchas las observaciones que se le han hecho al trabajo, muchas de ellas bastante sólidas que deben ser resueltas o, por lo menos, explicadas por los autores para darle validez a la investigación. Entre las principales críticas tenemos los niveles de P presentes en las impurezas de los reactivos usados en la preparación de los medios de cultivo. Estos niveles, que de por sí son muy bajos, podrían ser más que suficientes para promover el crecimiento de las bacterias. Para ello faltaría determinar los niveles mínimos de P requeridos por GFAJ-1 para poder vivir y la forma como asimila tanto el As como el P.
Por otro lado, la inestabilidad del arsenato dadas las condiciones fisiológicas del citoplasma de las bacterias, hacen bastante improbable la presencia del As en diferentes biomoléculas, más aún el ADN. Esta barrera bioquímica es una de las más difíciles de superar.
Wolfe-Simon ha respondido a todas las críticas en su respuesta técnica también publicada ayer en Science. Los autores se mantienen firmes con sus datos y conclusiones, ya que todas las críticas han podido ser respondidas, alguna de ellas un poco forzadas. Sin dudas, hay resultados que deben ser corroborados en otros laboratorios por otros investigadores, para ello, se pueden solicitar muestras de la cepa GFAJ-1 (GFAJ1samplerequest@gmail.com), para que cada uno haga sus propios experimentos.
Referencias:
 Cotner, J., & Hall, E. (2011). Comment on "A Bacterium That Can Grow by Using Arsenic Instead of Phosphorus" Science DOI: 10.1126/science.1201943
Cotner, J., & Hall, E. (2011). Comment on "A Bacterium That Can Grow by Using Arsenic Instead of Phosphorus" Science DOI: 10.1126/science.1201943
Redfield, R. (2011). Comment on "A Bacterium That Can Grow by Using Arsenic Instead of Phosphorus" Science DOI: 10.1126/science.1201482
Schoepp-Cothenet, B., Nitschke, W., Barge, L., Ponce, A., Russell, M., & Tsapin, A. (2011). Comment on "A Bacterium That Can Grow by Using Arsenic Instead of Phosphorus" Science DOI: 10.1126/science.1201438
Csabai, I., & Szathmary, E. (2011). Comment on "A Bacterium That Can Grow by Using Arsenic Instead of Phosphorus" Science DOI: 10.1126/science.1201399
Oehler, S. (2011). Comment on "A Bacterium That Can Grow by Using Arsenic Instead of Phosphorus" Science DOI: 10.1126/science.1201381
Alberts, B. (2011). Editor's Note Science DOI: 10.1126/science.1208877
Borhani, D. (2011). Comment on "A Bacterium That Can Grow by Using Arsenic Instead of Phosphorus" Science DOI: 10.1126/science.1201255
Benner, S. (2011). Comment on "A Bacterium That Can Grow by Using Arsenic Instead of Phosphorus" Science DOI: 10.1126/science.1201304
Foster, P. (2011). Comment on "A Bacterium That Can Grow by Using Arsenic Instead of Phosphorus" Science DOI: 10.1126/science.1201551
Wolfe-Simon, F., Blum, J., Kulp, T., Gordon, G., Hoeft, S., Pett-Ridge, J., Stolz, J., Webb, S., Weber, P., Davies, P., Anbar, A., & Oremland, R. (2011). Response to Comments on "A Bacterium That Can Grow Using Arsenic Instead of Phosphorus" Science DOI: 10.1126/science.1202098
Sirot, L., Wolfner, M., & Wigby, S. (2011). Protein-specific manipulation of ejaculate composition in response to female mating status in Drosophila melanogaster Proceedings of the National Academy of Sciences DOI: 10.1073/pnas.1100905108











![[bacterias_arsenato[4].jpg]](http://lh4.ggpht.com/_4607aWHTvKI/TPhy20XkS9I/AAAAAAAADAA/WQLbtTMYrxs/s1600/bacterias_arsenato%5B4%5D.jpg)