Los hongos (Reino: Fungi) forman parte importante de cualquier ecosistema del planeta, ya que son los principales responsables de la degradación de toda la biomasa muerta que hay en él y, además, pueden establecer relaciones simbióticas con muchas especies de plantas y animales. Sin embargo, se sabe muy poco acerca de su historia evolutiva. Científicos británicos descubrieron que existe un grupo muy diverso de hongos primitivos, muy relacionados con el género Rozella, a los cuales agruparon en un nuevo clado llamado Criptomycota (Cryptomycota), según reportaron hoy en Nature.
Los hongos se presentan de dos formas típicas: levadura y filamentos, caracterizándose por tener una pared celular rígida hecha a base de quitina, la cual les permitió adaptarse a los diferentes hábitats que hay en el planeta. Sin embargo, nuestro entendimiento sobre la historia evolutiva de los hongos sólo se limita a aquellos que hemos podido aislar y cultivar en el laboratorio. Existe una gran cantidad de especies de hongos que no han podido ser cultivados y mucho menos descritos. Por esta razón, se deben idear estrategias que permitan estudiarlos in situ.
Fue así que la Dra. Meredith Jones de la Universidad de Exeter y sus colaboradores colectaron muestras de agua y sedimentos de un pequeño estanque local, para extraer el ADN total presente en él (ADN ambiental). Luego, analizaron estas secuencias de ADN y buscaron las regiones correspondientes a la subunidad pequeña del ADN ribosomal (SSU ADNr) de los hongos para compararlas con las mismas secuencias pero de hongos ya caracterizados previamente —disponibles en el GenBank—, para así construir un árbol filogenético (un diagrama que permite observar las relaciones evolutivas entre diferentes especies).
Al observar el árbol se llevaron una gran sorpresa, las secuencias correspondientes al ADN ambiental formaban un grupo diferente de hongos. Para profundizar el estudio, Jones et al. hicieron el mismo análisis con datos de ADN ambiental de otros hábitats y regiones geográficas del mundo —también disponibles en el GenBank— y observaron muchas secuencias caían dentro de este nuevo grupo, tanto así que llegaba a ser tan diverso como los otros siete grupos hongos juntos. Por otro lado, el árbol filogenético mostró que esta nueva rama apareció muy temprano en la historia evolutiva de los hongos, junto con los miembros del género Rozella, quienes eran considerados como los hongos más primitivos hasta ahora. A este nuevo grupo lo llamaron los Criptomycota (sombreado en rojo).
[Click para agrandar la imagen]
Sin dudas fue un descubrimiento sorprendente… ¿cómo fue que un grupo tan diverso de hongos había sido ignorado por completo en el árbol de la vida este reino? El grupo es tan grande que hasta podría haber formado su propio filo.
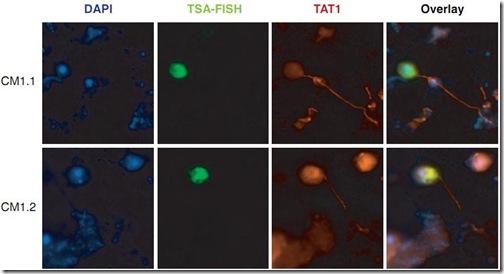
La excitación fue tan grande que ahora los investigadores querían saber todo sobre este nuevo clado. Para ello usaron sondas fluorescentes conjugadas con anticuerpos monoclonales y secuencias de ADN, que reconocen moléculas específicas de la célula o regiones específicas del material genético del hongo, respectivamente. A esta técnica se la conoce como FISH (Hibridación Fluorescente in situ).
Usando la Amplificación de la Señal con Tiramida (Primera figura: TSA-FISH), se pudo determinar la forma (ovoides) y tamaño (3 – 5μm) de los Criptomycotas. Luego, usando el anticuerpo monoclonal TAT1, el cual se une específicamente a la α-tublina —principal componente estructural de los flagelos— los investigadores evidenciaron la presencia de zoosporas (esporas flageladas móviles sumamente primitivas). También habían otras células que no presentaban flagelo (15 – 53%), lo que indicaría que se encuentran en una forma de quiste, tal como en los quitridios. Los Criptomycotas fueron identificados usando una sonda de ADN —que reconoce y se hibridiza una secuencia específica del genoma de esos hongos— marcada con DAPI.
Por otro lado, los investigadores observaron evidencias de una tercera fase en su ciclo de vida. En esta fase, los Criptomycotas no flagelados se adhieren a un grupo diferente de células, sobre todo con diatomeas, posiblemente en una asociación saprófita o parasitaria. Sin embargo, este comportamiento no fue muy común y tal vez se deba a la interacción directa que hay con los microorganismos que viven en el mismo ambiente con ellos.
Por ahora, la única forma que tenemos de estudiarlos es de esta manera, in situ, ya que si pudieran ser cultivados en el laboratorio, se podrían controlar muchos parámetros, para saber que tipo de asociación tienen con estas células. Estas técnicas in situ tampoco permitieron detectar estadíos celulares claves como los tubos germinativos de las esporas, rizoides, hifas, esporangios y características de la división celular (mitosis).
La pared celular en los hongos ha sido una de las adquisiciones más importantes en su evolución, ya que les ha permitido diversificarse en ambientes con altas presiones osmóticas y alimentarse directamente mediante la osmotrofía. El género Rozella, que se encuentra ahora dentro del clado Criptomycota, no tiene la capacidad de producir pared celular, pero si es capaz de tomarla “prestada” de las células a las cuales infecta.
Para determinar si el resto de los Criptomycotas ahora descubiertos producen o no pared celular, los investigadores usaron una combinación de TSA-FISH y dos tintes: el blanco de calcoflúor, para evidenciar la presencia de una pared celular rica en quitina y celulosa; y la Lectina (una aglutinina del germen de trigo), la cual evidencia la presencia de quitina. Los resultados fueron negativos, ninguno de los Criptomycotas genera pared celular micótica.
Con esto se podría concluir que una de las características claves del reino Fungi —una estrategia de crecimiento y desarrollo en base a pared celular rígida compuesta de quitina— no se encuentra presente en el ciclo de vida de los Criptomycotas. Sin embargo, aún faltan muchas cosas por investigar en este nuevo grupo de hongos, de ahí el origen de su nombre (krypto: oculto, misterioso”). Además, cabe recordar que el nombre Criptomycota aún es tentativo.
Referencia:
Jones, M., Forn, I., Gadelha, C., Egan, M., Bass, D., Massana, R., & Richards, T. (2011). Discovery of novel intermediate forms redefines the fungal tree of life Nature DOI: 10.1038/nature09984
Nota: Por mantenimiento de Blogger, esta entrada será publicada a las 00:45 (Hora local) del 12/05/11.







0 comentarios:
Publicar un comentario
Se respetuoso con tus comentarios y críticas. Cualquier comentario ofensivo será eliminado.