Bueno, este artículo que fue publicado hoy en Science ha vuelto a remecer a la comunidad científica, tal como lo hizo el artículo de las bacterias del arsénico, también publicado en Science a fines del año pasado. Y no es para más, de corroborarse los resultados, cambiaría la forma en la que estudiamos los genes, su relación con las enfermedades, el desarrollo del cáncer, en fin, una gran parte de la biología molecular.

Según el Dogma Central de la Biología Molecular, el ARNm refleja —de manera precisa— la información contenida en los genes, ya que la secuencia de aminoácidos que conforman cada proteína que será traducida a partir del ARNm, están determinadas por las secuencias de ADN (genes), en base al código genético.
Sin embargo, existen algunas excepciones donde el ARNm no refleja lo que dice la secuencia de ADN que lo originó. Entre estas excepciones encontramos: los producidos por errores al momento de transcribir el ADN a ARNm; los producidos por la edición de las moléculas de ARNm a través del splicing, donde se eliminan regiones del ARNm que no deben llegar a traducirse a proteínas (intrones), y los producidos por las enzimas que modifican algunos nucleótidos del ARNm a través de desaminaciones (Ej.: La enzima ADAR que desamina la Adenosina y la convierte en Inosina, la cual es reconocida por el ribosoma como una guanosina, o sea, hay un cambio de base de A por G).
Además, debido a que la ARN polimerasa —la enzima que se encarga de transcribir ADN en ARNm— tiene la capacidad de detectar errores en la transcripción y poder repararlos (también conocido como ‘proofreading’), este tipo de excepciones son poco frecuentes. Por otro lado, el splicing no cambia la secuencia de nucleótidos del ARNm, sino, elimina determinadas porciones para que no sean traducidas a proteína, y las que permanecen en el ARNm (exones), mantienen la misma secuencia de la cual fueron originados.
Por otro lado, las enzimas como ADAR y APOBEC —la cual cambia la C por U— no son muy comunes y sólo se limitan a editar las secuencias de ARNm de determinados genes y en determinados tejidos, como aquel que codifica para la apolipoproteína B.
En el trabajo publicado hoy en Science, un grupo de investigadores liderados por la Dra. Vivian Cheung de la Escuela de Medicina de la Universidad de Pennsylvania, reportaron haber encontrado más de 10,000 regiones en las cuales las bases del ARNm no correspondían a las bases del ADN que la codificaron, y muchos de estos ARNm llegaban a codificar proteínas, las cuales portaban estos cambios en sus secuencias de aminoácidos, incluso, algunos eran más grandes o más pequeños que los originales, debido a que las secuencias de término (codones STOP) eran modificados a causa de los cambios en los nucleótidos del ARNm.
Lo que hicieron Cheung y sus colaboradores fue obtener las secuencias de ADN y ARNm de un tipo de glóbulos blancos (los linfocitos B) de 27 personas diferentes, las cuales forman parte de los proyectos “1000 Genomes” e “International HapMap”. Luego, compararon cada una de las secuencias de ARNm con sus respectivas secuencias de ADN de las cuales se originaron, encontrando más de 28,000 diferencias en al menos 10,000 regiones exónicas diferentes (~4,700 genes conocidos), de los cuales, el 71% de estos cambios no eran sinónimos (al traducirse provocaba el cambio de un aminoácido por otro, muchas veces con propiedades químicas diferentes)
Para dar una mayor robustez a los datos y evitar que estos se deban a errores en el secuenciamiento, sólo fueron consideradas aquellas diferencias que se presentaban en al menos dos individuos diferentes y en más del 10% de los ‘reads’ de cada nucleótido [Ver cobertura de secuenciamiento]. Además, los investigadores mandaron a secuenciar, en distintos laboratorios, regiones del ADN que eran monomórficas (no varían entre un individuo y otro), para corroborar que todos obtuvieran las mismas secuencias, sin errores.
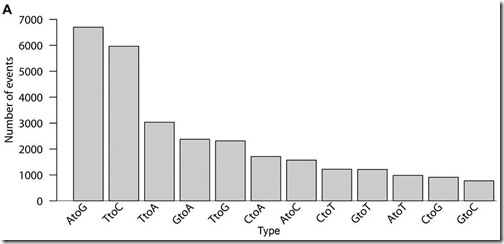
Bueno, como mencioné algunos párrafos atrás, las diferencias entre las secuencias de ARNm y el ADN del cual se originaron pueden deberse a distintas causas; sin embargo, estas no pueden explicar muchos de los cambios encontrados por Cheung et al. Por ejemplo, si descartamos los cambios que pueden ser explicados por la acción de las enzimas ADAR y APOBEC (A->G y C->T), aún así hay una gran cantidad de cambios (~43%) que no pueden ser explicados por algún otro mecanismo, principalmente las transversiones —cambio de una purina (A o G) por una pirimidina (C o T) o viceversa.
Otro dato importante que respalda los descubrimientos de Cheung y sus colaboradores es que estos cambios no sólo se encontraron en los linfocitos B, también se podían encontrar en las células de otros tejidos como de la epidermis, del cerebro, de los testículos, embrionarias y hasta cancerosas. Por otro lado, los investigadores también encontraron dichos cambios en secuencias de otras personas —fuera de los 27 individuos que formaron parte del estudio— a través del uso de secuencias EST disponibles en las bases de datos genéticas.
Pero, ¿estos ARNm cambiados llegarían a codificar proteínas?. Para responder a esta pregunta, los investigadores hicieron un análisis proteómico. Para ello, Mingyao Li —autor principal de la investigación— usó la espectrometría de masas acoplada a una cromatografía líquida para separar e identificar cada péptido y proteína presente en los linfocitos B. Los resultados mostraron que habían péptidos cuya secuencia de aminoácidos no correspondía al gen del cual fueron codificados, sino al ARNm modificado que fue traducido. En algunos casos, la proteína resultante era mucho más grande que la original, tal vez por que el cambio en el ARNm suprimió el codón de terminación y la traducción continuó hasta encontrar otro codón de terminación, generando una proteína con 55 aminoácidos de más (la RPL28).
Las Diferencias de ARN-ADN (RDD: RNA-DNA Differences) no estaban distribuidas uniformemente a lo largo del genoma. Por ejemplo, el cromosoma 19 era el que tenía más RDD, mientras que el 13 era el que tenía menos. Los investigadores también encontraron un patrón: los RDD eran bastante comunes en los genes que juegan roles importantes en la función helicasa y de unión a proteínas y nucleótidos. También se encontraron diferencias en su distribución dentro de los genes: la mayoría se encontraban en los exones codificantes (44%) y en las regiones no traducidas del extremo 3’ (39%).
Pero, no todos los ARNm eran diferentes a las secuencias de ADN que los originaban. En un determinado RDD, en promedio, el 20% de los ARNm eran diferentes, mientras que los demás eran transcritos de manera precisa. Sin embargo, habían algunas secuencias donde casi todos los ARNm eran diferentes al ADN correspondiente, por ejemplo, en el gen que codifica la enzima DCP1A. En otros genes, como en el que codifica para la proteína Ras RHOT1, había diferencias significativas a nivel de individuos: en una persona el nivel de ARNm modificado era del 18% mientras que en otra era del 90%.
Estas diferencias en los niveles de expresión de los RDD nos pueden dar pistas para encontrar las causas de ciertas patologías que no han podido ser explicadas a través de los estudios a nivel genético; ya que, al final, el responsable de la expresión de una proteína buena o defectuosa no es el ADN, sino el ARNm. Entonces, tal vez sea más importante hacer estudios transcriptómicos que estudios genómicos al momento de buscar diferencias entre una persona sana y una enferma.
Sin embargo, Li y sus colaboradores no han podido encontrar la respuesta sobre el origen de estos cambios en el ARNm con respecto a su ADN molde. Tal vez hay aspectos, tanto a nivel transcripcional como pos-trancripcional que por ahora desconocemos. Por ello, los investigadores se dedicarán a buscar este mismo fenómeno en otras especies, para ver si este mecanismo de edición del ARNm es universal, o sólo se da en ciertos grupos de organismos.
De lo que si estoy seguro es que esta investigación dará que hablar durante los próximos días. Sólo han pasado unas horas desde que fue publicado este artículo en Science Express (la versión online de Science, donde se publican los artículos antes de ser aceptados en la revista) y ya han aparecido una gran cantidad de comentarios al respecto, muchos de ellos mostrando un gran escepticismo y, tal vez tengan razón.
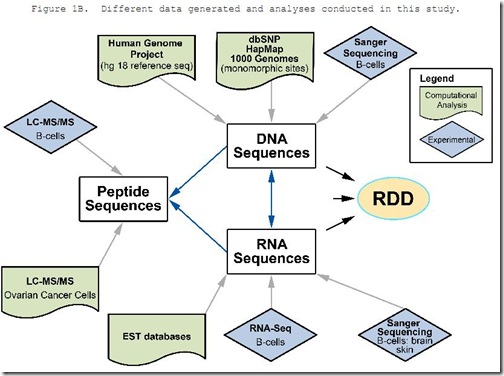
Sin embargo, las pruebas que presentan los autores en este estudio están bien fundamentadas, tomando todas las precauciones del caso, ya sea comparando la calidad y reproducibilidad de las secuencias generadas en diferentes laboratorios, tomando en cuenta solo aquellas que tienen un buen número de reads por nucleótido (buena cobertura de secuenciamiento), corroborando los resultados a nivel proteico mediante espectrometría de masas, encontrando los mismos cambios en diferentes tejidos e individuos, etc. (Ver figura):
Esto no quiere decir que podamos descartar errores en el secuenciamiento, pero, esto no alcanzaría para explicar todas las diferencias en las secuencias obtenidas a nivel de ARNm comparado con sus secuencias genéticas respectivas. Así que a esperar las críticas del presente estudio.
Referencia:
Li, M., Wang, I., Li, Y., Bruzel, A., Richards, A., Toung, J., & Cheung, V. (2011). Widespread RNA and DNA Sequence Differences in the Human Transcriptome Science DOI: 10.1126/science.1207018






0 comentarios:
Publicar un comentario
Se respetuoso con tus comentarios y críticas. Cualquier comentario ofensivo será eliminado.