El viernes pasado, un grupo de investigadores del famosísimo Instituto J. Craig Venter liderados por el Dr. Dongying Wu de la UC Davis, publicaron un interesante artículo en PLoS ONE, el cual analiza una serie de datos metagenómicos, los cuales fueron colectados entre los años 2004 – 2007, a través de la Expedición Global de Muestro de los Océanos (GOS), encontrando secuencias genéticas que no corresponderían a ninguno de los tres dominios de la vida hasta ahora conocidos.

Actualmente, todos conocemos tres grandes dominios de la vida: los Eucariontes, el cual incluye a todos los animales, plantas, hongos y protozoarios, que se caracterizan por tener células con una membrana nuclear; las Bacterias, que incluyen a la mayoría de los microorganismos que conocemos, caracterizándose por no tener un núcleo definido y un genoma circular; y las Arqueas, que se caracterizan por vivir en ambientes extremos.
Al ver bajo un microscopio a las Arqueas y a las Bacterias, se notará que son bastante similares. Es por esta razón que las Arqueas no fueron consideradas como un dominio diferente a las Bacterias hasta los años 1970’s, gracias a los estudios realizados por Woese & Fox en 1977, que usando secuencias de ARN ribosomal pudieron construir el árbol de la vida que hoy conocemos.
Si se habrán dado cuenta, gracias a las novedosas técnicas basadas en el estudio y comparación de secuencias de ácidos nucleicos desarrolladas en los años 1970’s, los científicos pudieron identificar un nuevo dominio de la vida, el cual se mantuvo escondido —ante nuestros propios ojos— por cientos de años, desde que Leeuwenhoek observó por primera vez aquel mundo en miniatura en el siglo XVII.
Tal vez esté ocurriendo lo mismo en nuestros días… Lo dejaremos par más adelante.
Al comienzo, el estudio de las secuencias de ADN era un trabajo sumamente difícil, ya que se debía obtener grandes cantidades de material genético para poder ser secuenciados con la tecnología de la época. En los años 1980’s, el bioquímico estadounidense Kary Mullis inventó una novedosa técnica para multiplicar secuencias específicas de ADN llamada PCR. La técnica consistía en usar una enzima que replica el ADN (ADN polimerasa) y unos iniciadores (primers) que darán el punto de inicio de la replicación. De esta manera, se obtendrán millones de secuencias específicas a partir de una sola (Amplificación del ADN). Gracias a este invento se pudieron catalogar y ubicar muchas más especies en el árbol de la vida, todo en base a las secuencias del ARN ribosomal.
Pero, había un gran inconveniente que no había sido contemplado anteriormente. Para poder amplificar una secuencia específica, digamos el ARN ribosomal usado en los estudios filogenéticos y evolutivos, se debía extraer el ADN del organismo en cuestión, y para ello, el organismo debía ser aislado y cultivado en el laboratorio, ya sea en una placa petri o un tubo de ensayo. Sin embargo, en la naturaleza existen millones de microorganismos que no pueden ser cultivados en un laboratorio. Entonces, ¿cómo se podría caracterizarlos y ubicarlos en el árbol de la vida, si no tenemos forma de extraerle su ADN?
Gracias al avance en las técnicas de secuenciamiento, los cuales ahora son tan poderosas que permiten analizar millones de secuencias a la vez, los investigadores se han dedicado a secuenciar todo el ADN que puede ser encontrado en un determinado ambiente; por ejemplo, en el lecho marino, en nuestro tracto intestinal o en un relave minero; y compararlo con secuencias previamente identificadas y estudiadas para estimar los posibles organismos que habitan una determinada zona, sin la necesidad de aislarlos y cultivarlos. A esto se le llama la metagenómica.
En el año 2004, el famoso científico Craig Venter, quien secuenció el genoma humano en menos de tres años (comparado con los más de diez que le tomó al consorcio internacional que se hacía cargo de ello), se enrumbó en una expedición por todos los océanos de mundo en el Sorcerer II. La expedición consistía en tomar muestras de mar, de diferentes regiones del mundo, para analizar la diversidad bioquímica y genética microbiana que poseían, sin la necesidad de aislar individualmente cada microorganismo que habita en ella.
Los resultados fueron asombrosos, Venter y sus colaboradores encontraron millones de nuevas secuencias, las cuales podrían codificar para una variedad similar de nuevas proteínas, muchas de ellas con importantes aplicaciones industriales. Pero, a partir de toda esta data, ¿se podrían identificar nuevas secuencias de ARN ribosomal o de familias de proteínas que sugieran la existencia de una nueva rama en el árbol de la vida?…
La respuesta, en base al artículo en PLoS One de Wu et al., es que sí.
Los métodos actuales para hacer las reconstrucciones filogenéticas y evolutivas, no permiten el descubrimiento de nuevas secuencias de ARN ribosomal, ya que sólo se basan en la comparación con secuencias previamente descritas, y si no hay coincidencia o similaridad, no las toman en cuenta, y tal vez podrían ser secuencias que si codifican para otro tipo de ARN ribosomal.
Por otro lado, las relaciones evolutivas basadas en secuencias de ARN ribosomal no son del todo correctas, ya que hay muchos factores que influyen en la posición de un organismo en el árbol de la vida. Por ejemplo, las secuencias incompletas de ARN ribosomal, que pueden ser a causa de mutaciones o de errores en el secuenciamiento, influyen en la posición de un organismo en el árbol de la vida. Por otro lado, la evolución convergente, aquella en que dos organismos completamente diferentes y lejanos evolutivamente, podrían compartir un mismo gen ya que viven en ambientes con condiciones fisicoquímicas similares. De esta manera, podrían estar relacionados —dentro del árbol de la vida—cuando evolutivamente no lo están.
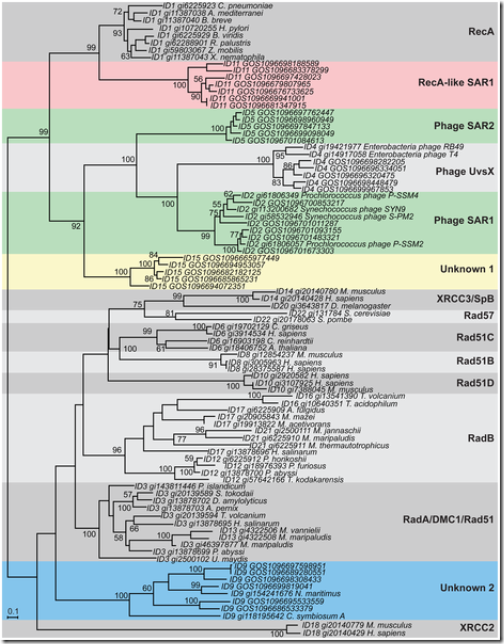
Así que, para superar estos inconvenientes, Wu et al. se enfocaron en el estudio de otras secuencias genéticas, como aquellas que codifican proteínas conservadas en todos los dominios de la vida. La primera que analizaron fueron los genes correspondientes a la superfamilia RecA. Esta superfamilia incluye a genes que codifican para una gran variedad de recombinasas —enzimas encargadas de la recombinación homóloga del ADN— y está presente en casi todas las especies de los tres dominios de la vida: Bacteria (RecA), Eucaria (Rad51, Rad57) y Arquea (RadA, RadB), hasta también está presente en los fagos (UvsX).
Para analizar esta superfamilia de genes, los investigadores dividieron cada familia de genes en pequeños subgrupos. Luego, tomaron a una especie representante de cada subgrupo y las agruparon usando complejos modelos estadísticos basados en el escore obtenido en el BLASTp —una herramienta bioinformática para comparar e identificar secuencias proteicas. Finalmente, se obtuvo un árbol filogenético con 15 grupos, de los cuales 2 (amarillo y turquesa) no se relacionaban con ninguna de las especies y subfamilias catalogadas. Estos dos grupos estaban formados por secuencias que fueron obtenidas en el GOS.
Luego, se hizo el mismo experimento pero, esta vez, usando otra familia de genes conservados llamada rpoB, la cual codifica para una de las subunidades de la ARN polimerasa —enzima encargada de transcribir el ADN en ARN mensajero, que posteriormente será traducido a proteína. En este caso, también se observaron dos grupos (amarillo y fucsia) que no se relacionaban con los otros y contenían secuencias obtenidas en el GOS.
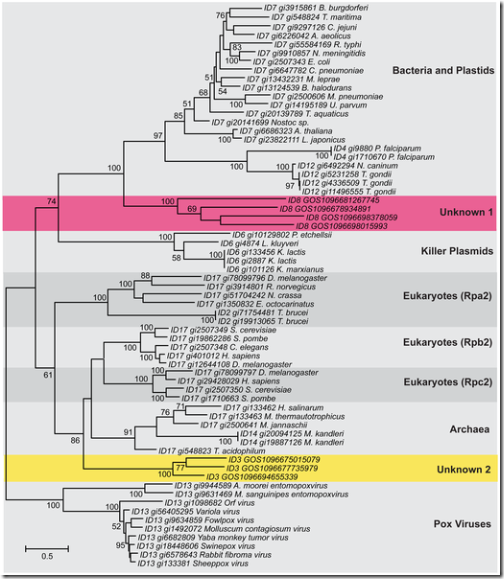
Ninguna de estos grupos encontrados en el análisis de las dos familias de genes encaja en algunas de las ramas del árbol de la vida. Sin embargo, los autores tiene algunos argumentos que podrían explicar esta “nueva” rama en el árbol de la vida.
Por un lado, estas nuevas secuencias que no encajan en las relaciones filogenéticas previamente establecidas podrían ser quimeras, que se pudieron dar como resultado de la combinación de secuencias de ADN de diferentes orígenes. Estas quimeras se pudieron formar al momento de ensamblar todas las pequeñas secuencias generadas en el secuenciamiento shotgun y no serían secuencias reales. También podrían ser algún tipo de recombinación genética de regiones de ADN de diferentes subfamilias. Otra posibilidad indicaría que son parálogos que surgieron como producto de la duplicación de un gen de dicha superfamilia, y que evolucionaron independientemente, hasta diferenciarse de los demás a pesar de tener un origen común.
Y la última posibilidad indicaría que sí se trata de un nueva rama en el árbol de la vida que se podrían haber originado en virus primitivos. Lo que si es cierto es que la cantidad de genes que aún faltan secuenciar y caracterizar es tan basta que todavía nos espera muchas sorpresas. Lo bueno es que las novedosas técnicas usadas en la actualidad nos permiten descubrir nuevas cosas, observando todo a otro nivel, tal como paso con los experimentos de Woese en 1977, donde se descubrió que las Arqueas estaban separadas evolutivamente de las bacterias y de los eucariotas.
Entonces, para poder determinar si estas secuencias, que no forman parte de ningún grupo conocido, corresponden o no a un nuevo dominio del árbol de la vida, se tendría que explorar más daros metagenómicos. Además, estos resultados nos demuestran la importancia de combinar diferentes métodos de reconstrucción filogenética y evolutiva, no solo basándonos en secuencias del ARN ribosomal, sino también en otras secuencias conservadas, como aquellas que codifican para enzima esenciales del metabolismo de todos los organismos vivos.
Referencia:
 Wu, D., Wu, M., Halpern, A., Rusch, D., Yooseph, S., Frazier, M., Venter, J., & Eisen, J. (2011). Stalking the Fourth Domain in Metagenomic Data: Searching for, Discovering, and Interpreting Novel, Deep Branches in Marker Gene Phylogenetic Trees PLoS ONE, 6 (3) DOI: 10.1371/journal.pone.0018011
Wu, D., Wu, M., Halpern, A., Rusch, D., Yooseph, S., Frazier, M., Venter, J., & Eisen, J. (2011). Stalking the Fourth Domain in Metagenomic Data: Searching for, Discovering, and Interpreting Novel, Deep Branches in Marker Gene Phylogenetic Trees PLoS ONE, 6 (3) DOI: 10.1371/journal.pone.0018011
Imagen #1: The Economist.
Esta entrada participa en el II Carnaval de Biología cuyo anfitrión es el blog La muerte de un ácaro.
Sun, G., Dilcher, D., Wang, H., & Chen, Z. (2011). A eudicot from the Early Cretaceous of China Nature, 471 (7340), 625-628 DOI: 10.1038/nature09811