Tal vez algunos se han hecho esta pregunta… si no se han puesto a pensar en ella ahora les explico por qué. A todos nos enseñaron en el colegio que en la división celular el ADN se replica, se forman los cromosomas y estos migran, mita mita, a cada una de las nuevas células hijas. A este proceso se le llama MITOSIS. Seguro a muchos les enseñaron la nemotecnia para recordar el orden de los cuatro pasos principales de este proceso: PRO-METo-ANA-llevarte al-TELO (PROfase, METafase, ANAfase y TELOfase). A ese paso en que los cromosomas son repartidos por partes iguales a las células hijas se le llama SEGREGACIÓN cromosómica y se da gracias al Huso Acromático.
Para recordar: Los centrosomas, que son los centros de organización de los microtúbulos, migran hacia cada uno de los polos de la célula. A partir de ahí empiezan a polimerizarse los microtúbulos, que son proteínas encargadas del movimiento intracelular. Los microtúbulos forman el huso acromático el cual reconoce el centrómero, que es una región del cromosoma que se une a los microtúbulos formando el cinetocoro. Una vez unidos, los microtúbulos jalan a cada una de las cromátides hermanas hacia cada uno de los polos, mientras la célula se va partiendo por la mitad, de esta manera, las dos células hijas tendrán la misma información genética.

Los microtúbulos son proteínas exclusivas de las células eucariotas… entonces, ¿Cómo hacen los procariotas (bacterias) para segregar correctamente su ADN si no tienen ni centrosoma, ni huso acromático, ni centrómeros?
En realidad, las bacterias no son tan simples como creemos, ellas también poseen un sistema similar al que poseen las eucariotas. Este sistema fue primero descubierto en la segregación de los plásmidos bacterianos. Los plásmidos son pequeñas moléculas de ADN circular, algo así como un pequeño cromosoma, el cual debe segregarse en la misma proporción a las bacterias hijas cuando esta se divide. Ahora, este sistema también es el responsable de la segregación del cromosoma bacteriano.
El sistema se llama PAR (de Partitioning) el cual consta con tres componentes principales: ParA, ParB y parS. El ParA sería como el análogo a los microtúbulos, tiene la capacidad de polimerizarse, formar fibras similares a un huso acromático y es dependiente de ATP. parS es una región del cromosoma bacteriano que actúa a manera de un centrómero. Y ParB es una proteína que reconoce la región parS y puede unirse a ParA para formar un cinetocoro bacteriano.
Para explicarlo mejor usaré un artículo del Dr. Jerod Ptacin, del Departamento de Biología del Desarrollo de la Universidad de Standford – quien formó parte del laboratorio de Carlos Bustamante – publicado en el presente número de Nature Cell Biology. Básicamente, el Dr. Ptacin usó dos moléculas fluorescentes para marcar ParA (verde) y ParB (rojo). Como podemos apreciar en la figura de abajo, la fluorescencia verde corresponde a ParA (análogo al huso acromático) y de fluorescencia roja a ParB, que junto a ParA y parS forman un “cinetocoro” bacteriano.
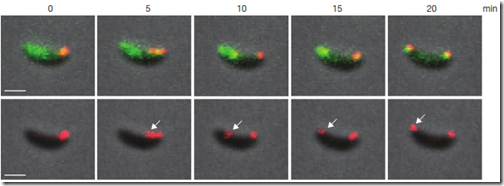
Esta figura explica casi todo el funcionamiento del sistema PAR. Primero observemos las figuras inferiores, las cuales corresponden a ParB. Como ParB reconoce una región del ADN (parS) nos indicará en todo momento donde se encuentra el cromosoma bacteriano recién replicado. Cuando empieza la división bacteriana, la región parS del cromosoma bacteriano se encuentra hacia uno de los polos (t=0 minutos), generalmente hacia el polo donde está ubicado el flagelo (si la bacteria es flagelada), pero siempre es predominante uno de los dos polos. Luego, el cromosoma se replica (t=5min) para formar dos cromosomas bacterianos, donde uno de ellos empieza a migrar hacia el otro polo (t=10, 15 y 20 min).
Ahora, con las figuras superiores veremos como se comporta ParA. En estas figuras se superpuso las fotos de ParB en ParA para ver a los dos interactuar. En t=0min, vemos a ParA difundido en casi toda la bacteria y al cromosoma bacteriano y, a medida que el cromosoma va migrando hacia el otro polo, ParA se va perdiendo en el camino (t=10min) ya que no se observa fluorescencia verde entre los dos cromosomas bacterianos. Debido a la superposición de imágenes, el cromosoma se ve amarillo (Verde + Rojo = Amarillo?… en este caso, así es). A los 20 minutos, cuando el segundo cromosoma llega al otro polo ya casi no hay presencia de ParA en la bacteria.
¿Que podemos concluir de esto? Que la segregación del cromosoma bacteriano se da por despolimerización de la ParA y que ParB es necesario para la unión al ADN y así pueda ser jalado hacia el otro extremo. Pero, esto no queda ahí, después de la segregación, ParA se vuelve a polimerizar y estar lista para una nueva ronda de replicación, sobretodo cuando las bacterias están en fase de crecimiento exponencial. Obviamente que no fue tan fácil llegar a esta conclusión ya que tuvieron que probar con una serie de mutantes, tanto para ParA como para ParB y parS.
 ParA, de por sí tiene afinidad por el ADN, pero en ausencia de ParB (mutante –ParB) no podía despolimerizarse y se mantenía todo el tiempo presente en la bacteria, la segregación del cromosoma bacteriano no podía llevarse a cabo. Pero, si se añadía ParB, automáticamente ParA empezaba a despolimerizarse y se restauraba la segregación del cromosoma. Cuando no estaba presente la región parS en el cromosoma, tampoco ocurría la segregación, ParB estaban diseminados por toda la célula porque no encontraba su sitio de reconocimiento y ParA se ubicaba en uno de los polos, asociado al ADN.
ParA, de por sí tiene afinidad por el ADN, pero en ausencia de ParB (mutante –ParB) no podía despolimerizarse y se mantenía todo el tiempo presente en la bacteria, la segregación del cromosoma bacteriano no podía llevarse a cabo. Pero, si se añadía ParB, automáticamente ParA empezaba a despolimerizarse y se restauraba la segregación del cromosoma. Cuando no estaba presente la región parS en el cromosoma, tampoco ocurría la segregación, ParB estaban diseminados por toda la célula porque no encontraba su sitio de reconocimiento y ParA se ubicaba en uno de los polos, asociado al ADN.
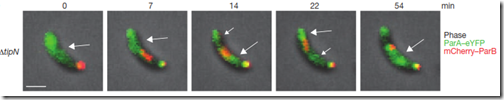
Ya identificamos un análogo a los microtúbulos, uno al centrómero y otro al cinetocoro, pero, ¿el análogo al centrosoma? Hasta este artículo se “desconocía mayormente” (como dicen los wachimanes) la existencia de algún tipo de centrosoma en las bacterias; pero, siempre se sospechó de la presencia de algo que hacía que la ParA se despolimerice en un sentido específico, hacia uno de los polos.
Así que, para demostrar la presencia de algún tipo de centrosoma se usó a la ParA bipolar marcada de verde y se mutó las proteínas que son expresadas en los nuevos polos (a donde migra el cromosoma bacteriano). Se observó que en estos mutantes, la segregación se pausaba constantemente y a veces retrocedía (figura de abajo), como si la ParB no supiera hacia donde ir o la ParA no supiera si despolimerizarse o no. A esta nueva proteína polar la llamaron TipN. Ahora sí el sistema de segregación ya estaba completo, ya tenemos el análogo al centrosoma.
Así que para quede más claro… en dibujitos!!!
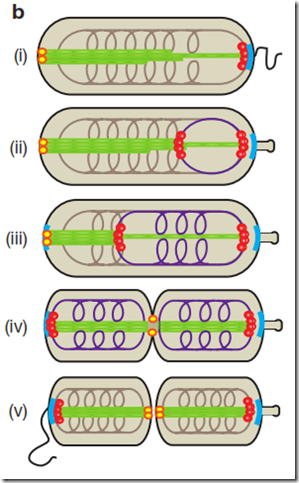
i) ParA (verde) se polimeriza hasta TipN (amarillo con rojo). ParB se une al sitio parS interaccionando con PopZ (proteína que forma parte del flagelo). El cromosoma bacteriano se ve de marrón y está atrapado en uno de los polos vía ParB.
ii) ParB se separa del PopZ y el sitio parS se empieza a replicar. Automáticamente es cubierto por nuevas moléculas de ParB. Mientras tanto, TipN estabiliza a ParA. El complejo ParB y parS se encuentran con ParA y forman el cinetocoro bacteriano.
iii) ParA empieza a despolimerizarse y jala consigo a ParB unido al nuevo cromosoma bacteriano que se está sintetizando.
iv) El nuevo cromosoma es atrapado en el nuevo PopZ en el otro polo de la bacteria a través del complejo ParB-parS. La estructura de ParA se vuelve a reorganizar y TipN es capturado en el sitio de corte para una nueva ronda de división celular. Este mecanismo de movimiento de TipN hacia el sitio de división aún no está bien entendido.
v) La bacteria se termina por dividir quedando con la misma información genética la una y la otra.
Así es como se da la segregación del genoma bacteriano. Este modelo es bastante similar al proceso de segregación de células eucariotas. Aún faltan determinar los mecanismos de regulación de los genes que expresan la ParA y ParB los cuales serían buenos temas de investigación para quienes les gusta la microbiología molecular y la biología del desarrollo.
Referencia:
 Ptacin, J., Lee, S., Garner, E., Toro, E., Eckart, M., Comolli, L., Moerner, W., & Shapiro, L. (2010). A spindle-like apparatus guides bacterial chromosome segregation Nature Cell Biology, 12 (8), 791-798 DOI: 10.1038/ncb2083
Ptacin, J., Lee, S., Garner, E., Toro, E., Eckart, M., Comolli, L., Moerner, W., & Shapiro, L. (2010). A spindle-like apparatus guides bacterial chromosome segregation Nature Cell Biology, 12 (8), 791-798 DOI: 10.1038/ncb2083



















