…Y al fin, salió el artículo científico publicado en Nature sobre Foldit… y soy uno de los “co-autores” del trabajo, junto a otras ~57000 personas, los cuales estamos nombrados en el paper dentro de los autores como “Foldit Players”, no se si lo podré poner en mi CV dentro de mis publicaciones. Lamentablemente, no alcanzó a ser la portada del presente número de la revista, pero aparecer en Nature ya es un gran logro, y si no me equivoco, es la primera vez que un juego de computadora aparece en un artículo de esta revista.
Para, recordar que es Foldit lean el siguiente artículo que publiqué en el 2008: “Foldit, el juego de computadora para científicos”; aunque tendría que rectificarme que no es un juego sólo para científicos, cualquier persona en el mundo que le guste los puzzles lo puede jugar. Ahora pasemos a lo que dice el artículo… pero, antes de entrar al tema, déjenme explicarles algo básico, muy básico de biología molecular para que no se me pierdan. Bueno, los biólogos perdónenme y omitan este cuadro.
Todos los seres vivos tenemos un genoma que es, en términos sencillos, toda nuestra secuencia de genes, la cual está conformado por 4 letras (A, T, C, G) correspondiente a cada uno de los nucleótidos que conforman el ADN. El genoma es como el manual de instrucciones para la construcción de un determinado organismo – desde una insignificante bacteria hasta una inmensa ballena azul. Un perro es perro y un gato es gato según los genes que posea y los genes que exprese.
Cuando un gen se expresa codifica para una proteína. ¿Como se transforma el ADN en una proteína? Ya dijimos que un gen es una secuencia de letras, por ejemplo: ATGGTCGATGCC, claro que un gen no es tan chiquito como el ejemplo, un gen puede tener más de 3000 letras. Si tomamos las letras de este gen y las agrupamos de 3 en 3 (ATG – GTC – GAT – GCC) obtendremos un código. Cada triplete codificará para un determinado aminoácido, conocido como el código genético. Así que traduciendo este código tendremos la siguiente secuencia de aminoácidos: Tyr – Gln – Leu – Arg. (Para los biólogos: ya lo transcribí a ARNm y de ahí lo traduje a aminoácidos).
Una proteína es simplemente una secuencia de aminoácidos. En el ejemplo, nuestra proteína tiene solo 4 aminoácidos, pero en la realidad, una proteína está conformada por decenas de ellos (90, 300, 850, 1000, etc…). Cada proteína, por ser una molécula sumamente grande, tiene una estructura tridimensional determinada y compleja, que está en base a su secuencia de aminoácidos, los cuales interactúan entre sí de diversas maneras hasta plegarse y obtener su estructura final. Ahora viene la pregunta del millón y lo más importante de este artículo: ¿Como determinamos la estructura tridimensional de una proteína?
El método más usado y con mejores resultados para determinar la estructura tridimensional de una proteína es la cristalografía de rayos X. Primero, debemos identificar la proteína, luego purificarla, luego cristalizarla y finalmente someterla a un equipo de difracción de rayos X. Este equipo bombardeará a la proteína cristalizada con rayos X en diferentes ángulos y con diferentes longitudes de onda, y según la ubicación de los átomos en la proteína, los rayos X se desviarán de su trayectoria, la cual es en línea recta (fenómeno conocido como difracción). Se ve bonito y parece sencillo, pero NO!! En primer lugar purificar una proteína es sumamente costoso y cristalizarla es un proceso sumamente largo, complicado y más costoso aún, en promedio cuesta alrededor de $100000 cristalizar una proteína. Además, no todas las proteínas son cristalizables. Las proteínas transmembrana, las cuales son ricas en grupos hidrofóbicos no pueden ser cristalizadas, a lo mucho purificadas sobre solventes orgánicos apolares – aunque hay reportes de investigadores que han llegado a cristalizar algunas de ellas, también en solventes orgánicos – pero debe ser más costoso y laborioso aún. Esta proteínas transmembrana son, para la industria farmacéutica, las más importantes ya que casi todos los virus y bacterias patógenas reconocen específicamente estas proteínas e infectan la célula a través de ellas.
Otro método más moderno para determinar las estructuras tridimensionales de las proteínas es la Espectroscopia de Resonancia Magnética Nuclear (NMR), la cual usa isótopos de los átomos que conforman los seres vivos (C, H, O y N) con diferente número de neutrones y protones (1H, 13C, 15N) de esta manera cambia el momento magnético del átomo y absorben de distintas formas la radiación electromagnética. En función a su comportamiento bajo un campo magnético, se asignan valores de resonancia a cada átomo así como tiempos de relajación y mediante programas informáticos, se obtendrán estructuras donde todos los datos converjan. La ventaja de esta técnica es que pueden determinarse las estructuras de cristales en fase líquidas, pero, su resolución no es tan alta como la cristalografía de rayos X, sólo sirve para proteínas pequeñas o determinados dominios de proteínas más grandes. Si embargo la NMR ha develado estructuras parciales de algunas proteínas transmembrana.
Foldit pretende superar todos estos inconvenientes. Foldit trata de encontrar las estructuras tridimensionales – de proteínas que no las tienen – a partir de sus secuencia de aminoácidos. Ya dijimos que la estructura de una proteína depende de ello. Foldit, a través de una interface gráfica a manera de un juego en línea (así como Dota, GunBound, etc.) busca que las personas – no necesariamente científicos – empiecen a mover, doblar, girar, abrir, cerrar, una proteína problema hasta encontrar la estructura termodinámicamente más estable con la menor energía libre requerida.
Para esto, el juego nos da muchas herramientas como congelar una sub-estructura (una hoja beta o una alfa hélice), acercar dos alfa hélices para formar puentes de hidrógeno (esos dan muchos puntos), esconder los grupos hidrofóbicos (naranja) hacia el centro de la proteína, exponer los grupos hidrofílicos (azules) hacia afuera, evitar que se formen repulsiones inter-atómicas, minimizar las gradientes electrónicas, hasta insertar un fragmento extra y reestructurar las estructuras beta plegadas. Además, para hacer más adictivo el juego, cuanto menor energía libre tenga la proteína más puntos obtendrás y mejor ranking tendrás. También podrás formar comunidades con otros jugadores y tratar de encontrar la estructura de la proteína problema en modo multi-jugador (todos colaboran por el mismo fin). Así como en otros juegos en línea, hay un ranking para las comunidades, puedes intercambiar información en foros y chats y compartir tus logros en facebook y twitter. No sólo es un juego, es toda una red social.
Foldit se basa en la plataforma Rosetta@home el cual es un proyecto bioinformático que busca predecir la estructura tridimensional de cualquier proteína mediante algoritmos estocásticos (probabilístico o al azar) y determinísticos (causal o predeterminado). Pero, para hacer este trabajo requiere de una gran cantidad de procesadores que hagan estos cálculos de manera independiente y plieguen las proteínas hasta encontrar la mejor estructura de todas, tal como Foldit requiere de una gran cantidad de usuarios para lograr el mismo objetivo. Como no existe tal súper-computadora que procese Teraflops de cálculos por segundo, usa las computadoras de miles de voluntarios en el mundo, quienes “donan” sus procesadores mientras están inactivos para desarrollar este proyecto.
Si quieres colaborar con Rosetta@home, descarga el programa e instálalo, tu decides cuanto porcentaje de rendimiento de tu PC pones a disposición de la ciencia. El programa solo correrá cuando la computadora este inactiva, priorizando siempre los trabajos que tu estés haciendo. Si tu PC encuentra la estructura más estable puedes ser puesto en la página principal como el usuario del día, además te dan con el paquete un bonito protector de pantalla donde vez como tu proteína se va plegando.
Las proteínas problema que te pone Foldit están en base al mayor escore obtenido con la plataforma Rosetta. A partir de ahí tu puedes modificarla o reconstruirla… las computadoras no son perfectas.
En el artículo se muestran dos estudios. Primero, los jugadores de Foldit buscan la estructura tridimensional de la proteína a partir de la estructura que obtuvo el mayor escore con la plataforma Rosetta. Para esto, determinaron la estructura nativa (original) de la proteína problema mediante NMR y la compararon con la estructura de la proteína determinada por el jugador de Foldit que alcanzó el mayor escore en ella, y adivinen qué… fueron casi idénticas, además, la estructura determinada mediante Foldit ayudó a develar la ubicación de dos residuos de la proteína que mediante NMR no se pudo hacer. Ver la figura:

escore con la plataforma Rosetta, la cual fue usada como punto de partida.
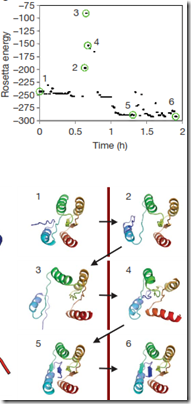
Sin embargo, a pesar de estos resultados prometedores a favor de los jugadores de Foldit, existe una gran dificultad que no se ha podido resolver y la plataforma Rosetta nos gana. Los jugadores de Foldit pueden predecir la estructura de una proteína a partir de una estructura inicial, pero, si parten desde cero, sólo a partir de una cadena proteínica extendida, se les hace muy difícil, por no decir imposible, llegar a la estructura final u obtener un escore mayor al de Rosetta, a pesar que Foldit cuenta con todas las herramientas necesarias para hacerlo. Esto indica que hay un perfecto balance entre la computadora y la mente humana… la primera da la estructura de partida en base al escore más alto obtenido por su plataforma Rosetta, mientras que el jugador de Foldit resuelve las características incorrectas, mejora el plegamiento y obtiene la estructura final. Con estos resultados podemos estar a puertas de la predicción de estructuras proteicas de novo (desde cero, de la nada), en este caso, a partir de la secuencia aminoacídica.
Para concluir, espero no haberlos aburrido y me hayan entendido por lo menos el 50% de lo que escribí, es el único juego que tiene una bran relevancia científica, puede ser jugado por cualquier persona en el mundo ya que las herramientas están puesta con un lenguaje nada técnico, lleno de colores y bastante interactivo. Es lo más cercano que tenemos a un híbrido humano-computadora usado para el desarrollo de problemas científicos complejos. Además, los costos que nos ahorrará una vez que la técnica sea perfeccionada. Así que todos a jugar Foldit en estas vacaciones.
Referencia:
Cooper, S., Khatib, F., Treuille, A., Barbero, J., Lee, J., Beenen, M., Leaver-Fay, A., Baker, D., Popović, Z., & players, F. (2010). Predicting protein structures with a multiplayer online game Nature, 466 (7307), 756-760 DOI: 10.1038/nature09304







Estimado David:
ResponderBorrarEstoy realizando un trabajo que ha llevado a preguntarme cuánto tiempo y/o dinero cuesta resolver la estructura de una proteína. He visto en tu artículo que en el caso de la difracción de rayos X la cifra asciende a $100000 de promedio. Me gustaría utilizar este dato pero necesito referenciarlo, ¿podrías hacerme el favor de indicarme dónde has obtenido esta información? Muchas gracias por adelantado.
Hola
BorrarEn realidad no recuerdo exactamente de donde saque el dato, pero viendo las referencias que tomé lo más probable es que lo saqué de la página de Wikipedia de Roset@Home, la cual hace referencia a su vez al libro de Hoboken "Structural Bioinformátics".
Espero que te sirva de ayuda.
Un saludo.
¡Claro que me sirve de ayuda! Muchísimas gracias.
Borrar