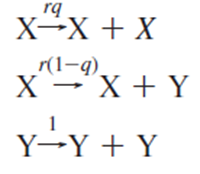[Artículo publicado originalmente el 23 de Abril del 2010]
¿Quién fue primero, el huevo o la gallina? Una pregunta que ha hecho pensar a muchos, pero no desde el punto de vista literal o filosófico, sino genético y evolutivo. O como diría Richard Dawkins: '
la gallina no es más un invento del huevo para producir más huevos'.
En lo que concierne al origen de la vida, siempre nos hemos preguntado que se formó primero: la maquinaria capaz de replicar el material genético o el material genético capaz de codificar (crear) esa maquinaria. La respuesta fueron las
ribozimas, pequeñas secuencias de ARN con la capacidad de auto-replicarse.
Vamos por partes. Todos los organismos vivos almacenamos nuestro “manual de construcción y funcionamiento” en secuencias de nucleótidos (ADN), y a toda esta información le llamamos genoma, el cual debe es replicado de manera precisa a través una enzima conocida como la ADN polimerasa. La copia de cada nucleótido siempre presenta una oportunidad de error —poner un nucleótido por otro— conocido como tasa de mutación por nucleótido (μ).
Por ejemplo, digamos que tenemos una pequeña secuencia de ADN de 100 nucleótidos de longitud y una μ del 2%. Entonces, después de replicar nuestra secuencia modelo tendremos dos secuencias: i) una normal (la que sirvió de molde) y ii) la replicada, que presentará dos nucleótidos mutados (diferentes al original). Tal vez dos mutaciones puntuales parezcan poco, pero la información que se transmitirá será errada, y tal vez genere descendientes que no podrán sobrevivir, perdiéndose por completo dicha información.
De este ejemplo podemos concluir con dos cosas: cuanto más larga es la secuencia o cuando mayor sea la tasa de mutación, más nucleótidos mutados se irán acumulando. Entonces, el tamaño de una secuencia será inversamente proporcional a su tasa de mutación (μ). Si está entendido esto, pasamos al siguiente punto.
En las células modernas, la que todos los organismos tenemos actualmente, existe una maquinaria que repara los nucleótidos mal ubicados (mutados), reduciendo considerablemente esta tasa de mutación a valores que varían entre 10-6 (0,000001) y 10-10 (0,0000000001), lo que permitiría —basándonos en la premisa anterior— a tener secuencias más largas. Sin embargo, los virus existen cerca del límite, ya que tienen secuencias relativamente largas con respecto a su tasa de mutación, la cual es bastante alta.
Pero, en el momento en que se empezó a originar la vida, a través de las primeras secuencias informativas, no habían maquinarias encargadas de replicar y corregir los errores. Además, estudios previos determinaron que la tasa de mutación en una replicación no enzimática —como la que hacen las ribozimas— es cercana al 20%, la cual es bastante alta, y aplicando la relación entre tamaño de secuencias y tasa de mutación, el genoma solo consistiría de cinco nucleótidos. Sin embargo, para formar una ribozima se necesita de al menos una secuencia de 30 nucleótidos de largo. Esta discrepancia genera la paradoja de la aparición de secuencias funcionales: una secuencia funcional será tan larga que acumulará tantas mutaciones, que al final de unos cuantos ciclos de replicación, las secuencias resultantes serán tan diferentes a la original que la secuencia funcional se habrá perdido por completo.
Hubo algunas aproximaciones que intentaron dar salvedad a esto. Por ejemplo, un análisis encontró que una ribozima podía mutar el 25% de sus nucleótidos y aún así no perder su capacidad autoreplicativa. Entonces, el tamaño físico de su genoma excede en un 25% la longitud de su secuencia informativa. De aquí surge un término conocido como umbral de error, que permitiría la emergencia de secuencias tan largas como sean posibles para seguir siendo funcionales. Pero, también vimos que a mayor longitud, más mutaciones se acumularán… Otra paradoja!
Todo esto vamos a explicarlo, de manera más sencilla, con las ecuaciones del modelo de Eigen (1971):
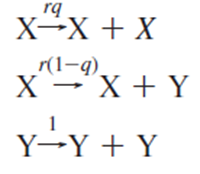 X
X, es la longitud de la secuencia molde;
r, es la aptitud genética (fitness) que debe ser >1; y
q, es la probabilidad de replicar
X sin ningún error, siendo q=(1-μ)
L. De esta manera, si la tasa de mutación es más grande, la probabilidad de replicar sin errores (q) será menor, y si la longitud del genoma es mas grande (L), que también será más chiquito.
a. En la primera ecuación vemos como sería en condiciones óptimas. La secuencia X, con un q de 1 (100% de probabilidad de replicar sin errores) y con un buen fitness, generará más secuencias X idénticas, así que la información se perpetuará.
b. Pero, si la tasa de mutación es mayor veremos que se obtendrán secuencias idénticas (X) y secuencias mutantes (Y), si mayor es la tasa de mutación o más larga la secuencia, Y será mayor (Ecuación 2). Además, Y también se replicará (sin importar su tamaño o tasa de mutación, de todas maneras saldrá mutada, por eso ponemos un valor de 1). Esto provocará que el número de mutantes aumente más rápido que el número de secuencias originales. Las secuencias mutantes consumirán todos los recursos y en una población finita, la secuencia original (X) eventualmente desaparecerá.
La única salvedad es reducir la velocidad en que se producen las secuencias mutantes, de esta manera evitamos que éstas se acumulen y consuman todos los recursos, permitiendo que las secuencias informativas emerjan y persistan a través del tiempo hasta llegar a evolucionar. No estamos tocando ni la tasa de mutación ni el tamaño del genoma, vamos a enfocarnos en la velocidad en que las secuencias mutantes se generan.
Entonces un grupo de investigadores liderados por el Dr. Sudha Rajamani de la Universidad de Harvard demostraron que la replicación de secuencias correctas se da más rápido que la generación de secuencias mutantes usando sistemas de replicación no enzimáticos, bajo las condiciones que se presumen que hubo al originarse la vida. Sin dudas, un hallazgo importante para entender un poco más sobre el posible origen de la vida.
Para hacerlo comprensible para todos —sin entrar a complicadas metodologías— lo que hicieron estos investigadores fue diseñar cuatro secuencias iniciadoras de la replicación (primers), cuatro moldes de ADN para hacer pruebas de incorporación de nucleótidos incorrectos “misincorporation” (para ver como afecta la velocidad de replicación de una secuencia que ha incorporado un nucleótido por otro), y cuatro moldes de ADN para las pruebas de extensión después de un mal emparejamiento “mismatch” (para ver la velocidad de replicación de una secuencia que se ha unido a un primer con un nucleótido incorrecto), recordemos que A se empareja con T, y G con C:
Ej:
Primer: 5’ GG GAT TAA TAC GAC TCA CTG-
misincorporation: 5’ AGT GAT CTA CAG TGA GTC GTA TTA ATC CC
mismatch: 5’ AGT GAT CTA TAG TGA GTC GTA TTA ATC CC
Emparejando el primer con cada secuencia molde:
5’ AGT GAT CTA CAG TGA GTC GTA TTA ATC CC
GTC ACT CAG CAT AAT TAG GG 5’
5’ AGT GAT CTA TAG TGA GTC GTA TTA ATC CC
GTC ACT CAG CAT AAT TAG GG 5’
En el primer emparejamiento el nucleótido que sigue es una T; pero, con una tasa de mutación alta, es probable que un nucleótido diferente se meta, que pasaría con la replicación en ese caso?
En el segundo emparejamiento vemos que hay mal emparejamiento del primer y la cadena molde, una T se emparejó con una G, cuando debería haber sido una A como en el emparejamiento anterior, que pasará con la replicación en este caso?
Rajamani encontró que en ambos casos hay un estancamiento en la replicación. En el primer caso, las secuencias correctas se replicaban hasta diez veces más rápido que cuando se incorporaba un nucleótido que no era el adecuado (misincorporing), y este estancamiento dependía del tipo de nucleótido, cuando era G o C era más rápido que cuando era T o A, esto porque G y C tienen tres enlaces (puentes de hidrógeno) mientras que A y T tienen dos.
En el segundo caso fue más evidente, cuando había un mismatch, la velocidad de replicación se reducía hasta en 300 veces. Estos dos ordenes de magnitud de ventaja permitieron que las secuencias correctas (con información relevante) sean las más abundantes.
De esta manera, a pesar que la tasa de mutación era alta y las secuencias informativas eran largas, este estancamiento es de suma importancia para evitar la catastrófica paradoja. Así que, este tipo de replicación no enzimática potencialmente pudo incrementar el número de secuencias lo suficientemente largas como para ser informativas, a pesar de la alta tasa de mutación. Además, permitió que los genomas primitivos codifiquen más secuencias o secuencias más largas con una alta actividad; y este estancamiento, pudo ser la base para la evolución de las maquinarias de corrección de errores y hasta de la replicación de secuencias.
Referencia:
 Rajamani, S., Ichida, J., Antal, T., Treco, D., Leu, K., Nowak, M., Szostak, J., & Chen, I. (2010). Effect of Stalling after Mismatches on the Error Catastrophe in Nonenzymatic Nucleic Acid Replication Journal of the American Chemical Society, 132 (16), 5880-5885 DOI: 10.1021/ja100780p
Rajamani, S., Ichida, J., Antal, T., Treco, D., Leu, K., Nowak, M., Szostak, J., & Chen, I. (2010). Effect of Stalling after Mismatches on the Error Catastrophe in Nonenzymatic Nucleic Acid Replication Journal of the American Chemical Society, 132 (16), 5880-5885 DOI: 10.1021/ja100780p Mann, R., Ali, J., Hermann, S., Tiwari, S., Pelz-Stelinski, K., Alborn, H., & Stelinski, L. (2012). Induced Release of a Plant-Defense Volatile ‘Deceptively’ Attracts Insect Vectors to Plants Infected with a Bacterial Pathogen PLoS Pathogens, 8 (3) DOI: 10.1371/journal.ppat.1002610





















![megavirus[5] megavirus[5]](http://lh5.ggpht.com/-TpDQtaO9vOg/TpOi3Pzpx0I/AAAAAAAAAYc/bCjKwRLSHxE/megavirus%25255B5%25255D_thumb%25255B8%25255D.jpg?imgmax=800)