Ya vimos como, hace un par de semanas, Venter publicó su trabajo sobre su primer organismo que funciona con un genoma hecho de manera artificial. Sin embargo, todavía falta algo muy importante, algo con que la biología sintética podría consolidarse. Diseñar y ensamblar genomas se ve ‘relativamente sencillo’ a comparación de diseñar y ensamblar una estructura biológica más compleja como una membrana o un organelo. Si se lograría hacer esto estaríamos muy cerca de crear vida. Pero, no se asusten, aún faltan muchos años de desarrollo científico y tecnológico para poder realizar un proyecto como este, aunque ya hay científicos que están empezando a trabajar en esto.
Si queremos ensamblar una estructura biológica compleja de manera sintética, debemos empezar por la más sencilla (tal como Venter buscó el genoma más sencillo). ¿Cual creen que puede ser? Sin dudas sería la de los virus… una cápside viral. Muchos dirán que los virus no son células y que no se sabe si son considerados seres vivos o no, pero no vamos a entrar en la discusión de esto. Obviamente los virus no son células, pero su cápside (lugar donde esta almacenado su material genético, en los fagos se presenta como una cabeza) está formada por proteínas ensambladas de manera armónica, formando estructuras geométricas perfectas.
Sun et al. del departamento de química aplicada de la Universidad de Tokio, se inspiró en las cápsides espéricas de los virus para sintetizar sus propias esferas químicas con esa misma disposición. Pero, hacer todo esto no es nada sencillo. Las cápsides de los virus son estructuras geométricas sumamente complejas, formadas por subunidades poliédricas matemáticamente bien definidas. Sun vio que los poliedros esféricos se basan en la siguiente fórmula general: MnL2n, donde M son los puntos de coordinación de cada plano de la esfera poliédrica y L los ligandos que unen los puntos M (Fig. A). Por ejemplo: en el grafeno, M serían los átomos de Carbono y L los enlaces covalentes. En una cáside, M podría representar a las proteínas y L a los puentes peptídicos que las unen.
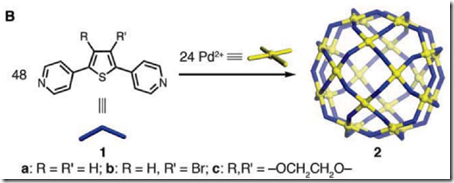
Como podemos ver en la figura, estas estructuras se asemejan mucho al andamiaje (scaffold) de una cápside. Si podemos formar esta estructura, nos podría servir como un soporte o esqueleto para formar estructuras biológicas más complejas. Así que Sun et al. usaron átomos de paladio (el mismo que usa IronMan para su mini-reactor nuclear) como puntos de coordinación (M) de los ligandos (L) que son moléculas de dipiridiltiofeno. La disposición debía ser entrópicamente favorable para que la estructura pueda mantenerse. Recuerden que la tercera ley de la Termodinámica dice que todo tiende al caos o desorden, es estadísticamente improbable que una mezcla de compuestos se agreguen y formen otro más complejo (entropía). Para que sea entrópicamente favorable el valor de “n" está limitado a los siguientes valores: 6, 12, 24, 30 y 60.
La síntesis del dipiridiltiofeno fue relativamente sencillo usando el procedimiento de Suzuki-Miyaura. Para formal la estructura de M24L48 usaron 5uM de Pd(NO3)2 y 10uM de diíridiltiofeno y lo mezclaron en 0.7ml de DMSO-d6 (un solvente marcado con Deuterio para el posterior análisis mediante espectroscopía de resonancia magnética o NMR) a 70°C por 17 horas (Fig B). Después del tiempo de reacción y el análisis de NMR observaron los picos característicos del piridil, el tiofeno y la esfera.
Mediante un espectrometría de masas particular (CSI-TOF-MS) se determinó la composición química en base a su peso molecular (~21946Da) la cual fue {[Pd24(C14H9BrN2S)48]48+*48(BF4-)}, la cual correspondía a M24L48. Luego, para determinar su estructura exacta, se lo cristalizó y se le sometió a un equipo de difracción de rayos X y pudieron observar la estructura rombicuboctaédrica, con 8 caras triangulares y 18 rectangulares. El diámetro de la esfera fue de 5nm.
Este, sin dudas es el primer paso hacia el autoensamblaje de sistemas biológicos complejos para el diseño de estructuras más resistentes y con nuevas funciones en base a las proteínas sintetizadas por un genoma. A partir de esto podríamos diseñar transportadores de nuevos compuestos farmacéuticos el cual podría focalizar la liberación de sustancias químicas o radiactivas en el tratamiento del cáncer, sin afectar las células sanas. Podríamos cubrir la superficie con anticuerpos específicos para estas células cancerígenas y diseñar tratamientos más eficientes.
Referencia:
Sun, Q., Iwasa, J., Ogawa, D., Ishido, Y., Sato, S., Ozeki, T., Sei, Y., Yamaguchi, K., & Fujita, M. (2010). Self-Assembled M24L48 Polyhedra and Their Sharp Structural Switch upon Subtle Ligand Variation Science, 328 (5982), 1144-1147 DOI: 10.1126/science.1188605







0 comentarios:
Publicar un comentario
Se respetuoso con tus comentarios y críticas. Cualquier comentario ofensivo será eliminado.