Casi 10 años después de la presentación del primer borrador del genoma humano, y siete años después de su versión final, aún parece que no ha podido ser aprovechado ni ha podido responder muchas preguntas que se plantearon al momento de elaborar el proyecto: que características nos hacen susceptibles a determinadas enfermedades o de que depende nuestra respuesta a determinadas drogas. Para poder saber esto, se necesita más que conocer el genoma humano, se deben secuenciar muchos más, de individuos con características especiales que nos permitan observar las diferencias entre ellos a nivel genético.
Fue por esta necesidad que nació el proyecto “Estudio de Asociacmiento Genómico Amplio (GWAS: Genomic Wide Association Studies) que busca secuenciar los genomas de miles de personas para identificar las variaciones genéticas y relacionarlos con características específicas. El proyecto “1000 genomes” es parte de GWAS, tiene el objetivo de buscar aquellas variantes genéticas que se presentan al menos en el 1% de los individuos estudiados. En otras palabras, si tenemos 1000 genomas, el proyecto busca las mismas diferencias genéticas que se presentan al menos en 10 individuos diferentes.
Secuenciar un genoma humano aún sigue siendo muy costoso, a pesar que las tecnologías de secuenciamiento han avanzado un montón. Es lógico pensar que cuanto más largo es un genoma mayor es el costo. Como el genoma humano es tan largo, debe ser cortado en pequeños fragmentos y leerlos uno por uno, para luego ensamblarlos usando computadoras. Pero, no basta con secuenciar una vez el genoma ya que pueden haber fragmentos que se pierdan en el proceso, otros que sean mal secuenciados y otros que se secuencien repetidas veces. Estos problemas pueden generar vacíos al momento de ensamblar el genoma. Así que para tener un genoma completo, sin errores y espacios en blanco, la profundidad del secuenciamiento debe ser mayor. Para esto, con los secuenciadores con los que hoy contamos, debemos secuenciar el ADN de una persona unas 28 veces (28X) para obtener un genoma sin espacios vacíos o errores. Además, se debe contar con genomas patrones para poder hacer el ensamblaje de los pequeños fragmentos generados.
Así que el día de ayer se publicó en Nature (el artículo está bajo una licencia de Creative Commons, así que pueden descargarlo libremente) el piloto del proyecto 1000 genomes, el cual analizó todo el genoma de 179 individuos, con una cobertura de 3.6X en promedio, generando un catálogo de más de 8 millones de variantes genéticas de un nucleótido, conocidos como los famosos SNPs. También catalogaron más de 1 millón de variantes estructurales debido a inserciones o deleciones (indels) de secuencias de ADN.
Además, hay que recordar que sólo el 1.5% de todo nuestro genoma es expresado en proteínas, así que los genes estarán dentro de este 1.5%. Los genes dentro del genoma están conformados por exones (regiones que se llegarán a expresar) e intrones (regiones que serán procesadas y removidas del ARN mensajero). Debido a que muchas de las variaciones genéticas se encuentran en los exones y están envueltas directamente con determinadas características de importancia biomédica (resistencia o susceptibilidad a enfermedades, preponderancia a cáncer, diabetes, Alzheimer, etc.), los investigadores secuenciaron, en promedio, 1.4Mb de exones en 697 individuos, con una cobertura de 56X. Aproximadamente, 1.4Mb de exones corresponde a un poco más de 1000 genes, donde encontraron casi 13000 SNPs y 100 indels.
Secuenciar sólo los exones, es sin dudas, muy efectivo si hablamos de costos, ya que la información que proveen es sumamente importante; pero, tampoco hay que subestimar al ADN no codificante ya que en él se encuentran muchos factores que regulan la expresión de los genes.
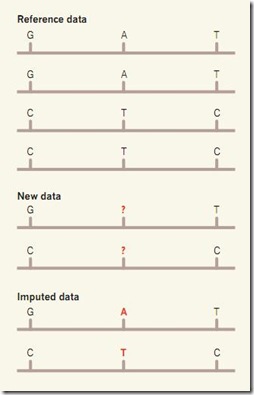
Una gran desventaja del secuenciamiento con baja cobertura es que se generan una gran cantidad de errores y espacios vacíos, si bien se ha desarrollado una metodología que permite “llenar” los espacios en blanco. Esta metodología, según los investigadores, permite tener una tasa de error baja, la cual varía entre el 1 y 3%. Aún así, el número de variantes podría estar sobre-estimado, ya que este 1 a 3% de errores podrían ser consideradas como variantes genéticas cuando en realidad no las son.
Además, otra observación importante es que estos datos son tomados en base ha haplotipos, sin embargo, sabemos que nuestro genoma es diploide, así que caracterizar variantes genéticas en sitios heterocigotos es mucho más complicado y la tasa de error aumenta considerablemente de 5 al 30% según el presente estudio. Aún así, la propuesta de combinar un secuenciamiento completo de baja cobertura con un secuenciamiento de exones de alta cobertura, parece ser una muy buena estrategia, mucho mejor que las técnicas tradicionales. Aún falta mejorar su metodología de corrección de errores, pero, este proyecto recién se inicia, así que con el paso del tiempo se irá mejorando y el conocimiento del genoma humano pasará a un nuevo nivel.
Referencias:
The 1000 Genomes Project Consortium (2010). A map of human genome variation from population-scale sequencing Nature, 467 (7319), 1061-1073 DOI: 10.1038/nature09534
Nielsen, R. Genomics: In search of rare human variants. doi: 10.1038/4671050a





0 comentarios:
Publicar un comentario
Se respetuoso con tus comentarios y críticas. Cualquier comentario ofensivo será eliminado.