La adquisición de resistencia a diferentes antibióticos por parte de muchos microorganismos patógenos es algo muy conocido por todos nosotros. Tal como en una carrera armamentista, mientras que los científicos desarrollan antibióticos más potentes y efectivos, los microorganismos desarrollan mecanismos de resistencia que contrarrestan sus efectos. Sin embargo, los estudios realizados para determinar las causas de la resistencia se han enfocado principalmente a nivel genético (mutaciones), mas no sobre las interacciones que pueden haber entre los microorganismos de una determinada población.
El Dr. Henry Lee y sus colaboradores del Howard Hughes Medical Institute hicieron un interesante estudio y demostraron que los microorganismos actúan de manera colectiva para resistir al efecto de los antibióticos. Lo que hizo Lee et al. fue coger una cepa de E. coli silvestre y cultivarla por 10 días en un medio suplementado con un antibiótico – la Norfloxacina – a una concentración que inhiba el 60% de su crecimiento. A esta concentración le llamaron MIC (concentración mínima inhibitoria). Cada día, los investigadores determinaban el MIC de la población y ajustaban la concentración del antibiótico a este valor.
A parte de eso, los investigadores cada día tomaban 12 muestras del cultivo y analizaban su MIC individualmente y observaron que en casi todos los aislamientos su MIC era menor al MIC de la población en el cultivo, pero, sorprendentemente aislaron un mutante que tenía un MIC superior al MIC poblacional. En otras palabras, de las 120 muestras tomadas en los 10 días, 119 tenían un MIC igual o menor al MIC de la población en el cultivo primario y sólo un aislamiento tuvo un MIC mucho mayor a este (ver Figura).
La falta de aislamientos de mutantes con un MIC superior al MIC poblacional del cultivo indica que su abundancia en el medio es mínima y los investigadores especulan que son estos pocos mutantes con alto MIC los que benefician a los numerosos individuos con un MIC bajo, por esta razón, las muestras obtenidas del cultivo principal casi siempre tenían un MIC bajo. Pero, que tienen de diferentes los mutantes que tienen un alto MIC con los que tienen un bajo MIC.
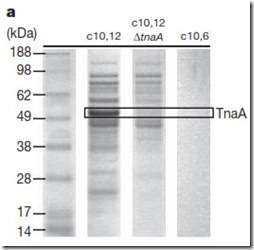
El medio de cultivo donde crecen los mutantes con un MIC alto es líquido, así que la proteína responsable de su alta resistencia al antibiótico estará disuelta en ella. Pero, no será esta proteína la única disuelta sino muchas más que el microorganismo produce normalmente. Así que para separar cada una de las proteínas disueltas en el medio se hace una técnica llamada electroforesis. Se prepara un gel (parecido a una gelatina), que si lo vemos al microscopio veremos que es como una red. Ponemos un volumen del líquido donde están disueltas mis proteínas en la parte superior y con una corriente eléctrica hacemos que bajen por el gel hacia la parte inferior. Las proteínas más chiquitas pasarán con mayor facilidad por la red, mientras que las más grandes quedarán atrapadas en la parte superior, de esta manera separamos cada una de las proteínas en función a su peso (kDa). Más pesadas… más grandes!
Como vemos en la figura, hay una bandita bien pronunciada (c10,12). Esa bandita fue extraída del gel, luego purificada y finalmente analizada por un espectrómetro de masas. Con esta herramienta se determinó que esta banda correspondía a la proteína TnaA. Para comprobar si en verdad era esta proteína diseñaron un mutante donde esta proteína está ausente y repitieron el experimento (c10,12 ∆tnaA), como se ve en la figura, esa misma banda ya no aparece al igual que en los mutantes con un MIC bajo (c10,6).
Pero, a que proteína corresponde la TnaA? La TnaA es una enzima, la triptofanasa, la cual rompe el triptófano en amonio, purivato e indol. Entonces, uno de estos tres componentes debe ser el responsable del incremento del MIC en estas bacterias. El indol es una importante molécula señalizadora envuelta en la tolerancia al estrés en E. coli. Lo que proponen los autores es que el indol producido en grandes cantidades por los mutantes con alto MIC protege a los individuos con bajo MIC. Usando un HPLC (una cromatografía de alta presión) cuantificaron cuanto indol producían tanto los mutantes con alto MIC y bajo MIC con y sin antibiótico. Sin antibiótico ambos producían la misma cantidad de indol, pero con antibiótico, los debajo MIC ya no lo hacían mientras que los de alto MIC no se veían afectados.
Entonces, si el indol es el responsable de aumentar el MIC de los mutantes, lo más lógico es que si le agregamos indol de manera artificial a los mutantes de bajo MIC lograremos que resistan concentraciones más altas de antibiótico. Los investigadores realizaron este experimento y añadieron indol al mutante de MIC bajo consiguiendo que aumente su MIC, tal como lo habían predicho. además, probaron este mismo experimento usando otro antibiótico, la Gentamicina, y encontraron los mismos resultados que con la Norfloxacina.
Finalmente el modelo queda así:
Este estudio nos da nuevas claves de como se desarrolla la resistencia en los microorganismos patógenos. Muchos pensaban que las mutaciones que otorgaban resistencia a un microorganismo eran adquiridos por todos los individuos de una población, ya sea de manera directa o por transferencia horizontal de genes (los genes de resistencia pasan de un individuo a otro por conjugación o transformación genética), pero hemos visto que sólo son algunos individuos quienes adquieren esta mutación y de manera altruista protegen a los más débiles ofreciéndoles moléculas protectoras y otro tipo de señales químicas que les permiten resistir a los antibióticos.
Este estudio debe ser tomado en cuenta por los desarrolladores de antibióticos para tratar de cambiar las estrategias para combatir a los microorganismos patógenos, reformulando las dosis, la prescripción y el tiempo de duración del tratamiento, a fin de evitar que este tipo de estrategia de resistencia perjudique a los pacientes. Sería bueno poner como aditivos a los agentes terapéuticos compuestos que capturen o degraden estas moléculas señalizadoras a fin de evitar que estos microbios “altruistas” hagan de las suyas.
Referencia:
Lee. HH, Molla. MN, Cantor. CR, & Collins. JJ (2010). Bacterial charity work leads to population-wide resistance Nature, 467, 82-85 DOI: 10.1038/nature09354








0 comentarios:
Publicar un comentario
Se respetuoso con tus comentarios y críticas. Cualquier comentario ofensivo será eliminado.