¿Por qué envejecemos? Es una de las preguntas más intrigantes de la biología. Existen muchas hipótesis, todas muy convincentes, pero funcionan sólo en ciertas condiciones. Las dos principales teorías más aceptadas tienen que ver con los telómeros y con las mitocondrias. Ayer, científicos norteamericanos liderados por el Dr. Ergün Sahin del Instituto del Cáncer Dana-Farber, encontraron la molécula clave que unifica estas dos teorías. Los resultados fueron publicados en Nature.
Los síntomas del envejecimiento son conocidos por todos. Primero viene la pérdida de elasticidad de la piel y luego viene un progresivo deterioro y falla en el funcionamiento de nuestros principales órganos como el corazón, hígado, riñones, cerebro, etc. ¿A qué se debe?
Una de las causas se encuentra en el núcleo de las células. El ADN no puede replicarse por siempre, así que nuestras células no podrán regenerarse por siempre, sobre todo los tejidos que tienen una alta tasa de división celular, como el tejido intestinal. Esto se debe a que en cada proceso de replicación del ADN hay una pérdida, la cual se da porque la enzima responsable de este proceso no puede replicar los extremos de los cromosomas por no tener donde aferrarse.
Para evitar perder importante información genética debido a esta ‘falla en el diseño’, los extremos de los cromosomas estas protegidos por unas secuencias de ADN repetidas que se extienden por varios miles de pares de base. Estas secuencias repetidas son conocidas como los telómeros. Pero, los telómeros se irán acortando con cada división celular, hasta que finalmente se agotarán. Si ya no hay telómeros, habrá pérdida de información genética importante, la cual podría generar células defectuosas que podrían desarrollar un cáncer.
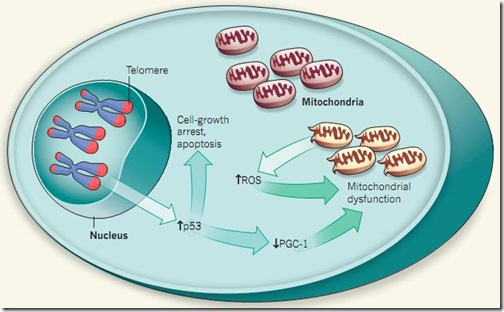
Para evitar esto, las células detectan cuando los telómeros están muy cortos y activan la expresión de un factor de transcripción conocido como la p53. La p53 es conocida como el guardián del genoma, ya que se activa ante la presencia del daño en el ADN, deteniendo su replicación, activando los mecanismos de reparación, y si ya no hay solución, induciendo la muerte celular programada (apoptosis). Con la vejez, no tendremos más telómeros y nuestras células dejarán de dividirse y morirán, provocando disfunciones en los tejidos y órganos.
Por otro lado, la otra causa del envejecimiento se encuentra en las mitocondrias, quienes son las responsables de proveer de toda la energía que requieren las células para funcionar. Las mitocondrias también tienen su propio material genético, el cual codifica para las proteínas requeridas para la maquinaria productora de energía —la cadena transportadora de electrones— y también es propensa a sufrir mutaciones.
Así que, con los años, las mitocondrias irán acumulando mutaciones hasta que finalmente la producción de energía para la célula se verá afectada. Al no haber energía, la célula no podrá cumplir sus funciones y dejará de dividirse y proliferarse. Los tejidos no podrán regenerarse y empezarán a fallar, especialmente los tejidos con bajas tasas de división celular (quiescentes) como el corazón y el cerebro.
Al parecer estos dos mecanismos de envejecimiento funcionan de manera independiente, pero sus efectos sobre los tejidos y órganos pueden ser concertados. Sahin y sus colaboradores descubrieron el nexo entre estos dos. Estudios previos en ratones modificados para que expresaran fallas en los telómeros demostraron que también había una reducción en la capacidad de proliferación de sus células, lo que indicaba que las mitocondrias también podían ser afectadas. Fue así que Sahin et al. hicieron unos experimentos con los mismos ratones y demostraron que las mitocondrias mostraban fallas en su funcionamiento, así que de alguna manera estaban ligados las fallas en los telómeros con las fallas en las mitocondrias.
Las mitocondrias poseen dos principales reguladores de la expresión de sus genes: la PGC-1α y la PGC-1β. Durante el envejecimiento, estos dos factores reducen sus niveles de expresión. Al analizar los resultados de los experimentos con los ratones modificados, también se encontró una reducción en la expresión de las proteínas PGC-1, los cuales provocaban fallas cardiacas y hepáticas, principales indicios de envejecimiento.
Entonces, ¿cómo hacía la falla en los telómeros para inactivar a las proteínas PGC-1? Los investigadores se enfocaron en la p53, ya que este factor de transcripción es el que principalmente se expresa cuando hay fallas en los telómeros, y además, tiene la capacidad de regular muchas funciones celulares, tal vez una de ellas tenía que ver con las mitocondrias.
Sahin et al. encontraron que cuando los niveles de p53 aumentaban, los niveles de PGC-1 disminuían. Este simple resultado fue clave para poder unificar las dos principales teorías del envejecimiento. Para corroborar estos resultados, redujeron los niveles de expresión de la p53 en los ratones modificados y vieron que automáticamente la expresión de las PGC-1 volvían sus niveles normales. Fue así que Sahin et al. presentaron el siguiente modelo:
Este modelo no sólo permite relacionar las dos principales teorías del envejecimiento en una, sino también ayuda a explicar como se da el envejecimiento en tejidos con altas tasas de división celular y en tejidos quiescentes mediante el mismo mecanismo.
Sin embargo, este modelo también ha generado muchas preguntas. Por ejemplo, en algunos cáncer, los bajos niveles de p53 provocan una caída en la función mitocondrial, lo contrario a lo encontrado por Sahin. Entonces, ¿puede ser que la p53 actúe de manera diferente según el tejido? Por otro lado, cuando se reducen los niveles de PGC-1 en las mitocondrias, se ha observado que hay una mayor producción de especies reactivas de oxígeno las cuales son sumamente tóxicas para las células porque dañan el ADN. De esta manera, se aceleraría el proceso de envejecimiento a través de la mitocondria, ya que las mutaciones en su ADN se acumularían más rápido, provocando una deficiencia energética más acentuada.
Aún falta investigar más a fondo como se da este mecanismo. De poder entenderlo podríamos diseñar estrategias más efectivas para reducir el envejecimiento. Reducir el envejecimiento es completamente diferente a prolongar la vida. Para que quereos vivir más si a partir de los 60 años ya empezamos a sufrir de los achaques de la vejez, lo ideal vivir más, pero jóvenes, para eso deberíamos retardar el envejecimiento, para así llevar una vida plena hasta los 80 o más.
Referencia:
Sahin, E., Colla, S., Liesa, M., Moslehi, J., Müller, F., Guo, M., Cooper, M., Kotton, D., Fabian, A., Walkey, C., Maser, R., Tonon, G., Foerster, F., Xiong, R., Wang, Y., Shukla, S., Jaskelioff, M., Martin, E., Heffernan, T., Protopopov, A., Ivanova, E., Mahoney, J., Kost-Alimova, M., Perry, S., Bronson, R., Liao, R., Mulligan, R., Shirihai, O., Chin, L., & DePinho, R. (2011). Telomere dysfunction induces metabolic and mitochondrial compromise Nature DOI: 10.1038/nature09787
Kelly, D. (2011). Cell biology: Ageing theories unified Nature DOI: 10.1038/nature09896






 Algunas teorías creen que cuando se formaron los fosfolípidos, o los primeros ácidos grasos, estos se juntaron y formaron estructuras
Algunas teorías creen que cuando se formaron los fosfolípidos, o los primeros ácidos grasos, estos se juntaron y formaron estructuras 



