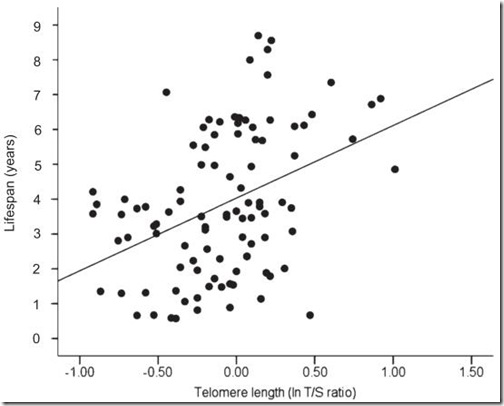
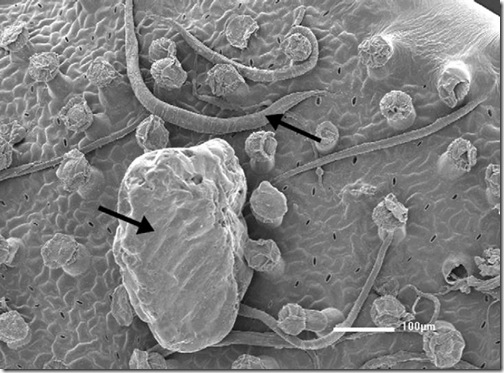
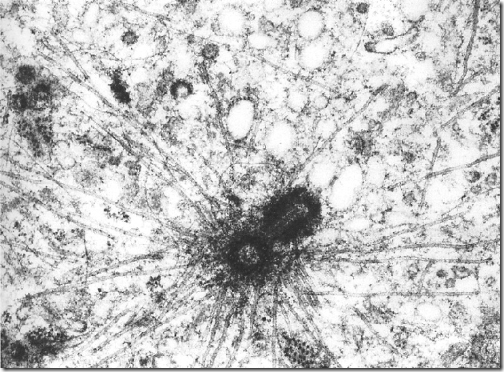
La presencia o ausencia de indol en la feromona atrae o repele a los que son hermafroditas.
El nemátodo Caenorhabditis elegans es un buen modelo biológico para hacer estudios básicos sobre el comportamiento social, especialmente los que involucran la alimentación, la densidad poblacional, el apareamiento y la agregación. Estudios recientes han demostrado que un grupo de moléculas, conocidas como los ascarósidos, regulan diversos aspectos del comportamiento del diminuto animal.
Los ascarósidos ascr#1, ascr#2 y ascr#3 han sido identificados como los principales componentes de las feromonas que promueven la agregación de los machos. Sin embargo, los hermafroditas, quienes conforman la mayor parte de la población de los nemátodos, son insensibles a bajas concentraciones de estos ascarósidos y son repelidos a concentraciones usadas por los machos para reunirse.
Con el fin de identificar las señales químicas que promueven la agregación de los hermafroditas, un grupo de investigadores, liderados por el bioquímico Frank Schroeder de la Universidad de Cornell, analizaron los compuestos generados tanto en nemátodos silvestres como en mutantes para la proteína DAF-22, la cual es requerida en la biosíntesis de las feromonas de estas especies. Según reportaron el 10 de Enero en PLoS BIology, los indol-ascarósidos ejercen un potente efecto atractivo en los nemátodos hermafroditas, sugiriendo que estos pequeños gusanos tienen un lenguaje químico de comportamiento social bastante complejo.
Schroeder y sus colaboradores identificaron muchos compuestos unidos al indol —una molécula derivada del tritófano— en los gusanos silvestres, pero no en los mutantes para el gen daf-22; siendo el indol unido al ascr#3 (icas#3), el más abundante de todos estos compuestos [Figura de portada].
Entonces, como había una diferencia sustancial (químicamente hablando) entre el ascr#3 y el icas#3, los investigadores quisieron saber si estas moléculas generaban respuestas diferentes en el nemátodo. Para ello, sintetizaron químicamente el icas#3 y otros ascarósidos indolados más como el icas#1 e icas#9, y los pusieron en el medio de cultivo. A concentraciones elevadas, estos indol-ascarósidos promovían la agregación de machos y hermafroditas. Pero cuando se repitió el experimento a bajas concentraciones, los icas#3 e icas#9 sólo promovieron la agregación de los hermafroditas mas no de los machos. El efecto de estos indol-ascarósidos es potente.
Los investigadores también analizaron que neuronas eran activadas por esta novedosa feromona. Lo que encontraron fue que los hermafroditas respondían al icas#3 aún en ausencia de las neuronas RMG, las cuales detectan las condiciones del entorno, integran la información y mandan la señal a los músculos para promover la agregación; por ejemplo, cuando hay abundancia de alimento. Sin embargo, cuando las neuronas ASK estaban ausentes, la respuesta no se daba.
Estos resultados contradicen lo encontrado en estudios previos. En estos se decía que los ascarósidos actúan tanto a nivel de las neuronas RMG como las ASK. Sin embargo, como podemos ver en el párrafo anterior, cada respuesta es mediada por un circuito neuronal diferente: los ascarósidos (feromonas que repelen a los hermafroditas) son reconocidos por las neuronas RMG y los indol-ascarósidos (feromonas que promueven la reunión de los hermafroditas) son reconocidos por las neuronas ASK.
Estos dos tipos de feromonas actúan en una coordinación magistral. Cuando está presente la icas#3, aún en presencia de ascr#3, los hermafroditas se agregan, siempre y cuando las concentraciones sean bajas. Sin embargo, cuando se incrementa la concentración de ascr#3, las hermafroditas se ven repelidas, aún icas#3 esté presente en el medio.
De estas observaciones podemos concluir que, cuando las densidades poblacionales son elevadas, la concentración de ascr#3 aumenta gradualmente. Esto provoca que los hermafroditas, quienes conforman la mayor parte de la población, se vean repelidas, reduciendo así la competencia por el alimento.
Además, los investigadores sugieren que los nemátodos tienen la capacidad de ajustar los niveles de feromonas liberados. Pueden promover la agregación liberando la versión no-indolada y así reunirse para reproducirse, o pueden reprimirla, liberando la versión indolada para evitar la competencia por el alimento. Estos resultados demuestran el alto nivel de complejidad en la comunicación social de los nemátodos.
Referencias:
Srinivasan, J., von Reuss, S., Bose, N., Zaslaver, A., Mahanti, P., Ho, M., O'Doherty, O., Edison, A., Sternberg, P., & Schroeder, F. (2012). A Modular Library of Small Molecule Signals Regulates Social Behaviors in Caenorhabditis elegans PLoS Biology, 10 (1) DOI: 10.1371/journal.pbio.1001237
Weaver J (2012) New Signaling Chemicals Spur Worms to Seek Company. PLoS Biol 10(1): e1001240. doi:10.1371/journal.pbio.1001240