Este año, el británico Sir John B. Gurdon y el japonés Shinya Yamanaka fueron galardonados con el Premio Nobel de Medicina o Fisiología por descubrir que las células maduras y especializadas pueden revertir su capacidad de diferenciarse en cualquier otro tipo célula, revolucionando nuestro entendimiento sobre el desarrollo celular y ampliando las perspectivas de la medicina regenerativa.
| Sabías que… |
|
Madurez celular
Todo se inicia con la fertilización del óvulo por el espermatozoide formando el zigoto, el cual empieza a dividirse rápidamente. Estas primeras células tienen la capacidad de convertirse en un embrión completo, incluyendo la placenta. Por esta razón son conocidas como células totipotentes.
La siguiente etapa del desarrollo es el blastocisto, en el cual podemos encontrar dos capas de células diferentes: la masa celular interna que formará al embrión propiamente dicho y la masa celular externa o trofoblasto que formará los tejidos externos que darán soporte y proveerán de nutrientes al embrión.
La masa celular interna tiene la capacidad de convertirse en cualquier tipo de tejido (cardiaco, nervioso, óseo, incluso germinal), pero ya no formará la placenta. En otras palabras, son células pluripotentes y son conocidas como células madre embrionarias.
A partir de aquí, las células empiezan a adquirir funciones cada vez más especializadas, diferenciándose unas de otras y formando todos los tejidos y órganos del cuerpo. Sin embargo, en algunas locaciones hay células que aún mantienen cierto grado de indiferenciación para poder regenerar células que se van perdiendo constantemente, tales como: las células que cubren nuestro cuerpo y nuestros intestinos (células epiteliales) o los glóbulos rojos, blancos y plaquetas que se forman en la médula ósea a partir de las células madre hematopoyéticas. Estas células son multipotentes.
Las células ya diferenciadas de un organismo son extremadamente estables. No se transforman en otros tipos de células ni vuelven a un estado de pluripotencia de manera natural. Cada tipo de célula cuenta con un patrón específico de proteínas que son las responsables de su función. El entorno bioquímico que envuelve el núcleo celular “enciende” y “apaga” determinados genes de manera eficiente.
Todo esto hizo pensar a muchos investigadores del siglo XX que ya no se podía hacer nada una vez las células se diferenciaban. Sin embargo, habían otros que estaban buscando la forma de romper esta barrera.
Transferencia de núcleos
Por los años 1950 había la teoría que si un núcleo de una célula diferenciada era transferido al citoplasma de un huevo fertilizado —al que le habían quitado su núcleo previamente— éste podría generar un nuevo embrión.
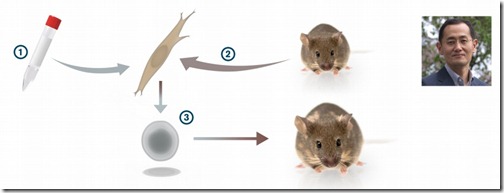
Fue así que en 1955, los investigadores norteamericanos Robert Briggs y Thomas King probaron esta hipótesis en la rana leopardo (Rana pipiens). Primero transfirieron el núcleo de un embrión previamente formado a un huevo fertilizado sin núcleo y observaron que esta nueva célula logró desarrollar un renacuajo. Sin embargo, cuando repitieron el experimento tomando el núcleo de una célula de renacuajo, ya no lo consiguieron, concluyendo que los núcleos de células diferenciadas sufrían cambios irreversibles durante su desarrollo.
El experimento fue repetido por el británico John Gurdon con algunas variaciones y aplicando una técnica que le permitía distinguir a las células generadas a partir de un núcleo trasplantado. Gurdon transfirió los núcleos de células epiteliales del intestino de un renacuajo de la rana africana (Xenopus laevis) a huevos fertilizados a los cuales había eliminado el núcleo aplicando radiación UV, obteniendo así muchos renacuajos vivitos y coleando. Su trabajo fue finalmente publicado en 1962 y en este concluía que los núcleos de las células somáticas diferenciadas tenían la capacidad de revertir la pluripotencia.
Como era de esperarse, la comunidad científica se mostró reacia a aceptar estas afirmaciones. Gurdon no se quedó con los brazos cruzados y realizó más experimentos para dar mayor soporte a sus afirmaciones. En 1966 logró formar renacuajos a partir de núcleos de células diferenciadas de ranas adultas y en 1970 logró desarrollar una rana adulta a partir de núcleos de células embrionarias.
Fue así que Gurdon cambió el paradigma de la biología del desarrollo y por primera vez demostró que los núcleos de células somáticas diferenciadas tienen la capacidad de generar todos los tipos de tejidos de un organismo si son puestos dentro del citoplasma de un huevo fertilizado.
Con los años, la transferencia de núcleos fue perfeccionada y gracias a esta técnica, en 1997 se logró clonar por primera vez a un mamífero —la famosa oveja Dolly— a partir del núcleo de una célula mamaria.
Reprogramando las células
Pero, ¿qué diferencia bioquímica había entre el citoplasma de un huevo fertilizado y de una célula diferenciada que le permitía al núcleo reactivar la pluripotencia? El japonés Shinya Yamanaka empezó a buscar la respuesta a principios del siglo XXI con el fin de identificar las moléculas necesarias para reactivar la pluripotencia directamente en una célula diferenciada.
Yamanaka y su equipo estudiaron a fondo los factores involucrados en el mantenimiento de la pluripotencia de las células madre embrionarias, cultivadas y caracterizadas por primera vez por Martin Evans (ganador del Nobel de Fisiología en el 2007); logrando caracterizar aproximadamente 24 factores de transcripción candidatos.
Los Factores de Transcripción (FT) son un grupo de proteínas encargadas de modular la expresión de una gran variedad de genes. En otras palabras, son los interruptores genéticos. Están presentes en casi todos los sistemas bioquímicos de las células eucariotas, creando “programas regulatorios” que definen los diversos estados de desarrollo de un organismo así como su adaptación a una gran variedad de ambientes diferentes.
Los genes que codifican estos 24 FT candidatos fueron introducidos uno por uno dentro del genoma del células diferenciadas de ratones para ver cuáles eran los responsables de revertir el estado pluripotentes de estas células diferenciadas. En el 2006 descubrieron que la combinación de cuatro de ellos: Myc, Oct3/4, Sox2 y Klf4 fue suficiente para convertir a las células embrionarias del fibroblasto de ratones en células indiferenciadas con la capacidad de generar cualquier tipo de célula. A estas células las llamaron células madre pluripotente inducidas (iPSC).
Un año después, el equipo de Yamanaka y del estadounidense James Thomson lograron generar las primeras iPSC humanas usando la combinación de estos cuatro FT (aunque el equipo de Thomson usó los FT Lin28 y Nanog en vez de Myc y Kfl4).
Perspectivas a futuro
En nuestros días, el uso de las células madre embrionarias ha generado opiniones encontradas. Éstas células sólo pueden conseguirse destruyendo un embrión, que para ciertos sectores de la población, un atentado contra la vida humana. Mejorar las técnicas que permitan desarrollar células madre a partir de células adultas de manera eficiente es algo en lo que vienen trabajando los científicos hoy en día.
El descubrimiento de nuevos FT involucrados con la formación de un determinado tipo de célula o tejido está permitiendo saltarse el paso de convertir una célula diferenciada en otra completamente diferente sin la necesidad de convertirla antes en una iPSC [Aquí un ejemplo y otro ejemplo].
Por otro lado, las iPSC tienen el potencial de reemplazar las células dañadas que pueden encontrarse en pacientes con enfermedades degenerativas como el Parkinson o diabetes tipo 1, o para regenerar los tejidos dañados, o para curar desórdenes fisiológicos. Sin embargo, se ha observado que el desarrollo de las iPSC muchas veces no pueden ser controlada y terminan por dividirse de manera descontrolada, que podrían generar tumores si son insertadas dentro de una persona.
Las iPSC también pueden usarse para recrear distintas enfermedades humanas en el laboratorio, con la finalidad de probar una gran cantidad de fármacos y drogas de manera rápida y económica.
Finalmente, una de las aplicaciones más importantes de esta tecnología es que nos permite estudiar a fondo el desarrollo celular, identificar y caracterizar los factores involucrados en los distintos procesos celulares, analizar el efecto de diferentes sustancias biológicas en su fisiología, entre otras cosas.
Sin dudas, el descubrimiento hecho por estos dos excepcionales personajes ha abierto todo un campo de estudio dentro de las ciencias naturales. Felicitaciones por ello.
Referencia:
MLA style: "The 2012 Nobel Prize in Physiology or Medicine - Advanced Information". Nobelprize.org. 8 Oct 2012 http://www.nobelprize.org/nobel_prizes/medicine/laureates/2012/advanced.html