Todas las células tienen proteínas que reconocen y se unen a secuencias específicas del ADN. Estas proteínas pueden actuar como efectores o reguladores, según promuevan o repriman la expresión de un determinado gen. Entre las más comunes tenemos los dedos de zinc (muchos factores de transcripción de mamíferos) hélice-giro-hélice (en todos los procariotas y eucariotas) y el cierre de leucina (abundante en el genoma humano).
Se ha descubierto en el género Xanthomonas, un patógeno importante de las plantas -principalmente del arroz-, un nuevo motivo de unión al ADN que juega un papel primordial en la activación de genes que promueven la virulencia del microorganismo. Este factor de transcripción denominado TAL (transcription activator-like) es traducido en las bacterias y depositado en las células de las plantas vía un sistema de secreción tipo III.
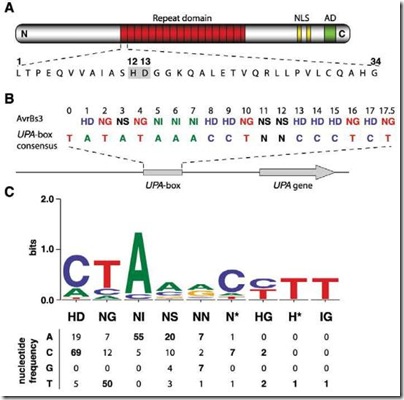
El dominio central de TAL consta de una señal de localización nuclear (NLS), un dominio de activación transcripcional acídico y, la parte más importante es un dominio que consta de un número variable de repeticiones en tandem de 34 aminoácidos, altamente conservados, a excepción de dos regiones adyacentes muy variables correspondientes a los aminoácidos 12 y 13. Este par de aminoácidos son los que dan la especificidad a la proteína TAL usando, para esto, un simple código de correspondencia. Al hacer un alineamiento entre los aminoácidos 12 y 13 con las secuencias de ADN se observó que este código binario tiene un significado biológico; cada par corresponde a un determinado nucleótido. Por ejemplo, AvrBs3 es una proteína TAL, la cual consta de 17.5 repeticiones (la última repetición solo conserva los 20 primeros aminoácidos, por eso se toma como la mitad), la cual reconoce una región específica del genoma denominada caja UPA (UPA box) de unos 18 ó 19 pares de base (pb), dándose de esta manera una correspondencia 1 a 1. Cuando a esta proteína se le quitó 4 repeticiones (de la 11 a la 14) ya no reconoció la caja UPA pero sí otra región más corta, demostrándose así que la especificidad de las proteínas TAL. Otra característica importante es la timina (T) conservada en el terminal 5’, a la cual se le denomina repetición 0. Se crearon cajas con A, C y G en la posición 0 y ninguna de ellas fue expresada, confirmándose la importancia de T en la posición 0.
Si bien linealmente se ve todo muy lindo y bastante obvio, no se ha podido determinar –o por lo menos imaginar- cual es el efecto o como ayuda la estructura tridimensional de TAL para que los aminoácidos 12 y 13 reconozcan las secuencias de ADN específicas.
Usando el código de correspondencia de las repeticiones con las secuencias de ADN, hallado con 8 proteínas TAL, se trató de predecir las secuencias diana de otros efectores TAL como Xa27, PthXo6, PthXo1, PthXo7 inspeccionando sus regiones promotoras, pudiéndose establecer correctamente este código de correspondencia.
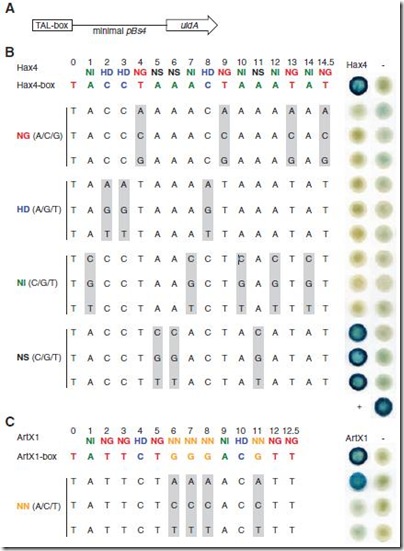
Para validar estos experimentos se crearon casetes usando a las cajas Hax2 (tiene repeticiones de 35 aminoácidos en vez de 34), Hax3 y Hax4, acoplados a al gen reportero GUS (uidA) (Fig2B) el cual evidencia la expresión de un gen mediante una coloración azul. Se uso como control positivo al promotor 35S del virus del mosaico de la coliflor acoplado al reportero GUS. Se observó que el reportero acoplado a una determinada caja sólo se expresaba en presencia de su proteína TAL (Fig2C).
Si bien ya se tenía un código de correspondencia de cada repetición con cada nucleótido en la secuencia diana, se quiso observar si esta correspondencia era específica. Se crearon cajas Hax4 cambiando cada nucleótido por los otros tres y acoplándolos al reportero GUS para evidenciar la expresión, para luego introducirlo en el genoma vía coinfección con Agrobacterium. Se observó que NG, HD y NI reconocen T, C y A respectivamente, mientras que NS aceptaba cualquiera de los 4 nucleótidos (Fig 3B). Muchas proteínas TAL poseen repeticiones NN pero no se sabía a que nucleótido correspondía; entonces, usando la caja ArtX1 artificial de 12.5 repeticiones, se observó que NN reconocía tanto A como G (Fig 3C)
Luego, querían probar si podían predecir la secuencia diana de una determinada proteína TAL, para esto analizaron el genoma de Arabidopsis thaliana y buscaron secuencias diana para Hax2 en base al código de correspondencia previamente determinado. Usando herramientas bioinformáticas se encontró que el promotor PAP1 tenía una secuencia de correspondencia de Hax2, y al hacer el experimento se observó que expresaba el gen para el factor de transcripción MYO, de esta manera se demostró que si se podían predecir las secuencias diana para las proteías TAL.
Finalmente se determinó cual es el número mínimo de repeticiones necesarias para que una proteína TAL funcione. Se creó una proteína TAL artificial, a la cual se le puso repeticiones HD (de 0.5 a 15.5 repeticiones) y también se creó cajas ArtHD con repeticiones de nucleótidos C (de 1 a 16) acoplado al reportero GUS y se observó que a partir de 6.5 repeticiones la proteína TAL empezaba a funcionar, pero con 10.5 repeticiones era mayor la expresión del gen.
Que aplicaciones en la biotecnología podrían tener? Imagínense crear factores de transcipción con nuevas especificidades para reconocer determinadas regiones del genoma. Se crearon 7 ArtX artificiales y todos respondieron únicamente a sus proteínas TAL. De esta manera, si quisiéramos introducir un gen en un organismo, lo podríamos hacer en el lugar exacto que nosotros queramos. Podríamos hacer cortes en los cromosomas en lugares específicos, entre otras aplicaciones.
Boch, J., Scholze, H., Schornack, S., Landgraf, A., Hahn, S., Kay, S., Lahaye, T., Nickstadt, A., & Bonas, U. (2009). Breaking the Code of DNA Binding Specificity of TAL-Type III Effectors Science, 326 (5959), 1509-1512 DOI: 10.1126/science.1178811







0 comentarios:
Publicar un comentario
Se respetuoso con tus comentarios y críticas. Cualquier comentario ofensivo será eliminado.