Cuanta gente que empieza a consumir cocaína, heroína o nicotina (o Terocal como yo) y nunca la pueden dejar, por más tratamientos e internamientos que tengan, ¿a qué se debe? La explicación es que, cuando se consume de manera constante una de estas drogas o cualquier otro estupefaciente, el cerebro se adapta a tenerla siempre en él y lo toma como si fuera “normal”, volviéndolo menos sensible a los efectos que tanto les gusta sentir, así que necesitan de más para alcanzar el efecto deseado. En este punto, la dosis que consume un drogadicto podría llevar a la muerte por sobredosis a una persona normal que por primera vez prueba una de estas drogas.
Pero, ¿cuales son las bases bioquímicas y moleculares que dan paso a la adicción? Hollander et al. pudo haber encontrado la respuesta a los cambios neuronales que terminan en la tolerancia a las drogas, debido a su consumo prolongado, estudiando los cerebros de ratas”coqueras”. Primero, debemos saber que, drogas como la cocaína, atacan y alteran los sistemas de recompensa del cerebro. Y, ¿quienes están detrás de esto? Las responsables son unas pequeñas cadenas de ARN de 21 a 23 nucleótidos de largo llamado microARNs (miARN).
Para recordar: Aquí un post que publiqué hace un par de años respecto al tema.
Como pueden ver, los miARNs regula la expresión de los genes a nivel post-transcripcional (después de ser transcritos de ADN a ARN mensajero). El genoma humano codifica para aproximadamente 1000 miARNs, y si están en el cerebro, de hecho regularán la expresión de genes que controlan su estructura y función, y tal vez puedan estar relacionados con la adicción.
 Hollander et al. encontraron que la cocaína aumentaba los niveles de un miARN llamado miR-212 en una región del cerebro llamada estriado dorsal. Pero, ¿de que manera estaba relacionado con la adicción? Para esto tomaron unas ratas drogadictas a las cuales les insertaron un vector cargando un miR-212. La característica de este vector era que el miR-212 se sobre-expresaría aumentando sus niveles en el cerebro. Los investigadores observaron que las ratas – que antes de insertarles el vector eran más coqueras que Maradona – disminuyeron su ansiedad por la cocaína. Para confirmar este experimento, diseñaron otro provocando un efecto contrario. Para esto silenciaron (“apagaron”) el miR-212 y observaron que las ratas drogadictas aumentaron su ansiedad y consumo de cocaína.
Hollander et al. encontraron que la cocaína aumentaba los niveles de un miARN llamado miR-212 en una región del cerebro llamada estriado dorsal. Pero, ¿de que manera estaba relacionado con la adicción? Para esto tomaron unas ratas drogadictas a las cuales les insertaron un vector cargando un miR-212. La característica de este vector era que el miR-212 se sobre-expresaría aumentando sus niveles en el cerebro. Los investigadores observaron que las ratas – que antes de insertarles el vector eran más coqueras que Maradona – disminuyeron su ansiedad por la cocaína. Para confirmar este experimento, diseñaron otro provocando un efecto contrario. Para esto silenciaron (“apagaron”) el miR-212 y observaron que las ratas drogadictas aumentaron su ansiedad y consumo de cocaína.
De estos dos primeros experimentos sacaron la conclusión que miR-212 tenía que ver, de cierta manera, con el control del sistema compensatorio del cerebro. Cuando se sobre-expresaban reducían la ansiedad y consumo de cocaína de las ratas drogadictas, mientras que en su ausencia, el consumo de cocaína aumentaba. La deficiencia de miR-212 las volvía más vulnerables a la drogadicción.
Ahora tenemos una nueva pregunta por responder. ¿El miR-212 a que nivel actúa, sobre que proteína tiene un efecto regulador, con que proteínas puede interactuar? Hollander encontró que la cocaína inducía la expresión de CREB, un importante señalizador del sistema compensatorio del cerebro, el cual reduce las propiedades motivacionales de la droga. Además, encontró que miR-212 respondía positivamente a la expresión de CREB, pero CREB sólo era funcional si estaba fosforilada. Así que aquí falta algo que fosforile a CREB. La respuesta fue que miR-212 inducía – de alguna forma – la producción de AMPc, la cual acetilaba a TORC – miembro de una familia de proteínas que actúa como co-activador de CREB – fosforilando a CREB.
El AMPc es una molécula envuelta en muchos procesos de activación y desactivación de la expresión de muchas proteínas, sin embargo, no está presente siempre, debe haber un desequilibrio en alguna vía metabólica para que se manifieste. Hollander encontró que miR-212 inhibía muchos genes que codificaban para represores de Raf1 la cual es la responsable de la acumulación del AMPc. En otras palabras, Raf1 sintetiza AMPc pero siempre se encuentra inactiva debido a que sus represores, como SPRED1, siempre se encuentran activos. miR-212 regula la expresión de SPRED1 (y otros inhibidores de Raf1) y el AMPc cíclico se produce. Esto se demostró usando un gen SPRED1 resistente a miR-212, el cual inhibía a Raf1 y no se producía AMPc.
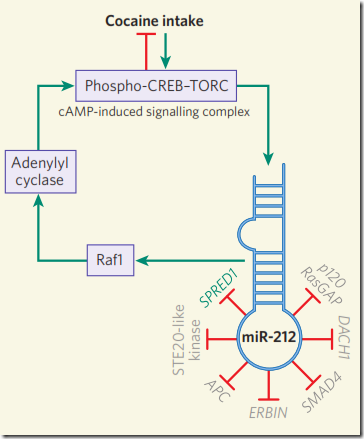
Entonces, para no marearlos más, la sobre-expresión de miR-212 se da debido al consumo excesivo de cocaína. miR-212 inhibe a SPRED1 y Raf1 queda libre para hacer lo que le da la gana, en este caso, producir grandes cantidades de AMPc. El AMPc activa – mediante acetilación – a TORC, el cual es un co-activador de CREB. CREB está relacionado directamente con el sistema de recompensa del cerebro, cuanto más CREB hay, el cerebro siente menos recompensa ante el consumo de cocaína y necesita de más para sentir placer. Esto se convierte en un círculo vicioso, porque, nuestro cerebro siente cada vez menos placer y necesita de más droga, o también, siente que una determinada cantidad de droga en el cerebro es normal y su ausencia provocará un desequilibrio y una necesidad por suplir esa carencia.
Ojo, cabe resaltar que este estudio fue hecho en ratas, pero, ha dado grandes respuestas y posibles mecanismos para tratar la adicción a la cocaína y otras drogas. Si se pudiera controlar la expresión de miR-212, se podría controlar la ansiedad, o por lo menos, mantenerla en ciertos valores que no lleven al uso excesivo de la misma.
Referencia:
 Hollander, J., Im, H., Amelio, A., Kocerha, J., Bali, P., Lu, Q., Willoughby, D., Wahlestedt, C., Conkright, M., & Kenny, P. (2010). Striatal microRNA controls cocaine intake through CREB signalling Nature, 466 (7303), 197-202 DOI: 10.1038/nature09202
Hollander, J., Im, H., Amelio, A., Kocerha, J., Bali, P., Lu, Q., Willoughby, D., Wahlestedt, C., Conkright, M., & Kenny, P. (2010). Striatal microRNA controls cocaine intake through CREB signalling Nature, 466 (7303), 197-202 DOI: 10.1038/nature09202










