
Todas las proteínas presentes en cualquier organismo vivo, desde una gigantesca ballena azul hasta una diminuta bacteria o incluso un virus, están formadas por aminoácidos, unas pequeñas moléculas que se “cogen de la mano” unas con otras para formar cadenas muy largas —la titina puede tener más de 30.000— que se doblan, enroscan y pliegan de diferentes maneras para cumplir con una función específica.
La función de una proteína depende de su forma y esta, a su vez, de la secuencia de su cadena de aminoácidos y las condiciones del entorno: el pH, la presencia de elementos metálicos o vitaminas llamados cofactores o las chaperonas que colaboran con el plegamiento de las proteínas recién formadas. Sin embargo, el número de aminoácidos diferentes que pueden formar parte de alguna proteína es sólo 20 (aunque existen casos exepcionales en los que ciertos organismos pueden usar otros aminoácidos).
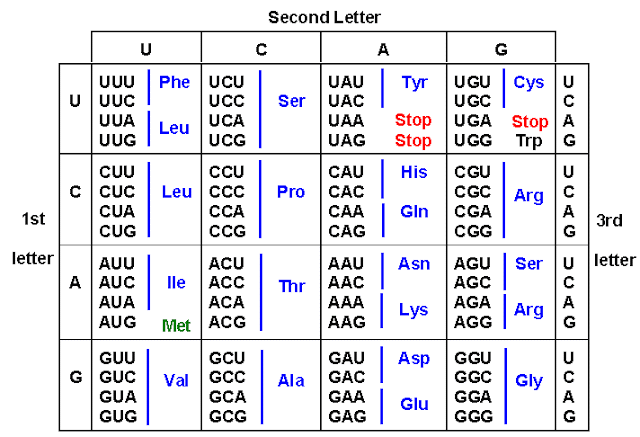
La secuencia de aminoácidos de una proteína está determinada por la secuencia de nucleótidos de un gen. Como existen sólo cuatro nucleótidos diferentes, estos se deben agrupan en tripletes para codificar cada uno de los 20 aminoácidos. La cantidad combinaciones diferentes que se puede obtener es de 64 (4 x 4 x 4), por lo tanto habrán aminoácidos que serán codificados por más de un triplete obedeciendo el siguiente código:
Como pueden ver, existen tres tripletes que no codifican aminoácido alguno, porque los ARN de transferencia (ARNt) que se unen de manera complementaria a ellos, no cargan ningún aminoácido, terminando así la reacción de síntesis de proteínas en los ribosomas. A estos se les llama codones de terminación.
Durante los últimos años, los científicos vienen trabajando en la expansión del código genético, es decir, ampliar el límite natural de 20 aminoácidos con la finalidad de insertar a las proteínas otros aminoácidos no-naturales para modificar su estructura y función, incluso lograr producir enzimas activadas por la luz a través de la inserción de aminoácido fotosensibles.
Durante los últimos años, los científicos vienen trabajando en la expansión del código genético, es decir, ampliar el límite natural de 20 aminoácidos con la finalidad de insertar a las proteínas otros aminoácidos no-naturales para modificar su estructura y función, incluso lograr producir enzimas activadas por la luz a través de la inserción de aminoácido fotosensibles.
La estrategia para lograr insertar un aminoácido no-natural en una proteína es mutando la enzima responsable de dotar de aminoácidos a los ARNt, también conocido como aminoacil-ARNt sintetasa, para que éste sea capaz de ligar un aminoácido cualquiera al ARNt complementario a un codón de terminación. El más usado es el aminoacil-ARNt sintetasa/ARNt(CUA), el cual responde al codón de terminación UAG.
Mediante esta estrategia se ha logrado incorporar aminoácidos no-naturales en bacterias, levaduras y cultivos celulares de mamíferos. Sin embargo, lograr esto en un animal superior ha sido una tarea sumamente complicada. El año pasado se dio un gran avance cuando dos investigadores británicos, Sebastian Greiss y Jason Chin, reportaron haber introducido un aminoácido nuevo en un animal, el nemátodo Caenorhabditis elegans.
Ahora, Greiss & Chin han dado un paso más. Según un artículo publicado esta semana en Nature Chemical Biology estos investigadores han incorporado de manera específica tres aminoácidos no-naturales en proteínas de moscas de la fruta (Drosophila melanogaster) en diferentes etapas de su desarrollo. Gracias a este experimento podemos contar con un método preciso de incorporación de nuevos aminoácidos en un modelo biológico ampliamente estudiado para explorar nuevas funciones de proteínas animales.
Básicamente el trabajo consistió en tomar los genes que codifican la enzima responsable de unir la pirrolisina (un aminoácido no-natural) al ARNt complementario al codón de terminación UAG de la bacteria Methanosarcina mazei e introducirla —mediante ingeniería genética— en embriones de D. melanogaster. Para activar estos genes en la mosca de la fruta, los pusieron bajo el control de un interruptor activado por iones de cobre, y para poder observar si la reacción fue exitosa (los tres aminoácidos no-naturales usados en el experimento lograron ser incorporados) se usaron moléculas fluorescentes unidas a través del codón TAG (UAG cuando pasan a ARN mensajero), tal como se muestra en el siguiente esquema:
Entonces, si los embriones expresan tanto la proteína fluorescente verde como la roja, indicaría que la reacción de traducción no se detuvo, el codón de terminación fue leído y uno de los aminoácidos no-naturales fue incorporado. Los resultados obtenidos al "alimentar" los embriones con iones de cobre y uno de los tres aminoácidos no-naturales que porbaron fueron los esperados. Además se confirmaron los experimentos usando otras construcciones genéticas basadas en la luciferasa (una enzima que cataliza una reacción de fluorescencia).
Las moscas adultas modificadas que fueron alimentadas con estos aminoácidos no-naturales lograron incorporarlos a sus proteínas en tejidos específicos, por ejemplo: los ovarios. Según el promotor usado, la incorporación de estos aminoácidos podría darse en otros tejidos del cuerpo de las moscas. Por otro lado, su capacidad reproductiva no se vio afectada ya que lograron cruzarse y generar embriones con la misma capacidad de usar estos aminoácidos.
Afrontar los desafíos de lograr una expansión del código genético en animales más complejos permitirá la extensión de las estrategias empleadas en estudio de los procesos biológicos, incluyendo el desarrollo embrionario, la morfogénesis de tejidos, la formación de los tumores y la plasticidad neuronal, a nivel del organismo.
Referencia:






0 comentarios:
Publicar un comentario
Se respetuoso con tus comentarios y críticas. Cualquier comentario ofensivo será eliminado.