Referencia:
The FASEB Journal. 2010. 24:2928-2937. DOI: 10.1096/fj.09-153130

Las mutaciones son la principal fuerza evolutiva de los seres vivos, son cambios en la secuencia de un gen a través de inserciones de nuevas secuencias, deleciones de uno o más nucleótidos o cambio de un nucleótido por otro (transiciones o transversiones), que provocan que la proteína codificada por el gen pierda su función, su especificidad o adquiera una nueva característica que le permita tener una ventaja en su supervivencia.
Sin embargo, la única forma de ver las mutaciones es a través de los fenotipos, o sea, la forma final de un determinado organismo. Por ejemplo: una mutación en un gen hace que los ojos de la mosca de la fruta sean blancos en vez de rojo. Los ojos de la mosca los podemos ver, y si son blancos sabemos que es debido a una mutación, pero no podemos ver directamente la mutación (a menos que secuenciemos el gen) y mucho menos el momento preciso en que se da esta mutación. Además, existen muchas mutaciones que no expresan un fenotipo característico. Hay mutaciones que no cambian la función de la proteína porque pueden cambiar un nucleótido por otro pero codificar para el mismo aminoácido o pueden codificar para otro aminoácido con características similares al anterior o aminoácidos que no forman parte del sitio activo de la enzima.
Además, las mutaciones son tan raras – en mamíferos es de 1 en 2.2x109 nucleótidos – que sería imposible ubicarlos en el preciso momento en que están ocurriendo durante la replicación del ADN. Pero, ¿por qué es tan baja la tasa de mutación? Las mutaciones se dan de manera más frecuente de lo que pensamos; pero, la evolución ha permitido el desarrollo una maquinaria de reparación de errores: mismatch repair machinery (MutS, MutL, MutH) y el proofreading de las enzimas polimerasas. Entonces, si queremos ver las mutaciones en vivo y en directo, por qué no marcamos una de las proteínas de estas maquinarias y le hacemos un seguimiento, de seguro que nos llevarán a los puntos donde se están dando las mutaciones!.
Esto fue lo que hizo Marina Elez et al. de la Universidad Paris Descartes y lo publicó el día de ayer en la revista Current Biology. Marina marcó con una molécula fluorescente a un derivado funcional de MutL – proteína clave en la maquinaria de reparación de errores. La proteína MutL se une alrededor de las mutaciones que no pueden ser reparadas formando agregados proteicos (“clusters”). Gracias a que la proteína estaba marcada con una molécula fluorescente, se podía ubicar rápidamente donde estaban las mutaciones, simplemente por el brillo que emitían. De esta manera pudieron ver en vivo y en directo las mutaciones; y además, pudieron cuantificar la cantidad de mutaciones que se daban en el ADN ya que era directamente proporcional a la intensidad de fluorescencia emitida. De esta manera pudieron corroborar la tasa de mutación global de E. coli el cual coincidía con los valores estimados previamente.
Si bien la técnica calcula la tasa de mutación de una célula individual, al repetir en experimento en varias células observaron que las tasas de mutación seguían una distribución de Poisson, lo que sugiere que todas las células en la población tienen casi la misma tasa de mutación. Esta técnica será muy usada para estimar de manera más exacta las tasas de mutaciones, no sólo en otras bacterias, sino también en organismos eucariotas, de manera independiente a su fenotipo. También ayudarán mucho en el diagnóstico temprano de cánceres y desarrollo de tumores, los cuales se caracterizan por una alta tasa de mutación en sus genomas.
Referencia:
Current Biology, DOI: 10.1016/j.cub.2010.06.071
Hace un par de semanas se hizo una grave denuncia a British Petroleum por falsear y modificar fotos para mostrar que el problema del Golfo de México estaba siendo resuelto y que los daños eran menos graves de lo que se pensaban. La explicación de BP para esta falta de ética fue que uno de los miembros de su personal quería demostrar sus habilidades con el Photoshop y por error cayó en las manos del webmaster quien publicó las fotos en la página oficial de la empresa.
En respuesta a esto, un montón de gente en el mundo se puso a hacer lo mismo, modificar las fotos en Photoshop pero de una manera más imaginativa, para reirnos un poco y olvidar por un instante este grave desastre ecológico…
Los pilotos de BP tomándose un “break”
Con las manos en la masa! Miembros de BP captados en el preciso momento
que falseaban las fotos en GIMP.
El problema es más grave de lo que se creía.
Hacen todo menos controlar el desastre.
Para ver la galería completa visitar:
http://www.wired.com/wiredscience/2010/07/bp-reader-photoshop/
Sin dudas, los ojos, son una de las más grandes maravillas de la evolución, pero aún hay mucha controversia acerca del origen de los mismos. Los ojos son órganos extremadamente complejos, con características sumamente sofisticadas, y varían mucho en su funcionamiento entre una especie y otra, lo cual, abre la posibilidad a que el origen de éste órgano no haya seguido una sola línea evolutiva, sino varias paralelas.
En los metazoos, existe una familia de genes, Pax, que codifican para una serie de factores de transcripción involucrados en una gran variedad de procesos del desarrollo celular. Estos genes divergieron antes que los cnidarios y bilaterios se separaran y evolucionaran independientemente. Pax-6, uno de los miembros de esta familia, controla el desarrollo de los ojos en muchos bilaterios, y mutaciones en este gen causan graves defectos morfológicos y funcionales.
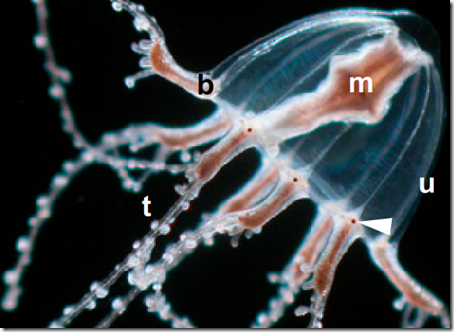
Los cnidarios son los animales más primitivos con ojos multicelulares; sin embargo, el gen Pax-6 no ha sido identificado. Entonces, ¿que gen es el responsable del desarrollo de los ojos en estos animales? Antes de todo, debemos tener conciencia de la importancia de encontrar un organismo con un mecanismo diferente de desarrollo de los ojos, porque nos ayudará a entender como se originaron y como han ido evolucionando a lo largo del tiempo.
Por ejemplo, en las medusas de la clase Cubozoa, es el gen Pax-B – un ortólogo del Pax-2/5/8 de los vertebrados – el responsable del desarrollo de los ojos. Se cree que Pax-6 divergió de Pax-B y fue reclutado por los miembros del grupo Bilateria. En la medusa Cladonema radiatum (Hidrozoa) se aislaron tres genes de la familia Pax: Pax-A, Pax-B y Pax-E. Estos genes fueron hallados mediante una PCR usando primers degenerados específicos para los dominios conservados de Pax. A diferencia de los cubozoos, en C. radiata, el gen Pax-B no tiene nada que ver con el desarrollo de los ojos, al igual que Pax-E. Los investigadores, liderados por el Dr. Hiroshi Suga de la Universidad de Basel, encontraron que el gen Pax-B se expresaba principalmente en el manubrio – el órgano digestivo-reproductivo de C. radiata – pero no lo hace en el bulbo de los tentáculos, que es donde están ubicados los ojos. Sin embargo, el gen Pax-A si se expresa en los tentáculos.
Y, ¿por qué se cree que Pax-6 se originó a partir de Pax-B? Lo que los científicos encontraron fue que al insertar el gen Pax-B del cubozoo en la mosca de la fruta (Drosophila melanogaster) este inducía el desarrollo de los ojos ectópicamente (fuera de su lugar normal). De la misma forma actuaba el gen Pax-A de C. radiata (crPax-A)
Estudios filogenéticos de los genes Pax-A, Pax-B y Pax-6, muestran que pertenecen a diferentes sub-familias, que divergieron antes de la separación entre los cnidarias y bilaterias. Además, gracias a que Pax-A y Pax-B (genes de los cnidarias) también pueden inducir el desarrollo de ojos en animales bilaterias como la mosca de la fruta, sugieren que estos tres genes tienen un origen evolutivo monofilético (un sólo ancestro común). Se cree que el origen de los otros genes Pax fue como producto de duplicaciones genéticas, que en las fases tempranas de la evolución de los animales, tenían funciones redundantes y a través del tiempo fueron especializándose, alguno de ellos llegando a controlar el desarrollo de los ojos.
Referencia:
Suga, H., Tschopp, P., Graziussi, D., Stierwald, M., Schmid, V., & Gehring, W. (2010). Flexibly deployed Pax genes in eye development at the early evolution of animals demonstrated by studies on a hydrozoan jellyfish Proceedings of the National Academy of Sciences DOI: 10.1073/pnas.1008389107
A cuantos no nos ha pasado que, cuando estamos extrayendo ADN o purificando alguna proteína o separando cultivos de células o bacterias, tenemos un número impar de tubos para centrifugar, o más que eso, tenemos un número primo como 5, 7, 11 ó 13 tubos para centrifugar, pero no sabemos como ubicarlos, así que primero centrifugamos una parte y luego otra, ¡qué pérdida de tiempo!".
En BiteSize Bio encontré una forma de hacerlo que nunca se me había ocurrido (¡que roche!). Primero, todos sabemos como centrifugar 3 tubos, las disponemos en forma triangular para lograr la estabilidad del rotor, tal como se muestra en la figura. Podemos marcar los pocillos con una resaltador o un marcador para ubicarlos más rápidamente… pero como hacemos para centrifugar 5 ó 7 ó más?… ya la vieron???
Es fácil, una vez ubicado los 3 tubos en forma triangular, colocamos otros dos en pocillos opuestos, otros dos en otros pocillos opuestos y así hasta completar cualquier número impar o primo. De hecho que se verá desordenado y pareciera que esta desproporcionado o desbalanceado y podría salir mal la centrifugación, pero NO!!! está bien, todos los tubos tienen su contraparte y el rotor está balanceado, así que pueden proceder a centrifugar sin miedo…
También BiteSize Bio nos muestra una forma de centrifugar tubos más chiquitos (de 0.2ml y 0.6ml) sin la necesidad de cambiar el rotor de 1.5 – 2ml. Para eso, cortamos la tapita del tubo Eppendorf de 1.5 ó 2ml y dentro de ella ubicamos el tubo de 0.6ml. Si queremos para tubos de 0.2ml, a parte de cortar la tapa del tubo de 1.5 ó 2ml, cortamos la tapita del tubo de 0.6ml que está dentro del más grande y ponemos nuestro tubito de PCR dentro de ellos.
Claro, que si vas a centrifugar algo con mucho valor como los productos de tu PCR para mandarlo a secuenciar, o fragmentos de ADN marcado con sondas radiactivas como el P-32 o proteínas marcadas con S-35, o vas a centrifugar a velocidades superiores a 10000rpm, es preferible mejor cambiar de rotor y evitar daños y perjuicios.
Hoy se publicó en Sciencexpress un estudio alentador para evitar el contagio de VIH en mujeres. Se trata del uso de un gel a base de 1% de Tenofovir – 9- [(R)-2phosphonomethoxy)propyl]adenine monohydrate (PMPA) – un análogo al nucleótido de adenosina con una actividad inhibitoria potente contra los retrovirus. Este compuesto ya ha sido probado como profiláctico en monos y actualmente es usado en el tratamiento del VIH (Viread®).
Este ensayo clínico a gran escala se llevó a cabo en África, donde el SIDA es una epidemia – el 70% de los infectados con VIH en el mundo son del África. Es aquí donde se han realizado una gran cantidad de ensayos clínicos para contrarrestar el contagio del VIH en la población sexualmente activa. Las estrategias probadas van desde la circuncisión masculina (~57% de efectividad) hasta una vacuna desarrollada por Tailandeses con un ~31% de efectividad. De las 39 estrategias probadas, sólo 5 han tenido resultados alentadores.
El estudio llamado CAPRISA 004, fue realizado entre mayo del 2007 y marzo del 2010. Se partió de una muestra inicial de 2160 mujeres sexualmente activas, entre 18 y 40 años, de las cuales quedaron finalmente 889 (611 de zonas rurales y 278 de zonas urbanas), las cuales fueron divididas en dos grupos al azar. Al primer grupo (n=445) les dieron el gel con Tenofovir y al segundo grupo (n=444) se les dio un gel placebo de características fisicoquímicas similares al tratamiento. El ~94% de los involucrados completaron el estudio. Además, a las mujeres participantes se les brindó condones, charlas de protección contra ETS y forma de uso del gel para que las condiciones del estudio sean las óptimas.
El tratamiento consistía en aplicarse el gel en la vagina 12 horas antes y 12 horas después de las relaciones sexuales, y cada mes se hacían los chequeos respectivos (VIH, embarazo, otras ETS). Las mujeres que cumplieron con el uso correcto del gel – dos aplicaciones por encuentro sexual – en más del 80% de las veces se les categorizó como de “alta adherencia”; las que usaban el gel de manera adecuada del 50 al 80% de las veces estaban en la categoría de “adherencia media” y las que lo usaban de manera correcta menos del 50% de las veces estaba en la categoría de “baja adherencia”. Esto se determinó en función de los geles devueltos y las relaciones sexuales tenidas durante el mes.
Los resultados obtenidos fueron que la incidencia de HIV en las mujeres que usaron el gel con Tenofovir fue 5.6/100 mujeres-año (número de mujeres infectadas por cada 100 mujeres por cada año) mientras que las mujeres que usaron el placebo la incidencia fue de 9.1/100 mujeres-año. La tasa de incidencia de VIH fue 50% menor en los primeros 12 meses y 40% menor a los 24 meses usando el gel con Tenofovir. Comparando a las mujeres con “alta adherencia”, la incidencia de las que usaron el gel con Tenofovir fue 54% menor al placebo; las de “adherencia media” y “baja adherencia”, la incidencia fue 38 y 28% menor que del placebo, respectivamente.
Finalmente, se estimó que la eficiencia del Tenofovir en la reducción de la infección por VIH fue del 39%. ¿Efectos secundarios? Si… pero no fue gran cosa…, al ~94% le dio al menos una vez una pequeña diarrea, nada grave, aunque se puede deber al gel porque a los que tomaron el placebo también tuvieron algunos casos de diarrea, aunque también se puede deber a otras causas… a quien no le ha da una diarrea una vez al año? Lo que se debe rescatar del estudio es que se debe concientizar más a los participantes para que sigan al pie de la letras las indicaciones dadas para que los resultados sean más alentadores, aumentar la “adherencia” del gel. También se deben estudiar aquellos casos que dieron positivos a pesar de usar el gel correctamente, esos aislamientos pueden ser la clave para encontrar a los virus resistentes y mejorar la efectividad del Tenofovir.
Referencia:
Karim, Q., Karim, S., Frohlich, J., Grobler, A., Baxter, C., Mansoor, L., Kharsany, A., Sibeko, S., Mlisana, K., Omar, Z., Gengiah, T., Maarschalk, S., Arulappan, N., Mlotshwa, M., Morris, L., Taylor, D., & , . (2010). Effectiveness and Safety of Tenofovir Gel, an Antiretroviral Microbicide, for the Prevention of HIV Infection in Women Science DOI: 10.1126/science.1193748
Existen muchos mecanismos que regulan la expresión genética, los cuales pueden darse a nivel transcripcional (promotores, operadores, terminadores), post-transripcional (formación de estructuras secundarias, ARN de interferencia, micro ARNs), traduccional (estructuras secundarias que evitan el paso del ribosoma) y post-traduccional (apoproteínas y chaperonas encargadas de dar la estructura final a la proteína).
Sin embargo, muchos de los ARNs que son transcritos a partir del ADN no llegan a codificar ningún tipo de proteína, y muchos de estos ARNs no codificantes están envueltos en mecanismos de regulación de la expresión genética, por ejemplo: los micro ARN (miARN), ARN de interferencia (ARNi), ARN pequeño de interferencia (siARN) y ARN asociados a piwi (piARN). Pero, aún existe un tipo de ARN misterioso cuya importancia aún se mantiene en discusión, este es el ARN largo no codificante (lncRNA).
Kondo et al. hicieron un descubrimiento en la mosca de la fruta (Drosophila melanogaster). Un ARN conocido como polished-rice (pri) tenía las características de un lncRNA, pero, codificaba para pequeños péptidos que controlaba la expresión de ciertos genes durante el desarrollo embrionario de D. melanogaster. Este gen pri ya había sido descubierto anteriormente en otros insectos donde también cumplía funciones similares. Lo que más llamó la atención de este gen pri es que era policistrónico, o sea, el gen transcribía un solo ARNm pero era traducido en varios pequeños péptidos de manera independiente, similar a lo que pasa en un operón bacteriano. pri codificaba cuatro o cinco pequeños péptidos de 11 a 34 aminoácidos, altamente conservados en los insectos.
Para investigar la función de pri, Kondo et al. usaron embriones de moscas de la fruta y estudiaron sus células epidérmicas, específicamente, unas especializadas en la producción de pequeñas protuberancias llamadas tricomas (parecido a los tricomas de las plantas). El gen responsable de la formación de los tricomas es el gen shavenbaby (svb), el cual codifica una proteína que actúa como factor de transcripción de genes relacionados con la formación de los tricomas. Cuando svb era mutado, los embriones eran incapaces de formar tricomas; y además, cuando pri era mutado, el embrión tampoco podía formar tricomas, pero la expresión de svb no se veía afectada. Uhm… para que no se pierdan… cuando pri y svb están presentes se forman los tricomas; cuando se mutaba svb pero no pri no se llegaban a formar los tricomas; y cuando se mutaba pri pero no svb, tampoco se formaban los tricomas, pero, la expresión de la proteína SVB no se veía afectada. Entonces, de esto se puede deducir que pri regula la expresión de svb a nivel post-traduccional, produciéndole algún tipo de modificación que lo vuelva activo, pero, ¿como es este mecanismo?
Primero, debemos saber que la proteína SVB tiene varios dominios, uno de ellos es un dominio que actúa como factor de transcripción. Kondo encontró que pri actúa como un interruptor temporal en la formación de los tricomas. Los pequeños péptidos codificados por pri tienen acción proteolítica, o sea, tienen la capacidad de cortar ciertos enlaces peptídicos. Los pequeños peptidos de pri cortan y remueven la región N-terminal de la proteína SVB, y es en esta región donde se encuentra el dominio de represión de la transcripción. Ahora, sin el represor, SVB está activo y listo para funcionar como factor de transcripción de los genes relacionados con la formación de los tricomas. Esto explica por qué cuando pri es mutado no se forman los tricomas, porque SVB no puede liberar su dominio de represión de la transcripción.
Ahora queda otra pregunta… ¿Estos pequeños péptidos proteolíticos son específicos a determinadas proteínas? Kondo también encontró que pri puede actuar de la misma manera en otras proteínas ya que los mutantes pri tenían otros fenotipos que no eran explicados solamente por la acción de SVB. Lamentablemente, debido al tamaño tan corto de estos péptidos, no pueden ser encontrados usando programas bioinformáticos, tampoco han sido anotados en las bases de datos genéticas, así que ahí hay un arduo trabajo por realizar para encontrar más representantes de los lncRNA en todos los genomas hasta ahora secuenciados. Tampoco se puede estudiar sus estructuras secundarias o terciarias – si las tienen – o sus funciones porque los algoritmos de predicción de proteínas tienen un umbral de detección de unos 100 aminoácidos. Sin embargo, se puede usar la misma técnica usada para buscar e identificar los miARNs, los cuales son pequeñas secuencias de ARN de unos 22 nucleótidos, usando mutantes e identificando que causa este fenotipo.
Otra pregunta… ¿cuantos pequeños péptidos funcionales como los pri pueden estar escondidos dentro de cualquier ARNm? Nosotros podemos saber que un determinado ARNm codifica para una determinada proteína, pero, si cortamos esta secuencia de aminoácidos en trozos más pequeños al azar, ¿cuántos de ellos podrían llegar a ser péptidos funcionales?. En los ARNm también podemos encontrar en extremo 5’ una región que no llega a ser traducida, llamada 5’-UTR. Se ha encontrado que el ~40% de los ARNm de D. melanogaster poseen pequeños ORFs (marcos abiertos de lectura) en la región 5’-UTR (uORF) y muchos de ellos llegan a ser altamente conservados. Y no sólo se han encontrado en insectos, también hay uORFs en levaduras y vertebrados, casi en la misma proporción.
Si estos pequeños péptidos tuvieran muchas proteínas como substratos, sería uno de los mecanismos de regulación de la expresión genética más importantes, pero eso no lo podemos saber hasta no hacer un estudio más profundo. Además, ciertas mutaciones podrían introducir codones de inicio en los lncRNA o en las 5’-UTR, lo cual generaría muchos pequeños péptidos, donde habría la posibilidad de que alguno de ellos sea funcional, y sabiendo que muchas proteínas tienen muchos dominios, estos péptidos podría escindir tales dominios y darle una nueva función o especificidad a la proteína. Sin dudas, esto sería un gran avance hacia el desarrollo de nuevas sustancias terapéuticas como antivirales y anticarcinógenos.
Una pregunta más… ¿por qué la naturaleza ha creado tantos mecanismos de regulación de la expresión genética? Fue la misma pregunta que se hizo al descubrirse los miARN y la respuesta es que todos los seres vivos son parsimoniosos o tienden a serlo, nadie gasta más energía de la que requiere. Para que gastar energía produciendo una proteína que regule o degrade un ARNm o una proteína si pequeñas moléculas (péptidos o ARNs) lo pueden hacer rápida y eficientemente. así que hay bastante trabajo en este campo de la biología molecular.
Referencias:
Kondo, T., Plaza, S., Zanet, J., Benrabah, E., Valenti, P., Hashimoto, Y., Kobayashi, S., Payre, F., & Kageyama, Y. (2010). Small Peptides Switch the Transcriptional Activity of Shavenbaby During Drosophila Embryogenesis Science, 329 (5989), 336-339 DOI: 10.1126/science.1188158
Imagen: 10.1126/science.1192769
