Existen muchos mecanismos que regulan la expresión genética, los cuales pueden darse a nivel transcripcional (promotores, operadores, terminadores), post-transripcional (formación de estructuras secundarias, ARN de interferencia, micro ARNs), traduccional (estructuras secundarias que evitan el paso del ribosoma) y post-traduccional (apoproteínas y chaperonas encargadas de dar la estructura final a la proteína).
Sin embargo, muchos de los ARNs que son transcritos a partir del ADN no llegan a codificar ningún tipo de proteína, y muchos de estos ARNs no codificantes están envueltos en mecanismos de regulación de la expresión genética, por ejemplo: los micro ARN (miARN), ARN de interferencia (ARNi), ARN pequeño de interferencia (siARN) y ARN asociados a piwi (piARN). Pero, aún existe un tipo de ARN misterioso cuya importancia aún se mantiene en discusión, este es el ARN largo no codificante (lncRNA).
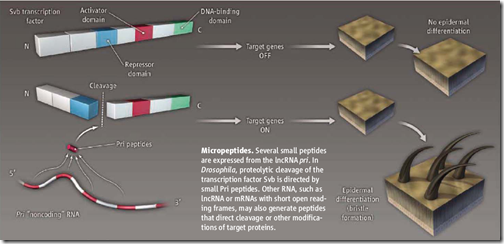
Kondo et al. hicieron un descubrimiento en la mosca de la fruta (Drosophila melanogaster). Un ARN conocido como polished-rice (pri) tenía las características de un lncRNA, pero, codificaba para pequeños péptidos que controlaba la expresión de ciertos genes durante el desarrollo embrionario de D. melanogaster. Este gen pri ya había sido descubierto anteriormente en otros insectos donde también cumplía funciones similares. Lo que más llamó la atención de este gen pri es que era policistrónico, o sea, el gen transcribía un solo ARNm pero era traducido en varios pequeños péptidos de manera independiente, similar a lo que pasa en un operón bacteriano. pri codificaba cuatro o cinco pequeños péptidos de 11 a 34 aminoácidos, altamente conservados en los insectos.
Para investigar la función de pri, Kondo et al. usaron embriones de moscas de la fruta y estudiaron sus células epidérmicas, específicamente, unas especializadas en la producción de pequeñas protuberancias llamadas tricomas (parecido a los tricomas de las plantas). El gen responsable de la formación de los tricomas es el gen shavenbaby (svb), el cual codifica una proteína que actúa como factor de transcripción de genes relacionados con la formación de los tricomas. Cuando svb era mutado, los embriones eran incapaces de formar tricomas; y además, cuando pri era mutado, el embrión tampoco podía formar tricomas, pero la expresión de svb no se veía afectada. Uhm… para que no se pierdan… cuando pri y svb están presentes se forman los tricomas; cuando se mutaba svb pero no pri no se llegaban a formar los tricomas; y cuando se mutaba pri pero no svb, tampoco se formaban los tricomas, pero, la expresión de la proteína SVB no se veía afectada. Entonces, de esto se puede deducir que pri regula la expresión de svb a nivel post-traduccional, produciéndole algún tipo de modificación que lo vuelva activo, pero, ¿como es este mecanismo?
Primero, debemos saber que la proteína SVB tiene varios dominios, uno de ellos es un dominio que actúa como factor de transcripción. Kondo encontró que pri actúa como un interruptor temporal en la formación de los tricomas. Los pequeños péptidos codificados por pri tienen acción proteolítica, o sea, tienen la capacidad de cortar ciertos enlaces peptídicos. Los pequeños peptidos de pri cortan y remueven la región N-terminal de la proteína SVB, y es en esta región donde se encuentra el dominio de represión de la transcripción. Ahora, sin el represor, SVB está activo y listo para funcionar como factor de transcripción de los genes relacionados con la formación de los tricomas. Esto explica por qué cuando pri es mutado no se forman los tricomas, porque SVB no puede liberar su dominio de represión de la transcripción.
Ahora queda otra pregunta… ¿Estos pequeños péptidos proteolíticos son específicos a determinadas proteínas? Kondo también encontró que pri puede actuar de la misma manera en otras proteínas ya que los mutantes pri tenían otros fenotipos que no eran explicados solamente por la acción de SVB. Lamentablemente, debido al tamaño tan corto de estos péptidos, no pueden ser encontrados usando programas bioinformáticos, tampoco han sido anotados en las bases de datos genéticas, así que ahí hay un arduo trabajo por realizar para encontrar más representantes de los lncRNA en todos los genomas hasta ahora secuenciados. Tampoco se puede estudiar sus estructuras secundarias o terciarias – si las tienen – o sus funciones porque los algoritmos de predicción de proteínas tienen un umbral de detección de unos 100 aminoácidos. Sin embargo, se puede usar la misma técnica usada para buscar e identificar los miARNs, los cuales son pequeñas secuencias de ARN de unos 22 nucleótidos, usando mutantes e identificando que causa este fenotipo.
Otra pregunta… ¿cuantos pequeños péptidos funcionales como los pri pueden estar escondidos dentro de cualquier ARNm? Nosotros podemos saber que un determinado ARNm codifica para una determinada proteína, pero, si cortamos esta secuencia de aminoácidos en trozos más pequeños al azar, ¿cuántos de ellos podrían llegar a ser péptidos funcionales?. En los ARNm también podemos encontrar en extremo 5’ una región que no llega a ser traducida, llamada 5’-UTR. Se ha encontrado que el ~40% de los ARNm de D. melanogaster poseen pequeños ORFs (marcos abiertos de lectura) en la región 5’-UTR (uORF) y muchos de ellos llegan a ser altamente conservados. Y no sólo se han encontrado en insectos, también hay uORFs en levaduras y vertebrados, casi en la misma proporción.
Si estos pequeños péptidos tuvieran muchas proteínas como substratos, sería uno de los mecanismos de regulación de la expresión genética más importantes, pero eso no lo podemos saber hasta no hacer un estudio más profundo. Además, ciertas mutaciones podrían introducir codones de inicio en los lncRNA o en las 5’-UTR, lo cual generaría muchos pequeños péptidos, donde habría la posibilidad de que alguno de ellos sea funcional, y sabiendo que muchas proteínas tienen muchos dominios, estos péptidos podría escindir tales dominios y darle una nueva función o especificidad a la proteína. Sin dudas, esto sería un gran avance hacia el desarrollo de nuevas sustancias terapéuticas como antivirales y anticarcinógenos.
Una pregunta más… ¿por qué la naturaleza ha creado tantos mecanismos de regulación de la expresión genética? Fue la misma pregunta que se hizo al descubrirse los miARN y la respuesta es que todos los seres vivos son parsimoniosos o tienden a serlo, nadie gasta más energía de la que requiere. Para que gastar energía produciendo una proteína que regule o degrade un ARNm o una proteína si pequeñas moléculas (péptidos o ARNs) lo pueden hacer rápida y eficientemente. así que hay bastante trabajo en este campo de la biología molecular.
Referencias:
Kondo, T., Plaza, S., Zanet, J., Benrabah, E., Valenti, P., Hashimoto, Y., Kobayashi, S., Payre, F., & Kageyama, Y. (2010). Small Peptides Switch the Transcriptional Activity of Shavenbaby During Drosophila Embryogenesis Science, 329 (5989), 336-339 DOI: 10.1126/science.1188158
Imagen: 10.1126/science.1192769





0 comentarios:
Publicar un comentario
Se respetuoso con tus comentarios y críticas. Cualquier comentario ofensivo será eliminado.