De manera sencilla, los fitoplasmas son bacterias patógenas de plantas transmitidas por insectos. Como pueden ver en la imagen de portada, son parásitos intracelulares estrictos, o sea, sólo pueden vivir dentro de las células, en este caso, las del floema. Su vector es un insecto de la familia de los cicadélidos llamado Macrosteles quadrilineatus o simplemente “chicharrita” o “cigarritas”.
Si bien el fitoplasma parece ser una bacteria muy sencilla, posee una característica fascinante: es capaz de manipular la fisiología del hospedero para atraer y favorecer el éxito reproductivo del insecto que lo transmite.
Parásitos manipuladores de hospederos hay muchos, y este año hemos visto a algunos de ellos, por ejemplo: los hongos del género Ophiocordyceps, que controlan la mente de las hormigas para que éstas se dirijan a zonas que favorecen la diseminación de sus esporas; los baculovirus que ejercen un efecto parecido a las Ophiocordyceps pero en la oruga de la polilla gitana. Otros parásitos como el Plasmodium falciparum —agente causante de la malaria— que vuelve a la persona infectada más atractiva para los mosquitos. En plantas, por ejemplo, el hongo Puccinia monoica induce a la planta infectada a generar unas flores falsas que logran engañar a los insectos polinizadores, quienes ingenuamente transmiten las esporas del hongo a otras plantas. Y en plagas, también hemos visto el caso del pulgón donde una de sus bacterias intestinales llamada Staphylococcus sciuri lo traiciona y produce sustancias volátiles que atraen a sus depredadores. Pero el caso que veremos ahora es único porque el efecto del fitoplasma se da a dos niveles, tanto en el hospedero como en el vector.
Gracias a un estudio publicado en PNAS, donde participaron un grupo de investigadores británicos del Centro John Innes, ahora podemos entender, a nivel fisiológico y molecular, la fascinante estrategia empleada por los fitoplasmas.

Para corroborar su hipótesis, Sugio y sus colaboradores analizaron el genoma completo de una cepa de fitoplasma, causante de la enfermedad “escoba de bruja”, un tipo de amarillamiento del áster. Esta cepa, conocida simplemente como AY-WB (Aster Yellow phytoplasma strain Witches’ Broom), afecta a un gran número de especies de plantas diferentes, principalmente del grupo de las brasicáceas, al cual pertenece la planta modelo Arabidopsis thaliana.
Al estudiar el genoma de AY-WB, no encontraron genes que codifiquen para el sistema de secreción tipo III. Esto indicaría que si el fitoplasma libera algún tipo de efector hacia la célula hospedera, lo debería de hacer vía el sistema de secreción dependiente de Sec. Este sistema de secreción requiere de una pequeña secuencia de aminoácidos específicos (señal) ubicada al inicio de la proteína que va ser secretada. Los investigadores encontraron secuencias que codifican para 56 proteínas con esta señal dentro del genoma de AY-WB.
Para determinar cuál de los 56 candidatos era el efector involucrado con la estrategia adoptada por el fitoplasma, Sugio y sus colegas probaron el efecto de cada uno de ellos sobre la interacción entre A. thaliana y el chicharrita. Para ello, los investigadores insertaron cada uno de los genes correspondientes a los candidatos en diferentes A. thaliana. De todas las plantas probadas, sólo la que poseía el efector SAP11 mostraba un efecto beneficioso sobre la fecundidad de las chicharritas.
Una vez reconocido al efector, los investigadores identificaron con qué proteínas de A. thaliana se involucraba. Los experimentos mostraron que SAP11 se unía y desestabilizaba un importantísimo grupo de factores de transcripción de las plantas llamado TCPs de clase II, cuya secuencia está muy relacionada al factor de transcripción CINCINNATA presente en el género Antirrhinum. Estos factores están involucrados con importantes procesos dentro del desarrollo de las plantas, por ejemplo: regulan la maduración de las hojas, la proliferación celular y están involucradas con la senescencia.
Sin embargo, los investigadores observaron que la desestabilización de los factores CIN-TCPs inhibían la expresión de una lipooxigenasa (LOX2). Esta enzima —que se encuentra activa durante la senescencia— participa en el primer paso de la biosíntesis del jasmonato, una fitohormona que protege a la planta del ataque de insectos herbívoros. Los investigadores corroboraron esto al ver que los niveles de jasmonato en las plantas infectadas disminuyeron en más del 50%.
Entonces, como el jasmonato controla la presencia de insectos devoradores de hojas, entre ellos la chicharrita, una reducción en sus niveles normales de concentración pueden favorecer la presencia del vector. Pero, no sólo eso. Las chicharritas suelen dejar sus huevecillos en estas plantas para que cuando eclosionen se alimenten de sus hojas, así que los bajos niveles de jasmonato de las plantas infectadas favorecerán la supervivencia y maduración de las ninfas, las cuales una vez maduren, trasmitirán el fitoplasma hacia otras plantas. Los investigadores corroboraron esto al observar que en las plantas infectadas por el fitoplasma hubo un aumento del 60% en el número de ninfas.
Por otro lado, Sugio y sus colegas también observaron que las chicharritas ponían más huevos de lo normal. Esto indicaría que hay otros efectores, dentro de los 56 candidatos, que también podrían modificar la fisiología del insecto para volverlo más fértil. Esta hipótesis aún falta ser corroborada pero esta investigación ha abierto nuevas perspectivas en las relaciones evolutivas entre los hospederos, parásitos y vectores.
Referencia:
Sugio, A., Kingdom, H., MacLean, A., Grieve, V., & Hogenhout, S. (2011). PNAS Plus: Phytoplasma protein effector SAP11 enhances insect vector reproduction by manipulating plant development and defense hormone biosynthesis Proceedings of the National Academy of Sciences DOI: 10.1073/pnas.1105664108
Esta entrada participa en el VII Carnaval de Biología celebrado este mes en el blog Curiosidades de la Microbiología.








![permafrost[1] permafrost[1]](https://blogger.googleusercontent.com/img/b/R29vZ2xl/AVvXsEjKBscCEkOFM9YsitdSFjdoed_AhC-QC9PCVLkfSi6nQRnWuzTc7mccNs5gRW7v30G5MVjZk7IX0lX6ej7Tk0NrdvQsSA0hix2V_IzGMXAu6IwvYaO55fQ-kKdFfVYn01_mtg7m/?imgmax=800)







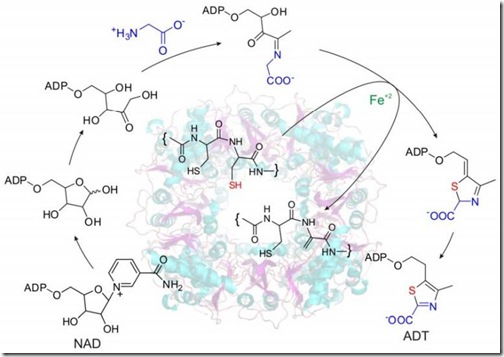
 Fragmento correspondiente al sitio activo. CC corresponde a las cisteínas. WT es la versión normal o silvestre y R301Q es la versión mutada. Las acetamidas (CM) sólo pueden unirse a la versión mutada porque el azufre de la cisteína-205 no puede pasar al tiazol. Cuando el azufre de la Cys-205 se pierde, se forma una dehidroalanina, la cual reacciona con el azufre del Cys-204 formando un enlace cíclico que no puede reaccionar con la acetamida.
Fragmento correspondiente al sitio activo. CC corresponde a las cisteínas. WT es la versión normal o silvestre y R301Q es la versión mutada. Las acetamidas (CM) sólo pueden unirse a la versión mutada porque el azufre de la cisteína-205 no puede pasar al tiazol. Cuando el azufre de la Cys-205 se pierde, se forma una dehidroalanina, la cual reacciona con el azufre del Cys-204 formando un enlace cíclico que no puede reaccionar con la acetamida.