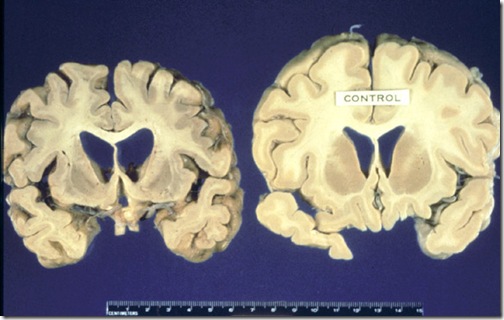
Enzima encargada de remover los grupos acetil de las histonas sería responsable de el desarrollo de enfermedades como el Huntington.
El ADN humano es tan largo que su longitud excede en cien mil veces el diámetro del núcleo de las células. Unas proteínas llamadas histonas se encargan de compactarlo de tal manera que quepa dentro de este pequeño espacio. Sin embargo, para que los genes puedan ser transcritos y expresados a proteínas, el ADN debe ser desenrollado para poder facilitar el trabajo de la ARN polimerasa.
Las histonas poseen unas pequeñas moléculas adicionales unidas a algunos de sus aminoácidos. Una de ellas es el grupo acetil que, cuando está ausente, el ADN puede desenrollarse. Gracias a la acción de unas enzimas conocidas como las histona deacetilasas (HDAC) estos grupos acetil pueden ser removidos. Pero, según un artículo publicado el 21 de Febrero en PLoS Biology, las HDAC pueden generar mutaciones a través del aumento de repeticiones de trinucleótidos, tanto en levaduras como en células humanas.
Las repeticiones nucleotídicas, tales como la citosina-timina-guanina (CTG)n, son comunes dentro de nuestro genoma. Normalmente el número de veces (n) que se encuentran repetidas es constante; pero hay casos en los que este número aumenta. Si exceden un umbral de 30 a 40 repeticiones, pueden generar mutaciones que afectan la actividad de los genes. En los humanos la cosa es más grave aún porque se ha demostrado que la expansión de las repeticiones trinucleotídicas causa al menos 17 enfermedades neurodegenerativas, entre ellas la enfermedad de Huntington y la distrofia miotónica tipo 1 (DM1).
Sin embargo, las causas de este incremento en el número de repeticiones trinucleotídicas es desconocido. Con el fin de desvelar este misterio, un grupo de investigadores de la Universidad Nacional de Irlanda Galway, liderados por Kim Debacker y Aisling Frizzell, usaron levaduras que presentaban repeticiones (CTG)20 a quienes le mutaron alrededor de 9,000 genes para identificar cuál de ellos promovían una reducción en el número de repeticiones. Para ello sometieron a los mutantes a un tratamiento con canavanina, una molécula que provoca daños en la estructura de las proteínas. Aquellas levaduras que presentan un mayor número de repeticiones CTG son más resistentes a este compuesto.
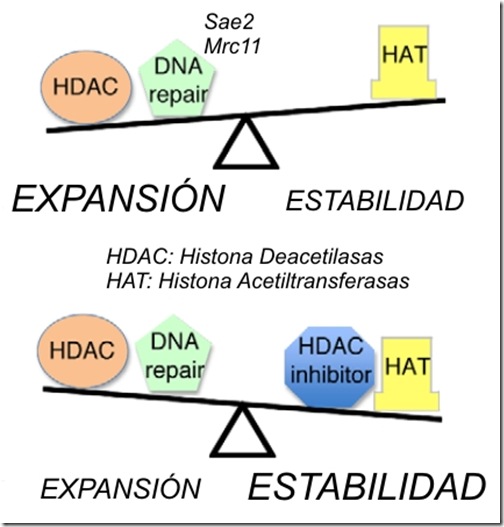
A partir de este experimento, los investigadores identificaron 11 genes mutantes que aumentaban la sensibilidad de la levadura a la canavanina, indicando una reducción en el número de repeticiones CTG. Tres de estos genes estaban relacionados con dos HDAC conocidas como Rpd3L y Hda1. Cuando se quitaban cualquiera de estos tres genes o se usaba la tricostatina-A (TSA) —un inhibidor químico de las HDAC—, el número de repeticiones CTG se reducían entre un 50% y 90%.
Para ver que ocurría en humanos, Debacker & Frizzell estudiaron la HDAC3, el homólogo de la Rpd3L de levaduras. Para ello usaron astrocitos, un tipo de células nerviosas humanas que muestran una expansión de las repeticiones trinucleotídicas en pacientes con la enfermedad de Huntington. Cuando usaron un inhibidor específico de las HDAC3 conocido como 4b, la frecuencia de expansión se redujo en un 75%. El mismo efecto se observó cuando se bloqueó la expresión del gen hdac3 mediante un ARN de interferencia. Sin embargo, la inhibición de otras HDAC como la HDAC1 y HDAC2, no tuvo el mismo efecto.
Por otro lado, un efecto opuesto se observó al bloquear la expresión de las proteínas encargadas de poner grupos acetil a las histonas, tales como la CPB o la p300. En este caso, la expansión de las repeticiones trinucleotídicas aumentó.
Otro experimento demostró que la acción de la HDAC3 se puede dar a la distancia y no necesariamente en el lugar donde se da la expansión de las repeticiones nucleotídicas. Esto indicaría que hay un mecanismo o una molécula intermediaria que une estos dos procesos.
Para determinar quién es el intermediario, los investigadores se enfocaron en el estudio de una enzima que corta el ADN (una nucleasa) conocida como Sae2. Estudios previos demostraron que Sae2 es estabilizada por la deacetilación de las histonas, y que su función es reparar el ADN junto a otra nucleasa llamada Mre11. Cuando Debacker & Frizzell mutaron el gen sae2, la expansión de las repeticiones trinucleotídicas se redujo parcialmente, lo que indicaría que la nucleasa Sae2 es uno —pero no el único— de los intermediarios en este proceso. Cuando mutaron el gen mre11, también se observó el mismo efecto. Sin embargo, esta nucleasa no interactúa con las HDAC de las levaduras, por lo que su efecto sería a través de la interacción con la Sae2.
Con estos resultados tenemos el siguiente esquema [Adaptado de Choi, CQ]
Entonces, las mutaciones o inhibición completa de la HDAC3 humana podría reducir la frecuencia de la expansión de las repeticiones trinucleotídicas y, por lo tanto, podría ser una buena estrategia para el tratamiento de enfermedades neurodegenerativas como el Huntington, la Distrofia Miotónica, entre otras. Incluso ya vienen siendo probadas tal como demuestran estas dos revisiones: Butler & Bates (2006) y Kasantzev & Thompson (2008).
En vista que muchos de estos desórdenes neurológicos son hereditarios, y en el caso del Huntington es dominante (si tienes un alelo mutante de un gen llamado “huntingtina” desarrollarás la enfermedad), los inhibidores de las HDAC podrían reducir los efectos o hacer más lenta la progresión de la enfermedad.
Referencias:
Debacker, K., Frizzell, A., Gleeson, O., Kirkham-McCarthy, L., Mertz, T., & Lahue, R. (2012). Histone Deacetylase Complexes Promote Trinucleotide Repeat Expansions PLoS Biology, 10 (2) DOI: 10.1371/journal.pbio.1001257
Choi, CQ. (2012). Enzyme Inhibitor May Offer Dual Protection against Brain Disease. PLoS Biol, 10(2): e1001270. doi:10.1371/journal.pbio.1001270






0 comentarios:
Publicar un comentario
Se respetuoso con tus comentarios y críticas. Cualquier comentario ofensivo será eliminado.