El ejemplo más representativo de una estructura capaz de auto-replicarse —hacer copias de sí misma— es el ADN. En las células, este proceso se lleva a cabo, de manera autónoma, gracias a la presencia de enzimas como la ADN polimerasa o la ARN polimerasa. Sin embargo, hace unos 30 años se descubrió unas moléculas de ARN capaces de realizar funciones catalíticas como si fueran enzimas, por ejemplo: cortar otras moléculas de ARN y replicarse a sí misma. A estas moléculas las llamaron “enzimas de ARN” o simplemente ribozimas. Años más tarde, los científicos desarrollaron estos sistemas auto-replicativos de manera artificial.
El ADN es una estructura hermosa conformada por dos cadenas de cuatro nucleótidos (Adenina, Timina, Guanina y Citosina) los cuales interactúan de manera complementaria —A con T y G con C. Esta complementariedad le permite al ADN transmitir y almacenar información. En el año 2006, Rothemund tomó una larga cadena de ADN y la plegó para formar nanoestructuras bidimensionales sumamente complejas, demostrando así que el ADN se podía programar para ensamblarse de formas específicas. Este proceso es conocido como “origami de ADN”.
Ahora, un grupo de investigadores de la Universidad de Nueva York liderados por el químico Tong Wang han usado unas estructuras de ADN de triple hélice como si fueran piezas de Lego®, capaces de reconocer y unirse a otras piezas complementarias para formar estructuras más complejas pre-programadas. Según el artículo que aparece publicado en Nature este trabajo puede ser el primer paso hacia el desarrollo de materiales capaces de auto-replicarse.
Como pueden ver en la figura inicial, cada pieza está formada por varias cadenas de ADN, de diferentes tamaños, que se ordenan en una estructura de triple hélice conocida como motivo de triple entrecruzamiento doblado o BTX (Bent Triple Crossover motif). Cada BTX cuenta con cuatro cadenas de hebra simple laterales de 7 nucleótidos, los cuales le permiten reconocer e interactuar lateralmente con otros BTX. También cuentan con seis cadenas de hebra simple —tres en cada extremo— de 9 nucleótidos cada una para unirse a otros BTX linealmente y así formar cadenas más largas.
Para que capten mejor la idea, consideren a los BTX como si fueran nucleótidos. Estos se unirán a otros BTX para formar cadenas lineales. Además, cada BTX tendrá su BTX complementario que se unirá lateralmente.
La ventaja de estas estructuras es que funcionan como un código de 428 letras. Cada BTX tiene 4 cadenas de hebra simple laterales. Cada cadena está formada por 7 nucleótidos que pueden ser A, T, C o G. Entonces, el número total de combinaciones será 4(7x4).
Para demostrar la validez y funcionalidad de su trabajo, Wang y sus colaboradores usaron dos tipos de piezas (A y B) con sus respectivas piezas complementarias (A’ y B’). Para facilitar su identificación, los investigadores les pusieron unas horquillas de ADN perpendiculares a los BTX que pueden ser marcados y observados bajo el microscopio de fuerza atómica. [Pueden ampliar las imágenes en una ventana extra para observar mejor los detalles].

De esta manera se tendrá una secuencia LNNLNLN, donde L representa a los bloques marcados y N a los que no están marcados. Los siete componentes se mezclan en una solución durante 48 horas. Al cabo de este tiempo, la cadena molde estará formada y puede ser observada bajo el microscopio.
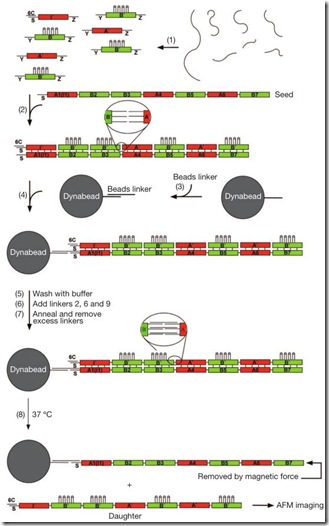
Luego, se eleva la temperatura para que los enlaces laterales de 7 nucleótidos se separen. La temperatura no afecta a los enlaces longitudinales porque estos son más largos y numerosos (tres regiones complementarias de 9 nucleótidos cada una, ricos en G y C, los cuales forman enlaces triple). Una vez separada la cadena de BTX complementarios, se aplica una fuerza magnética para jalar la esfera portando la secuencia molde, la cual puede ser usada para un nuevo proceso replicativo.
Por otro lado, la cadena BTX hija puede ser usada como molde para una segunda ronda de replicación siguiendo este mismo proceso, de esta manera, la cadena BTX nieta será similar a la cadena molde original.
Bajo el microscopio de fuerza atómica, las cadenas se ven así:
Por ahora el proceso es un poco largo y laborioso, requiere de muchos ciclos químicos y térmicos, además, las cadenas complementarias no se pueden generar exponencialmente ya que el número de cadenas molde siempre será la misma. Sin embargo, la PCR, en sus inicios, fue similar y ahora es un proceso completamente automatizado que no tarda más de una hora desde el momento que se extrae el ADN hasta que se analizan los resultados en la computadora.
Pero, lo fascinante de este trabajo es que se pueden generar un número elevado de combinaciones diferentes, además, se puede aprovechar la capacidad de las cadenas de ácidos nucleicos para formar estructuras secundarias y terciarias, de esta manera poder elaborar materiales tridimensionales sumamente complejos con capacidad de autoensamblarse y autoreplicarse. Por otro lado, se pueden usar híbridos de ADN-ARN para dar características especiales al material, con elasticidades diferentes en función a su secuencia de nucleótidos —aquellas secuencias ricas en G y C serán más rígidas que las secuencias ricas en A y T.
Wang, T., Sha, R., Dreyfus, R., Leunissen, M., Maass, C., Pine, D., Chaikin, P., & Seeman, N. (2011). Self-replication of information-bearing nanoscale patterns Nature, 478 (7368), 225-228 DOI: 10.1038/nature10500








0 comentarios:
Publicar un comentario
Se respetuoso con tus comentarios y críticas. Cualquier comentario ofensivo será eliminado.