Uno de los principales objetivos de la biología sintética es el desarrollo de sensores biológicos con la capacidad de generar una respuesta lógica —o “inteligente”— en función al estímulo recibido. Pongamos un ejemplo para ilustrar mejor esta idea:
Tenemos un individuo que ha sido infectado por un microorganismo patógeno. Lo que normalmente se hace es tomar una muestra del paciente —ya sea sangre, orina, líquido cefalorraquídeo, o algún otro fluido corporal— para hacerle una serie de ensayos en el laboratorio con el fin de identificar al agente infeccioso. Una vez identificado, se le administra un determinado fármaco que controle la infección y elimine al organismo indeseado. Todo este proceso demanda mucho tiempo y puede llegar a costar mucho dinero.
Imaginen ahora que cuentan con un biosensor capaz de realizar todo este proceso en un solo paso. Este hipotético biosensor, el cual fue desarrollado gracias a la biología sintética, es capaz de reconocer al agente infeccioso a través de antígenos específicos liberados por él. Además, en función al antígeno reconocido, el biosensor activa una ruta metabólica que sintetiza el antibiótico específico contra este patógeno, terminando así con la infección. Y una vez cumplido su trabajo, se autoelimina.
En teoría, esto se puede realizar a través del uso de moléculas señalizadoras, factores de transcripción y genes, que pueden ser conectados para formar un circuito lógico, tal como los que se encuentran en cualquier dispositivo electrónico. Un circuito lógico no es más que la unión y conexión de varias operaciones lógicas donde una señal de entrada (input) genera una respuesta (output), definida por una operación específica (gate).
En los últimos años, muchos investigadores e ingenieros han desarrollado circuitos lógicos biológicos usando bacterias y moléculas de ADN. El principio es bastante sencillo. Un estímulo es reconocido por una molécula señalizadora que activa la expresión de un gen, el cual puede codificar para un factor de transcripción o una molécula represora. Estas moléculas, a su vez, pueden activar o reprimir la expresión de otros genes, creándose así un circuito lógico genético.
Sin embargo, hay dos inconvenientes que afectan el desarrollo de esta tecnología: la interacción de los circuitos genéticos con los propios genes y moléculas del organismo que lo transporta (ruido genético), el cual puede generar reacciones cruzadas; y el número limitado de señales químicas capaces de formar parte de un circuito genético. En un estudio publicado en Nature Communications, investigadores del Imperial College London liderados por el profesor Martin Buck, han desarrollado dos circuitos lógicos genéticos modulares (AND y NAND) que han logrado superar estos inconvenientes.
Antes de describir el trabajo, definiremos que son los operadores lógicos AND y NAND. Digamos que tenemos dos estímulos diferentes (I1 e I2). En la operación lógica AND, sólo se generará una respuesta cuando los dos estímulos están presentes al mismo tiempo. Por el contrario, en la operación lógica NAND, la respuesta se dará sí y solo sí ninguno de los dos estímulos esté presente.
Para implementar estas operaciones lógicas a nivel genético, el Dr. Baojun Wang —autor principal del trabajo— y sus colegas usaron el sistema de respuesta hipersensible y patogenicidad (Hrp) de la bacteria Pseudomona syringae. Este sistema está conformado por tres componentes: HrpR, HrpS y HrpL. ¿Cómo funciona?. Cuando HrpR y HrpS se expresan y se juntan activan un factor de transcripción conocido como σ54. Este factor de transcripción reconoce la región promotora HrpL, activándola y expresando los genes que conforman un sistema de transporte de proteínas, importante para la virulencia de la bacteria.
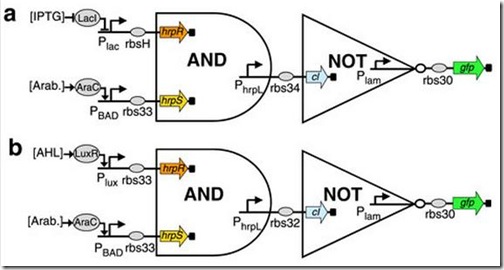
Lo que hizo Wang fue poner los componentes HrpR y HrpS bajo el control de dos promotores diferentes (P1 y P2), que se activan sólo en presencia de sus moléculas inductoras (I1 e I2); y en el otro extremo, el gen reportero —también conocido como output (gfp: proteína fluorescente verde)— estará bajo el control del promotor HrpL. Entonces, si I1 e I2 están presentes, HrpR y HrpS podrán ser expresados, activando al factor σ54 que se unirá a la región promotora HrpL, permitiendo la expresión del gen reportero. Sin embargo, si sólo está presente uno o ninguno de los inductores, no se podrá formar el factor σ54 y no se podrá expresara la gfp.
Este pequeño circuito lógico fue probado en distintas cepas de E. coli, funcionando correctamente en la mayoría de ellas. Como inductores usaron tres mecanismos ampliamente conocidos: i) el IPTG, que induce el promotor Plac, ii) la arabinosa, que induce el promotor PBAD y, iii) la homoserin-lactona (AHL), molécula responsable de la comunicación bacteriana que activa el promotor Plux. También probaron distintas secuencias RBS (secuencias reconocidas por los ribosomas al momento de traducir el ARNm), las cuales presentan eficiencias de traducción variables. Estas secuencias RBS sirvieron para ajustar los niveles de expresión de los componentes del circuito lógico (HrpS, HrpR y GFP).
Con este experimento, Wang y sus colaboradores demostraron que se podía reducir el efecto del ruido genético mediante el uso de mecanismos de regulación que naturalmente no se encuentran en determinados organismos, y que también se podían desarrollar circuitos genéticos a manera de módulos, que pueden ser “introducidos” o “quitados” del organismo, tal como las tarjetas de video, sonido o red de una computadora. Los investigadores creían que el desarrollo de estos módulos podría facilitar la formación de circuitos lógicos mucho más complejos.
Para demostrar este segundo supuesto, Wang y sus colegas desarrollaron el operador lógico NOT. Para ello usaron el promotor del fago lambda Plam que regula la expresión del gen reportero gfp. Este promotor esta activo todo el tiempo a menos que esté presente su molécula represora cI. Luego, conectaron este pequeño operador lógico al primero (AND) —al que le reemplazaron el output gfp por el represor cI— para así obtener el operador lógico NAND.
En este segundo caso, cuando los dos inductores se encuentran presentes, el factor σ54 activa el promotor HrpL permitiendo la expresión del represor cI. El represor inactivará la expresión del gen reportero gfp —no habrá un output. Pero, si sólo está presente uno o ninguno de los inductores, el represor cI no podrá expresarse y el output seguirá expresándose.
Bueno, desarrollar pequeños circuitos lógicos es una tarea relativamente sencilla, el reto viene cuando se pretende hacer circuitos lógicos mucho más complejos, capaces de reconocer diferentes estímulos, por ejemplo: agentes infecciosos, sustancias contaminantes, toxinas, células cancerosas, etc. Para ello, primero debemos tener un profundo entendimiento de los procesos de transducción de señales que se llevan a cabo dentro de las células, en la cual participan una serie de proteínas que reaccionan mediante fosforilaciones y desfosforilaciones, ubiquitinaciones y desubiquitinaciones. Sin embargo, podemos decir que estamos viviendo una época donde se están sentando las bases de la biología sintética y la computación molecular. Con lo rápido que avanza la ciencia, tal vez dentro de algunos años obtengamos los primeros biosensores basados en circuitos lógicos integrados.
Referencia:
Wang, B., Kitney, R., Joly, N., & Buck, M. (2011). Engineering modular and orthogonal genetic logic gates for robust digital-like synthetic biology Nature Communications, 2 DOI: 10.1038/ncomms1516
Esta entrada participa en el III Carnaval de la Tecnología celebrado en el blog Idea Secundaria.







0 comentarios:
Publicar un comentario
Se respetuoso con tus comentarios y críticas. Cualquier comentario ofensivo será eliminado.