[Antes de leer esta entrada, pueden leer acerca de Foldit en el artículo que escribí en el blog el año pasado]
En tan solo tres semanas, los jugadores de Foldit han resuelto la estructura tridimensional de una enzima de un retrovirus llamada proteasa, cuya configuración ha sido esquiva a los científicos por más de una década. Según el artículo publicado hoy en Nature Structural & Molecular Biology, este logro provee una excelente oportunidad para diseñar fármacos más eficientes para tratar diferentes enfermedades virales como el VIH.
Las proteínas están formadas por una determinada secuencia de aminoácidos. Cada aminoácido tiene propiedades químicas diferentes que influyen en la forma y función de la proteína. Como simple analogía, imagínense alambre metálico que adquiere una determinada forma en función a la fuerza que le apliques. En una proteína, las fuerzas son de atracción o repulsión en base a la naturaleza química de cada uno de los 20 aminoácidos diferentes que pueden conformarla.
Para determinar la estructura de una proteína no basta con conocer su secuencia de aminoácidos, ya que las formas como éstos pueden interactuar son infinitas. Los científicos deben purificarlas, cristalizarlas y someterlas a los rayos X o a la resonancia magnética (RMN) para analizar la forma como dispersan o absorben la radiación electromagnética, y mediante programas de computación, analizar los datos para obtener la estructura final. Este proceso es sumamente largo y costoso, y muchas veces las proteínas no pueden purificarse o cristalizarse ya que se desnaturalizan (pierden su forma) en el proceso.
Foldit es un juego de computadora en línea creado por investigadores de la Universidad de Washington, donde los jugadores manipulan las estructuras de proteínas cuyas configuraciones espaciales aún son desconocidas. Usando una serie de herramientas de modelamiento dispuestas en una interfaz gráfica amigable —algo así como el AutoCAD® que usan los ingenieros—, los jugadores doblan, giran, abren y cierran las cadenas de aminoácidos a fin de encontrar la disposición más estable.
En Foldit, los jugadores no parten desde cero, o sea, no parten desde una cadena estirada de aminoácidos, sino parten desde una estructura predicha por una herramienta bioinformática llamada Rosetta. Este programa usa el siguiente algoritmo para encontrar la mejor estructura proteica: i) comienza con una cadena de aminoácidos completamente estirada; ii) mueve una parte de la cadena para obtener una nueva forma; iii) calcula la energía libre de la nueva forma; iv) acepta o rechaza el cambio de energía obtenido (diferencia entre la energía antes y después del movimiento); y v) repite los pasos ii, iii y iv hasta obtener una estructura final predicha. A todos estos pasos se llama “trayectoria” y el programa analiza aquella estructura que tuvo la trayectoria con la cantidad de energía más baja. Finalmente, se hace los ajustes de cada estructura mediante ligeros cambios en las interacciones químicas. Sin embargo, no siempre se obtiene la misma forma de baja energía porque las posibilidades son infinitas.
Debido a la cantidad de cálculos que hace Rosetta, los desarrolladores usan los recursos de las computadoras de personas que voluntariamente las ofrecen al proyecto a través de una simple aplicación llamada Rosetta@home. Mientras no usas tu computadora, Rosetta@home entra en actividad y ejecuta el algoritmo el cual tu puedes observarlo, en tiempo real, como protector de pantalla.
Entonces, a partir de la estructura proteica predicha por Rosetta, lo jugadores de Foldit empiezan a realizar pequeños movimientos a fin de encontrar las formas con menores cantidades de energía libre (termodinámicamente más estables). Además, al ser un juego multijugador, existe una participación colaborativa que acelera el proceso, ya que los jugadores se encuentran agrupados en “clanes” que trabajan de manera conjunta a fin de obtener más puntos y subir en el ranking. Aunque no lo crean, la competencia es muy fuerte ya que, si bien puede ser tomado como un prejuicio, los científicos y la gente que gusta de la ciencia son bastante geeks.
Para el presente trabajo, el equipo del Dr. Firas Khatib puso un puzle a dos de los grupos más renqueados de Foldit —Contenders y Void Crushers. El puzle era la proteasa retroviral del Virus Mason-Pfizer del mono (M-PMV), el cual causa el SIDA en los simios. Estas enzimas cumplen un rol importante en la maduración y proliferación de los virus como el VIH y son usados como blancos de los retrovirales modernos, así que saber su estructura molecular precisa sería de gran ayuda para diseñar fármacos más específicos y eficientes.
Los científicos han tratado de encontrar la forma de esta proteasa usando el reemplazamiento molecular (MR) por más de una década. Esta técnica se basa en determinar la estructura de una proteína comparándola con otras proteína similares con estructuras ya resueltas o a partir de otras formas cristalinas de la misma proteína mucho más fáciles de obtener.
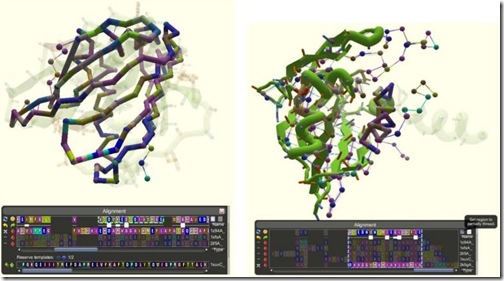
Durante las tres semanas que duró la competencia, los jugadores de Foldit lograron obtener la estructura de la proteasa —partiendo desde los modelos parciales obtenidos mediante RMN—, la cual fue bastante aproximada a la versión original determinada posteriormente mediante cristalografía de rayos X. Además, los modelos obtenidos por los jugadores (Fig. a) fue mejor que los obtenidos por la predicción de Rosetta (Fig. b, c y d).
Para que esta proteasa sea funcional, debe estar formada por dos de estas proteínas conocidas como monómeros. Entonces, la importancia de este trabajo es que al conocer la estructura más precisa de la proteína se pueden diseñar fármacos que actúen a nivel de su superficie y eviten que los monómeros se unan par formar la proteasa funcional. De esta manera, el virus perderá su capacidad de proliferación y la infección puede ser controlada.
Por otro lado, predecir las estructuras usando Foldit y Rosetta@home reduce considerablemente los costos económicos y laborales que acarrea la purificación y cristalización de proteínas, a través de la interacción de los videojuegos y la investigación científica.
Referencia:
Khatib, F., DiMaio, F., Cooper, S., Kazmierczyk, M., Gilski, M., Krzywda, S., Zabranska, H., Pichova, I., Thompson, J., Popović, Z., Jaskolski, M., & Baker, D. (2011). Crystal structure of a monomeric retroviral protease solved by protein folding game players Nature Structural & Molecular Biology DOI: 10.1038/nsmb.2119







0 comentarios:
Publicar un comentario
Se respetuoso con tus comentarios y críticas. Cualquier comentario ofensivo será eliminado.