Los Fusarium son los hongos patogénicos más diversos y ampliamente distribuidos de las plantas. Tenemos especies que tienen un rango de infección muy selectivo como F. graminearum (Fg) y F. verticilliodes (Fv), que sólo infectan cereales; mientras que F. oxysporum (Fo) puede infectar tanto plantas monocotiledóneas como dicotiledóneas, así como personas inmunocomprometidas y otros mamíferos. Así como hay Fusarium malos —patógenos—, también hay buenos —no-patógenos. La diferencia entre ellos es que los primeros presentan genes que codifican para enzimas hidrolíticas, proteínas transportadoras de membrana y algunos factores de transcripción que facilitan la colonización e infección de las plantas. Ahora, la pregunta es, ¿por qué los Fo tienen un mayor rango de infección?
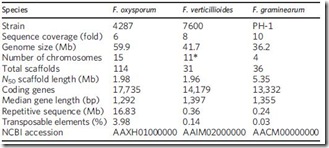

¿Pero que hace especial a la región Fol LS? Sólo el 20% de los genes que fueron predecidos para esta región codificaban para proteínas conocidas y, principalmente, correspondían a efectores de necrosis, factores de virulencia, factores de transcripción (inducción de producción de etileno) y proteínas envueltas en la transducción de señales, y todas secretadas a la planta infectada, en otras palabras, todos estos genes estaban relacionados con la patogenicidad de Fol. También presentaban genes envueltos en el metabolismo de los lípidos, ya que su patogenicidad se basa en señales lipídicas. Otra especie de Fusarium, F. solani, también tiene 3 cromosomas LS, con genes diferentes al Fol LS, pero con la misma función de mejorar la patogenicidad.

Manda un mensaje de texto con tu opción favorita al 90990 y gana una placa petri con Fusarium. Las dos primeras hipótesis se descartaron por que al comparar las secuencias de los genes Fol LS con los genes del genoma central, sólo el 50% eran homólogos. Además, los codones más usados por los genes de Fol LS eran distintos al del genoma central (los genes Fol LS eran ricos en GC en la tercera posición del codón), lo cual indica un diferente origen evolutivo, esto ha sido confirmado con el análisis filogenético, donde la región Fol LS se ubica fuera del grupo Fv, Fg y Fo. Además, los genes de Fol LS tenían el mismo grado de homología con genes de otras especies. Al hacer un BLAST —comparar las secuencias, con todas las secuencias presentes en el GenBank del NCBI—, los genes de Fol LS, también eran homólogos a genes de otros ascomicetos, principalmente de otros Fusarium, lo que indica que la respuesta más parsimoniosa del origen de Fol LS es la transferencia horizontal.
Ahora surge otra pregunta. Si el origen de Fol LS ha sido por transferencia horizontal, entonces, ¿Fol debe tener la capacidad de transferir su región LS a otros Fusarium no-patógenos y volverlos patógenos? Entonces se hicieron pruebas de co-cultivo de una cepa patogénica, Fol007 (como el agente) con una no patogénica, Fo-47, y dos patógenas de otras especies, Foc (patógena del plátano) y Fom (patógena del melón). Se identificó que los principales genes de virulencia del tomate se encontraban en el cromosoma 14 de Fol (uno de los cromosomas Fol LS). Así que al cromosoma 14 se le adjuntó un gen de resistencia a zeocina (un antibiótico selectivo, si no esta presente, el Fusarium muere si el medio tiene zeocina) y a los Fusarium que serán los receptores se les introdujo el gen de resistencia a la higromicina (otro antibiótico selectivo). Se mezclaron y sembraron 1:1 (Fol:[Fo-47, Foc, Fom] una por una) las conidias de los Fusarium y después de 6 semanas germinaron. Se seleccionaron solo las colonias que sobrevivieron a medios de cultivo con los dos antibióticos selectivos (las que llegaron a recibir el cromosoma 14 de Fol). Solo Fo-47 llegó a sobrevivir, y se le denominó Fo-47+. Tal como lo esperaron, Fo-47+ se volvió patógena e infectó las plantas de tomate. Y por si fuera poco, dos de ellas eran altamente patógenas. Al analizar sus cromosomas, se observó que las Fo-47+ más patógenas tenían otro cromosoma Fol LS extra, a parte del cromosoma 14).
Para terminar, la transferencia de genes de virulencia puede ser un mecanismo muy común en la naturaleza, entender mejor como se da este mecanismo y como evolucionan los patógenos nos ayudará a diseñar mejores estrategias para controlar sus daños a los cultivos. Además, este descubrimientos nos indica que los patógenos evolucionan rápidamente, se adaptan fácilmente, aumentando la incidencia de enfermedades.
Referencia:
Ma, L., van der Does, H., Borkovich, K., Coleman, J., Daboussi, M., Di Pietro, A., Dufresne, M., Freitag, M., Grabherr, M., Henrissat, B., Houterman, P., Kang, S., Shim, W., Woloshuk, C., Xie, X., Xu, J., Antoniw, J., Baker, S., Bluhm, B., Breakspear, A., Brown, D., Butchko, R., Chapman, S., Coulson, R., Coutinho, P., Danchin, E., Diener, A., Gale, L., Gardiner, D., Goff, S., Hammond-Kosack, K., Hilburn, K., Hua-Van, A., Jonkers, W., Kazan, K., Kodira, C., Koehrsen, M., Kumar, L., Lee, Y., Li, L., Manners, J., Miranda-Saavedra, D., Mukherjee, M., Park, G., Park, J., Park, S., Proctor, R., Regev, A., Ruiz-Roldan, M., Sain, D., Sakthikumar, S., Sykes, S., Schwartz, D., Turgeon, B., Wapinski, I., Yoder, O., Young, S., Zeng, Q., Zhou, S., Galagan, J., Cuomo, C., Kistler, H., & Rep, M. (2010). Comparative genomics reveals mobile pathogenicity chromosomes in Fusarium Nature, 464 (7287), 367-373 DOI: 10.1038/nature08850






0 comentarios:
Publicar un comentario
Se respetuoso con tus comentarios y críticas. Cualquier comentario ofensivo será eliminado.