Hace un par de días habíamos hablado del como pudo haber sido el origen del código genético y, para que tenga una continuidad, hoy hablaremos de como funciona exactamente los ribosomas. Recordando: Los ribosomas son unas estructuras especializadas formadas por algo más de 50 proteínas y ARN ribosomal y se encarga de leer el ARN mensajero (ARNm) y traducirlo a proteínas a través de los codones del ARNm y los anticodones del ARN de transferencia (ARNt) que cargan en el otro extremo un determinado aminoácido.
Los ribosomas tienen tres sitios bien determinados. El sitio A es donde el ARNt cargando su respectivo aminoácido (aminoacilado) se une al ribosoma, luego pasa al sitio P dejando el sitio A libre para la entrada de otro ARNt aminoacilado y se forme el enlace peptídico que une a los dos aminoácidos. El ARNt que estaba en el sitio P se queda sin su aminoácido y pasa al sitio E de donde es liberado para que vuelva a conseguir otro aminoácido. El ARNt que estaba en el sitio A pasa al P cargando un aminoácido más, repitiendo este ciclo hasta terminar de formar la cadena de aminoácidos (proteína) con un ARNt de finalización (no carga aminoácido alguno). Este paso de translocación es hecha por la enzima GTPasa EF-G. ¿Se ve sencillo?… Pasar de A a P y luego a E. Sin embargo, detrás de esto hay todo un trabajo coordinado de muchos componentes. Uemura et al. reportaron el uso de una técnica extremadamente sensible para detectar, a nivel molecular, como se da este proceso.
El método que emplearon se llama “guía de onda en modo cero” (zero-mode waveguide: ZMW) el cual consiste en una nanoestructura de una capa de aluminio, con pequeños agujeros circulares de 50 a 200nm de diámetro en cuya base hay una capa de cristal de cuarzo. Este aparato permite analizar volúmenes de zeptolitros (10-21L), reduciendo las señales de fondo y otras interferencias.
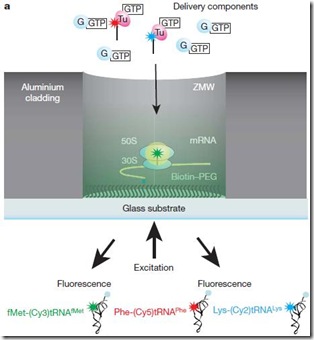
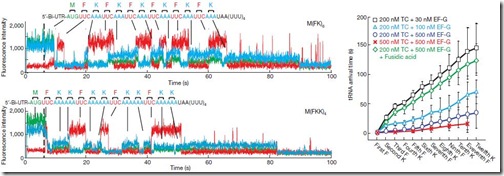
Primero diseñaron dos ARNm que codifican para una pequeña cadena de 4 aminoácidos (MFFF y MFKF). Probaron este experimento a bajas concentraciones de ARNt y GTPasa EF-G (30nM = 30 nanomoles por litro). Cuando no pusieron GTPasa EF-G en la reacción, como era de esperarse, sólo hubo un pulso rojo (de la F) —seguido al verde inicial de la metionina que siempre estará presente— y de ahí nada. Esto confirma que es necesaria la presencia de la GTPasa EF-G para la translocación de sitios. Luego pusieron 30nM de GTPasa EF-G y si observaron tres pulsos rojos (MFFF). A bajas concentraciones, la traducción tomaba ~20 segundos por codón. El ARNt era liberado del sitio E, la fluorescencia del ARNt del sitio P se extinguía a los 13.7seg, antes que llegue el nuevo ARNt al sitio A, así que no se pudo determinar exactamente cuanto tiempo se queda el ARNt en el ribosoma. Lo mismo se hizo con la secuencia MFKF. Entonces, el perfil de fluorescencia a bajas concentraciones es por pulsos. Uno verde, seguido por uno rojo, luego el verde desaparecía porque el ARNtMet dejaba su aminoácido y era liberado por el sitio E, luego el pulso rojo pasaba al sitio P y desaparecía a los 13 segundos. A los 20 aparecía otro pulso celeste correspondiente a K terminando con un pulso rojo correspondiente a F.
Bonito no?… Pero en las células no ocurre de esta manera. Las concentraciones de ARNt y GTPasa EF-G están en un orden micromolar (uM = 1000nM) y las cadenas de ARNm tienen muchos codones. Entonces, los investigadores diseñaron ARNm de 13 codones que codifican para 13 aminoácidos (M[FK]6 y M[FKK]4). Esta vez la concentración de ARNt se subió a 200nM y de GTPasa EF-G a 500nM. Lo primero que se observó fue que la velocidad de traducción por codón se incrementó considerablemente y había superposición de fluorescencias, esto quiere decir que había más de un ARNt en el ribosoma al mismo tiempo. A más concentración de GTPasa EF-G, menos tiempo entre la llegada de un ARNt a otro en el sitio A. En promedio eran ~4.1 segundos los que cada ARNt estaban unidos al ribosoma. A máxima concentración de ARNt y GTPasa EF-G, la velocidad fue de casi 1 codón por segundo, muy cercano a lo obtenido por organismos vivos in vitro. Sin embargo, el tiempo de llegada del primer ARNt siempre fue el mismo, sea cual sea la concentración de GTPasa EF-G. Otra cosa que se observó fue que M[FKK]4 se tradujo más rápidamente que M[FK]6, esto debido a que el carácter hidrofóbico de la Phe hace más lenta la traducción.
Pero, estos tiempos se dan cuando las dos sub-unidades del ribosoma están ensamblados. Que pasa cuando las sub unidades 30S y 50S están separados. Para esto, la sub-unidad 30S fue ensamblada a la ZMW (junto con el ARNtMet). Y se volvió a hacer el experimento. La traducción se desarrolló normalmente con un pequeño retraso inicial —en la llegada del primer ARNt— de 12 segundos. Este es el tiempo que le toma a la sub-unidad 50S en ensamblarse a la 30S.
Ahora, solo falta saber si en el ribosoma llegan a haber tres ARNt en un determinado momento (en cada uno de los sitios: A, P y E). Hasta ahora se había visto que hay superposición de dos señales, entonces será la llegada del ARNt al sitio A el responsable de la liberación del ARN sin aminoácido del sitio E? o la liberación del sitio E se hace de manera independiente a la llegada del ARNt al sitio A?. Se observó que a pesar que se aumentaba la concentración de GTPasa EF-G y ARNt y el tiempo de permanencia de los dos ARNt en los sitios A y P disminuían, sólo en ~1.7% de las veces se presentaban los 3 ARNt en los 3 sitios del ribosoma. Pero esto todavía no explicaba si el ARNt era liberado del sitio E por la llegada de un nuevo ARNt al sitio A, así que diseñaron un experimento muy inteligente.
Usaron el ácido fusídico, una molécula que hace lenta la traducción del ARNm, inhibiendo la llegada de un nuevo ARNt al sitio A. Si la hipótesis de que la liberación del ARNt del sitio E es debido a la llegada de un ARNt nuevo al sitio A fuera cierta, el ácido fusídico prolongará el tiempo de permanencia del ARNt en el sitio E, hasta que el ARNt nuevo pueda alcanzar el sitio A. Sin embargo, observaron que a pesar que no llegó el ARNt al sitio A, el ARNt sin aminoácido del sitio E ya se había liberado. La liberación del ARNt del sitio E se da inmediatamente después de la translocación y no está relacionada con la llegada de un nuevo ARNt al sitio A. Lo que ocurre es que a altas concentraciones, todo se da de manera rápida que justo coinciden la llegada y la salida de los ARNt.
Referencia:
Uemura, S., Aitken, C., Korlach, J., Flusberg, B., Turner, S., & Puglisi, J. (2010). Real-time tRNA transit on single translating ribosomes at codon resolution Nature, 464 (7291), 1012-1017 DOI: 10.1038/nature08925
Explicación mas sencilla:







0 comentarios:
Publicar un comentario
Se respetuoso con tus comentarios y críticas. Cualquier comentario ofensivo será eliminado.