No… no confundí el título y puse ‘PCR’ en vez de ‘cámara fotográfica’. Para serles sincero, hasta el día de hoy ignoraba por completo la existencia de la PCR Digital (dPCR), una variante de la PCR tradicional desarrollada por Vogelstein & Kinzler en 1999, que consiste en una PCR hecha en un ‘biochip’ (un microchip biológico), permitiendo hacer miles de reacciones de un sólo golpe, aumentando así la precisión y sensibilidad de la técnica. Ahora, científicos de la Universidad de la Columbia Británica (Canadá) han desarrollado un dispositivo de dPCR que permite hacer un millón de reacciones en una sola corrida según un artículo publicado ayer en Nature Methods.
La PCR (Reacción en Cadena de la Polimerasa) es, sin lugar a dudas, una de las más importantes herramientas de la biología molecular. Todo biólogo que se respeta por lo menos lo ha hecho una vez en su vida, mientras que otros —donde me incluyo— la hemos hecho o la hacemos todos los días, incluso domingos y feriados. La técnica consiste simplemente en ‘fotocopiar’ una secuencia específica de ADN, la cual puede ser un gen o una porción de él, con el fin de secuenciarlo, cuantificarlo, identificar mutaciones, ver si está presente o no en una determinada muestra, etc.
La técnica fue inventada por Kary Mullis en los 80’s, lo que le valió el Premio Nobel de Química en 1993. Inicialmente la técnica era bastante laboriosa, pero con el paso de los años se empezó a volver cada vez más sofisticada. La primera innovación fue el uso de la ADN polimerasa termoestable obtenida de un microorganismo termófilo (Termophilus aquiaticus) —de ahí su nombre Taq Polimerasa—, luego fue la aparición de los sistemas automatizados de control de temperatura (termocicladores), y finalmente fue el uso de las sondas fluorescentes que permitían cuantificar y seguir en tiempo real la amplificación de una determinada secuencia de ADN.
Para resumir…
Sin embargo, la PCR cuantitativa (qPCR) no es muy precisa para medir las concentraciones absolutas de una determinada secuencia, sobre todo si la cantidad inicial de copias es sumamente baja. Por otro lado se corre el riesgo de que hayan amplificaciones inespecíficas y que se de la competencia con otras secuencias por el sitio activo de la enzima. Esto es un problema cuando se quiere hacer un diagnóstico preventivo de ciertos tipos de cáncer o enfermedades virales ya que la cantidad inicial de células portando el gen mutado o del virus en sangre es tan baja que las técnicas actuales de qPCR no pueden detectarlas.
En 1999, gracias al desarrollo de la nanotecnología, los microfluidos y las microemulsiones, Vogelstein & Kinzler desarrollaron la PCR digital. Esta técnica consistía en el uso de una lámina con pequeños pocillos donde se lleva a cabo la PCR de una única copia de ADN. Para esto, el número de copias inicial debe ser bajo —una ventaja si se trata de muestras para diagnóstico preventivo— para garantizar que sólo haya una copia de ADN por pocillo. Luego, a través del uso de sondas fluorescentes, se detectaría en cuales hubo amplificación y en cuales no, o en cuales hay copias mutantes a través del uso de dos o más sondas fluorescentes (uno para el tipo normal y otros para los mutantes). Por otro lado, gracias al uso de pequeñas cantidades de muestra, la especificidad y sensibilidad de la técnica es mayor mientras que el riesgo de contaminación se reduce y los costos bajan.
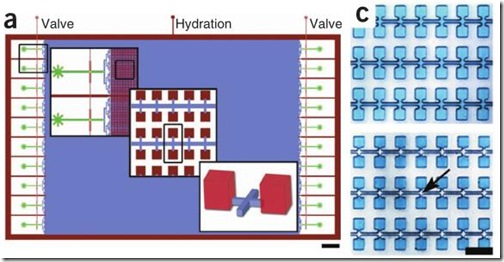
Actualmente, los dispositivos comerciales de dPCR cuentan con unos 36,960 pocillos de reacción (~6,000 pocillos por cm2). Sin embargo, Kevin Heyries y sus colaboradores de la Universidad de la Columbia Británica han desarrollado un dispositivo que puede realizar 1’000,000 de reacciones (~440,000 por cm2), con volúmenes de 10pL (10pL = 0.00000001mL) por pocillo.
El dispositivo esta hecho a base de silicona (polidimetilsiloxano: PDMS) recubierto de un aceite inmiscible fluorado, esto para que el ADN junto a los reactivos de la PCR puedan avanzar con normalidad, no quedarse pegados en las paredes y así poder rellenar el millón de compartimientos. Además, el dispositivo está protegido por una capa de Parileno C que evita que el vapor de agua generado durante la PCR se escape del dispositivo.
Heyries et al. probaron su dispositivo mediante una PCR del rpph1 y lo compararon con la qPCR. Las concentraciones de ADN molde que usaron fueron de 920fg/ul hasta 780ng/ul. El dispositivo fue capaz de detectar todo este rango de concentración de ADN sin llegar a saturarse, mientras que la qPCR —bajo las mismas condiciones— no lo pudo hacer a concentraciones menores a 0.7ng/ul y superiores a 700ng/ul. En otras palabras, el rango de detección del dispositivo fue de 7 órdenes de magnitud mientras que de la qPCR de 4.
Luego, los investigadores probaron el poder de detección de alelos mutantes de un gen. Para ello usaron dos genes con sus respectivas sondas fluorescentes, uno para la versión normal del gen jak2 y otro para la variante V617F. Los genes fueron mezclados en proporciones que van de 1:1 hasta 1:10,000, ya que ~1/10,000 (10-4) es la tasa de error de la Taq Polimerasa, representando la principal limitante de las técnicas basadas en la qPCR. Del millón de reacciones, el dispositivo sólo mostró 38 falsos positivos. Heyries et al. también probaron si el dispositivo era capaz de detectar los mutantes en proporciones 1:50,000 y 1:100,000. Los resultados fueron asombrosos —el dispositivo detectó 11 y 5 mutantes, respectivamente, en 500,000 reacciones. Esto indica que la PCR digital de 1Mp es 5,000 veces más sensible que la qPCR.
También probaron la capacidad de discriminar el número de copias de un determinado gen en una muestra de ADN. Primero hicieron una dPCR de dos genes humanos de única copia: el gen rpph1 del cromosoma 14 y el gen hlcs del cromosoma 21. Teóricamente, estos genes se encuentran en una proporción de 1:1 en todas las células. Los resultados de la dPCR mostraron una proporción promedio de 1.040:1, muy cercano al valor teórico. Luego, hicieron una variación al experimento mezclando la muestra inicial de ADN con un 1% al 6% de ADN de una persona con el síndrome de Down, la cual tiene tres cromosomas 21 en vez de dos, por lo tanto tendrá una copia más del gen hlcs. La dPCR fue capaz de discriminar un desbalance en la proporción en todos lo casos.
Sin dudas es un gran avance en la tecnología de la PCR ya que su sensibilidad se ha visto aumentada considerablemente (5,000 veces más que la qPCR). Por otro lado, los dispositivos pueden sub-dividirse en campos de 10,000 pocillos, de esta manera se pueden analizar 100 muestras diferentes por corrida, bajando los costos hasta el nivel de la qPCR, para poder ser implementado como metodología rutinaria en el diagnóstico de distintas enfermedades.
Referencia:
Kevin A Heyries, Carolina Tropini, Michael VanInsberghe, Callum Doolin, Oleh I Petriv, Anupam Singhal, Kaston Leung, Curtis B Hughesman, & Carl L Hansen (2011). Megapixel digital PCR Nature Methods DOI: 10.1038/nmeth.1640









aqui iba el comentario del post anterior. disculpar
ResponderBorrar